1月下半笔记(个人向)
最近才开始看d2l(这种东西早该在两年前看的,拖到现在了)
为了做项目还得学一手OpenGL(被windows安装GLFW逼疯了)
1.15
打完ICPC EC final回来,也许可以出一篇博客写下简单的题解。
对蛋白质相似空间子结构搜索的算法有了一些思路,简单分为传统算法和目标检测算法
1.16
想到要对蛋白质可视化,然后去找可以用于三维绘图的库,找到了OpenGL,在wsl里面装了一个,发现挺方便,准备开始学[傅老師/OpenGL教學 第一章] OpenGL自製3D遊戲引擎 (已更畢)_哔哩哔哩_bilibili
然后发现蛋白质三维结构是需要跑alphafold的(自己电脑根本跑不了),还不如直接下载网上的已经预测好结构的数据集。
1.17
1、UniProt
学习了UniProt网站的一些知识,还有待进一步探索
struct里面可以查看各种蛋白质的三维结构,蛋白质数据都是可以下载的
标了reviewed的数据可信度更高,做实验优先选择这类数据
2、蛋白质三维结构比对算法
主要分为分子间距离比较和分子内距离比较。
分子间距离比较就类似迭代的做法,通过刚体旋转使得蛋白质分子间的cRMS指标最小
分子内距离dRMS的判据一般用于蛋白质的局部空间结构比较,可以绕过刚体旋转拟合的过程
结构比对算法已经有很多优秀的算法了,比如CE、TM-align、DALI、VAST、K2、SHEBA等之类的,似乎不用自己再写一个了。
1.18
查看网络情况
ipconfig,ping+网址,ss,ip link。
查看conda环境列表:conda env list
删除conda环境:conda env remove --name xxx(必须在非激活状态下删除)
创建conda环境:conda create --name xxx python=3.x
激活conda环境:conda activate xxx
在某环境下安装某个包:直接pip install
d2l网站,之前找了好久都没找到,今天一找就找到了:安装 — 动手学深度学习 2.0.0 documentation
1.19
滑动窗口维护次大值
一个神奇的方法,单调队列,每次把z加入队列时比较队尾的两个元素x,y,
若z>=x&&z>=y,弹出x、y中的较小值,直到不满足该条件后,将z加入队尾
这样可以保证,区间最大值一定是前两个数之一,次大值一定是前四个数之一。
为什么呢
用反证法,最大值在第三位,那么,1、2位都会小于第三位,那么应该弹出1、2位之一,矛盾。
若次大值在第五位,那么1234位中一定有三位小于次大值,而最大值一定在1、2位,所以3、4位一定小于第五位的次大值,那么3、4位必定弹出一个,矛盾。
综上以上维护方式可以维护最大值与次大值。
yyq%%%%%我直接膜爆yyq,这么多年了,他还在c!他还在输出!yyq yyds!!!
1.20
休息日,和yyq打MC,发现金头盔可以避免自己被猪灵攻击。
1.21
看Struct2GO论文,看d2l
python os模块学习:第26天:Python 标准库之 os 模块详解 - 纯洁的微笑博客
和操作系统实验里面写的函数用法几乎一模一样,使得python可移植性很强。
将pandas的dataframe数据类型转换为tensor(先转numpy再转tensor)
A=torch.tensor(xx.to_numpy(dtype=xxx))(xx为dataframe类型的变量)
d2l笔记:
数据预处理,离散缺失值一般单独分一类或者用众数代替,连续缺失值可以用平均数或者众数代替。
torch.arange(n,dtype=xxxx)(生成0~n-1的数据类型为xxxx的一串数)
设A、B为tensor张量
A.reshape(n,m,……)(保证数据总数不变的情况下对数据进行变形)
A.sum(axis=0、1)(按照行、列求和)
A.cumsum(axis=0、1)(按照行列进行前缀和计算)
A+B,对应位置相加
A*B ,Hadamard积,就是对应位置相乘
torch.dot(A,B)向量点积
torch.mv(A,B)矩阵乘向量(A为矩阵n*m,B为向量m,乘出来为一个n维列向量)
(mv表示matrix*vector)
torch.mm(A,B)矩阵乘法,不解释
范数复习:
向量范数:

矩阵范数:F、1、2、∞
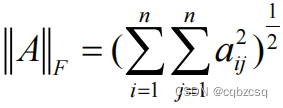

![]()
范数性质:

![]() (矩阵范数特有)
(矩阵范数特有)
torch.norm(A):计算向量A的2范数、矩阵A的F范数
torch.abs(A).sum():计算向量的1范数
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:chenni525@qq.com进行投诉反馈,一经查实,立即删除!