spatialRF
官网:Easy Spatial Modeling with Random Forest ? spatialRF (blasbenito.github.io)
spatialRF是一种在考虑空间自相关的前提下,利用随机森林对空间数据进行回归并解释的R包。
数据要求

参数命名
data:训练集,data frame。
dependent.variable.name:因变量(y)的列名。
predictor.variable.names:所有自变量(x)的列名。
xy:每个实例的坐标(一行一个坐标),按照 x、y 的顺序命名的 2 列,数据类型可以是 data frame 或 matrix。
distance.matrix:data中各个实例之间的距离,一个矩阵,矩阵的长宽皆为实例数量(行数)。
distance.thresholds:需要计算空间自相关的距离,一个向量。单位与distance.matrix相同。向量包含多个距离阈值,之后会根据每个距离阈值计算 Moran I。
数据探索
① 数据分布初探
通过 plot_training_df 函数来绘制每个x与y的散点图,初步判断每个x与y的大致关系,比如正相关、负相关、相关是否显著......
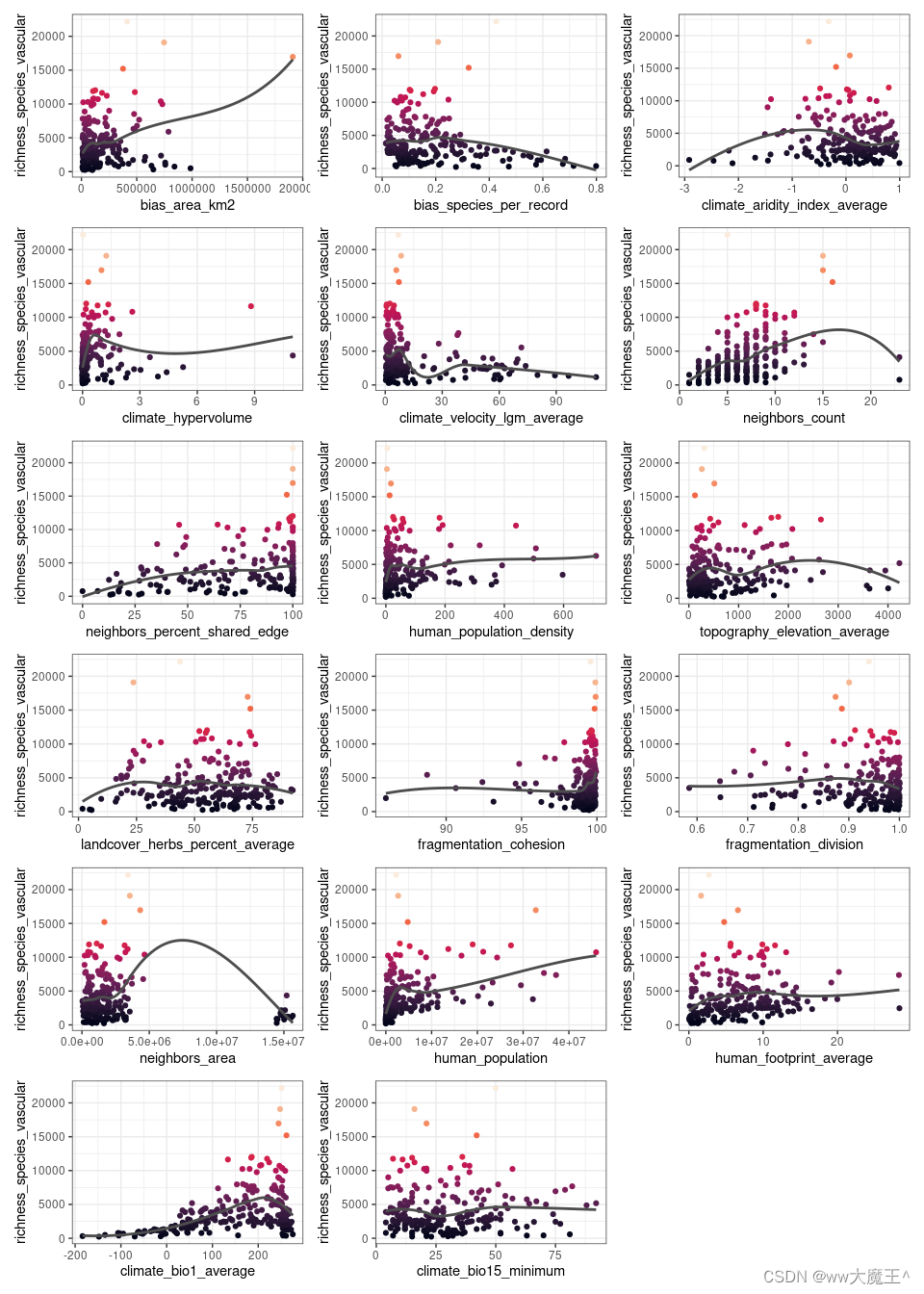 ②?空间自相关性(spatial autocorrelation)
②?空间自相关性(spatial autocorrelation)
通过 plot_training_df_moran 函数来绘制每个变量的空间自相关性,变量包括y和每个x。此函数用 Moran's I 来衡量空间自相关性,且该函数默认当p值大于等于0.05时视作该变量在给定距离阈值下的空间自相关性不显著。
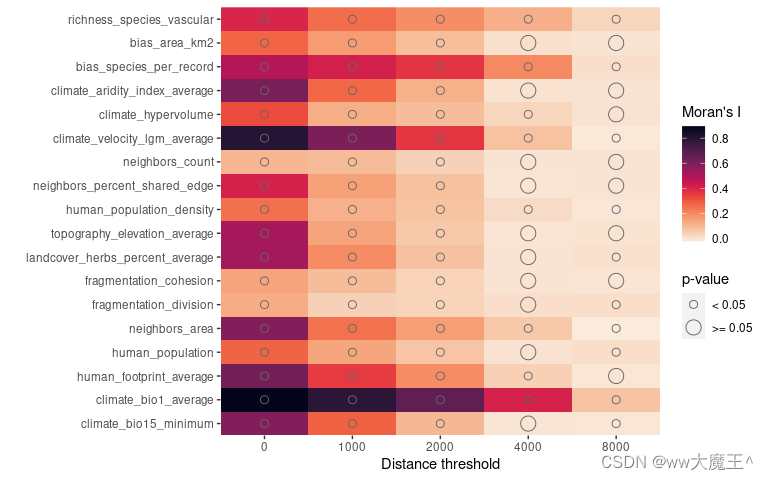
③?多重共线性(multicollinearity)
通过 auto_cor 和 auto_vif 两个函数来检查 x 的多重共线性。
auto_cor 使用双变量R方来度量多重共线性,auto_vif 使用方差膨胀因子来度量多重共线性。
用户可以通过参数preference.order来定义变量的偏好顺序(这个参数不是必须的,没有偏好可以不写。设置这个参数是为了引入专业知识来调控变量的选择。)
代码示例:
preference.order <- c( # 定义变量的偏好顺序,最重要的在最前面
"variable_1",
"variable_2",
"variable_3"
)
predictor.variable.names <- spatialRF::auto_cor(
x = dataframe[, predictor.variable.names], # dataframe为整个数据集,包含y和所有x。
cor.threshold = 0.75, # 默认值
preference.order = preference.order # 如果没有偏好,就删掉这一行
) %>%
spatialRF::auto_vif(
vif.threshold = 5, # 默认值
preference.order = preference.order
)
predictor.variable.names$selected.variables # 查看所有被选择的变量(去除多重共线性之后剩下的变量)输出示例:
## [auto_cor()]: Removed variables: variable_2
## [auto_vif()]: Variables are not collinear.
④ 特征工程
这其实是个比较宏大的问题,不过spatialRF给出一个懒人函数:the_feature_engineer,这个函数会对所有最重要的x之间的组合进行测试,输出最重要的一些组合。
个人觉得特征工程最好还是结合专业知识进行。
拟合与预测
① 原始的随机森林(non-spatial Random Forest?)
随机森林感觉没啥好说的,网上教程一搜一大把。。
代码示例:
# 模型拟合
model.non.spatial <- spatialRF::rf(
data = dataframe, # 你的数据集,必须包含y和所有模型需要使用的x。
dependent.variable.name = dependent.variable.name, # y的名称(列名)
predictor.variable.names = predictor.variable.names, # 所有模型需要使用的x的名称(如果进行了多重共线性处理、特征工程等操作,这里的x会和读取的不一样)
# distance.matrix、distance.thresholds、xy这三个参数,在non-spatial RF的拟合过程并不会用到。但是后续对结果的分析会用到(比如计算模型残差的自相关系数)
distance.matrix = distance.matrix,
distance.thresholds = distance.thresholds,
xy = xy,
seed = random.seed,
verbose = FALSE # 默认是True。当该参数是True时,会显示函数运行中的信息和图表。
)
# 模型应用:把上面拟合好的模型用到一个新的数据集上
predicted <- stats::predict(
object = model.non.spatial,
data = new_dataframe, # 新的数据集,必须包含之前拟合时用到的所有的x
type = "response"
)$predictions② 空间随机森林(spatial Random Forest)
该R包的核心。说是在随机森林中考虑了变量的空间自相关性,但我没找到模型具体是怎么考虑的说明。不过从后分析图表上可以看到,spatialRF 输出结果的残差的空间自相关性 确实比 non-spatialRF 小了不少。这个图表下一节“结果分析”会说。
代码示例:
model.spatial <- spatialRF::rf_spatial(
model = model.non.spatial,
method = "mem.moran.sequential",
verbose = FALSE,
seed = random.seed
)从代码可以看出,spatialRF是基于non-spatialRF建立的。
结果分析
① 模型拟合的残差(Residuals)
non_spatial_residuals <- model.non.spatial$residuals # non-spatialRF输出结果的残差(可以理解成预测值和真实值之间的误差)
spatialRF::plot_residuals_diagnostics( # 绘制一大波有关残差的图表
model.non.spatial,
verbose = FALSE
)
spatialRF::plot_moran( # 绘制莫兰指数(Moran's I),上面那个函数包含了这个图。
model.non.spatial,
verbose = FALSE
)一大波残差图大概长这样:最上面的图是正态性检验,中图显示残差和拟合值之间的关系,下图就是大家喜闻乐见的 Moran's I 图。
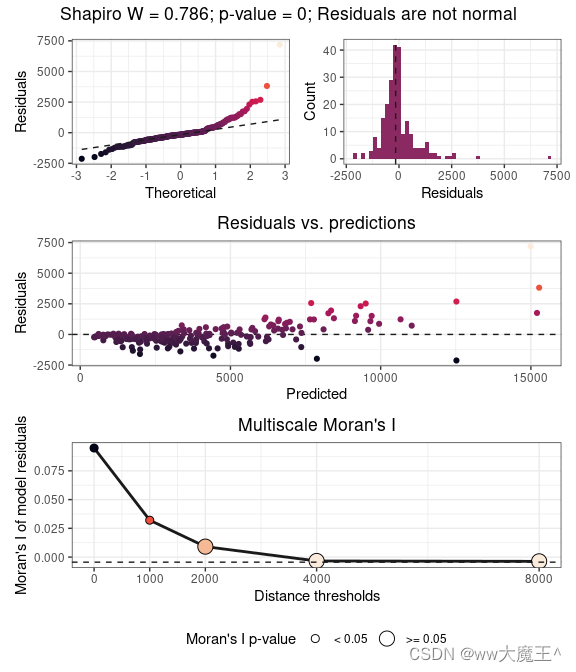
② 特征的重要性
(a) 全局重要性
non_spatial_variable_importances <- model.non.spatial$variable.importance
spatialRF::plot_importance( # 绘制特征重要性图
model.non.spatial,
verbose = FALSE,
scaled.importance = TRUE # 对特征重要性进行归一化处理。非必选项。
)
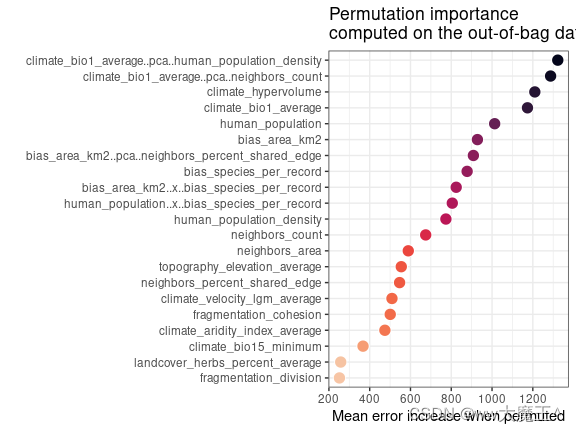
我的理解是,变量重要性(variable importance)表示当某个变量被剔除时,模型误差的增加。
(b) 局部重要性
分析x对每个实例(每一行)的重要性;即针对每个实例,都会有一套所有x的重要性。
local_importances <- spatialRF::get_importance_local(model.non.spatial)
# 显示local_importances 的一些行和一些列
kableExtra::kbl(
round(local.importance[1:10, 1:5], 0), # 1~10行,1~5列
format = "html"
) %>%
kableExtra::kable_paper("hover", full_width = F)
ps:其实我漏了一个调参没讲。因为我用这个包的应用场景不需要调参O(∩_∩)O哈哈~日后如果用到了会补充。(希望永远不要再碰R了,我爱python!!!)
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:chenni525@qq.com进行投诉反馈,一经查实,立即删除!
- Python教程
- 深入理解 MySQL 中的 HAVING 关键字和聚合函数
- Qt之QChar编码(1)
- MyBatis入门基础篇
- 用Python脚本实现FFmpeg批量转换
- 每个人的经济学-第五章“货币政策的难题”
- Fortune Telling
- SELinux介绍
- 【java/数据库课程设计】——基于springboot+vue+mysql的随机点名器
- 一个成功的camera案例:ros2+gazebo+摄像头
- 2023航天推进理论基础考试划重点(W老师)冲压&电推进
- java火车查询管理系统Myeclipse开发mysql数据库web结构java编程计算机网页项目
- android studio 按键点击事件的实现方法
- # .NET Framework中使用命名管道进行进程间通信
- TCP状态转换/ 半连接/ 端口复用代码实现