数据挖掘(作业3
任务一
对以下数据集使用K均值聚类算法:
1)观察实验结果是否符合预期;
2)利用SSE标准确定K值;
3)自行调参并观察对聚类结果的影响。
注意:需要把类别信息去掉。
“tutorial3_Data Exploration”中的鸢尾花数据集“iris.data”
“tutorial4_Data Preprocessing”中的癌症数据集“breast-cancer-wisconsin.data”
breast-cancer-wisconsin.data?
?
import pandas as pd
cancer = pd.read_csv('D:\\数据挖掘\\实验3 聚类 代码与数据\\breast-cancer-wisconsin.data',header=None,names=['id','Clump Thickness','Uniformity of Cell Size','Uniformity of Cell Shape','Marginal Adhesion','Single Epithelial Cell Size','Bare Nuclei','Bland Chromation','Normal Nucleoli','Mitoses','Class'])
cancer
import pandas as pd
import numpy as np
from sklearn import cluster
from sklearn.preprocessing import StandardScaler
from sklearn.impute import SimpleImputer
# 数据预处理
cancer.replace('?', np.nan, inplace=True) # 将 '?' 替换为 NaN
cancer.dropna(inplace=True) # 删除包含 NaN 值的行
cancer = cancer.astype(float) # 将数据类型转换为浮点型
# 使用SimpleImputer填补缺失值
imputer = SimpleImputer(strategy='median')
cancer_imputed = imputer.fit_transform(cancer)
# 数据缩放
scaler = StandardScaler()
data_scaled = scaler.fit_transform(cancer_imputed)
# 将缩放后的数据转换为 DataFrame
data_scaled_df = pd.DataFrame(data_scaled, columns=cancer.columns)
# 删除 'Class' 列
data = data_scaled_df.drop('Class', axis=1)
data = data_scaled_df.drop('id', axis=1)
# 使用K均值聚类算法
k_means = cluster.KMeans(n_clusters=2, max_iter=100, random_state=1)
k_means.fit(data)
labels = k_means.labels_
# 创建 DataFrame 来存储聚类结果
result_df = pd.DataFrame(labels, index=cancer.index, columns=['Cluster ID'])
print(result_df['Cluster ID'].value_counts()) # 打印各个簇的样本数量
pd.DataFrame(labels, index=cancer.id, columns=['Cluster ID'])?
# 训练不同K值下的KMeans模型,并记录SSE
sse = []
k_range = range(1, 11)
for k in k_range:
kmeans = KMeans(n_clusters=k, random_state=42)
kmeans.fit(data)
sse.append(kmeans.inertia_)
# 绘制SSE与K值的关系图
import matplotlib.pyplot as plt
plt.plot(k_range, sse, marker='o')
plt.xlabel('Number of clusters (K)')
plt.ylabel('Sum of Squared Errors (SSE)')
plt.title('Elbow Method for Optimal K')
plt.show()利用SEE标准确定K,发现拐点在k=2的时候,说明k的最优值是2,符合该数据集?
?
调整参数
max_iter最大迭代次数,通过改变最大迭代次数,发现对分类的影响不是很大

改变k值,可以得出和上面一样的结论,k为2的时候的效果最好

改变tol,发现收敛阈值对于模型的性能的影响不是很大
?
?
任务二
层次聚类层次聚类(学习笔记-CSDN博客
https://blog.csdn.net/qq_55552561/article/details/135165713?spm=1001.2014.3001.5501
任务三
查阅scikit-learn文档中的数据生成器(Samples generator,https://scikit-learn.org/stable/modules/classes.html#module-sklearn.datasets )请至少生成5种不同(形状或者分布)的数据集,并使用DBScan和谱聚类进行聚类分析,观察实验结果,结合算法原理进行分析。?
谱聚类和DBscan数据挖掘--聚类-CSDN博客
?生成数据:
# 生成数据集
datasets = [
? ? make_blobs(n_samples=1000, centers=3, cluster_std=1.0, random_state=42),
? ? make_moons(n_samples=1000, noise=0.1, random_state=42),
? ? make_circles(n_samples=1000, noise=0.05, factor=0.5, random_state=42),
? ? make_classification(n_samples=1000, n_features=20, n_classes=2, n_clusters_per_class=2),
? ? make_regression(n_samples=100, n_features=2)
]
from sklearn.datasets import make_blobs, make_moons, make_circles,make_classification,make_regression
from sklearn.cluster import DBSCAN, SpectralClustering
import matplotlib.pyplot as plt
# 生成数据集
datasets = [
make_blobs(n_samples=1000, centers=3, cluster_std=1.0, random_state=42),
make_moons(n_samples=1000, noise=0.1, random_state=42),
make_circles(n_samples=1000, noise=0.05, factor=0.5, random_state=42),
make_classification(n_samples=1000, n_features=20, n_classes=2, n_clusters_per_class=2),
make_regression(n_samples=100, n_features=2)
]
# DBSCAN 和谱聚类参数设置
dbscan = DBSCAN(eps=0.1, min_samples=5)
spectral = SpectralClustering(n_clusters=3, affinity='nearest_neighbors', random_state=42)
# 数据集索引
dataset_names = ['Blobs', 'Moons', 'Circles', 'S Curve', 'Swiss Roll']
# 分别对每个数据集进行可视化和聚类分析
for idx, (X, y) in enumerate(datasets):
# 可视化原始数据集
plt.figure(figsize=(18, 4))
# 绘制原始数据集
plt.subplot(1, 3, 1)
plt.scatter(X[:, 0], X[:, 1], c=y, cmap='viridis')
plt.title(f"Original {dataset_names[idx]} Dataset")
plt.xlabel("Feature 1")
plt.ylabel("Feature 2")
# DBSCAN 聚类
dbscan.fit(X)
db_labels = dbscan.labels_
# 谱聚类
spectral.fit(X)
spectral_labels = spectral.labels_
# 绘制DBSCAN聚类结果
plt.subplot(1, 3, 2)
plt.scatter(X[:, 0], X[:, 1], c=db_labels, cmap='viridis')
plt.title(f"DBSCAN Clustering on {dataset_names[idx]}")
plt.xlabel("Feature 1")
plt.ylabel("Feature 2")
# 绘制谱聚类结果
plt.subplot(1, 3, 3)
plt.scatter(X[:, 0], X[:, 1], c=spectral_labels, cmap='viridis')
plt.title(f"Spectral Clustering on {dataset_names[idx]}")
plt.xlabel("Feature 1")
plt.ylabel("Feature 2")
plt.tight_layout()
plt.show()
?运行得到的结果
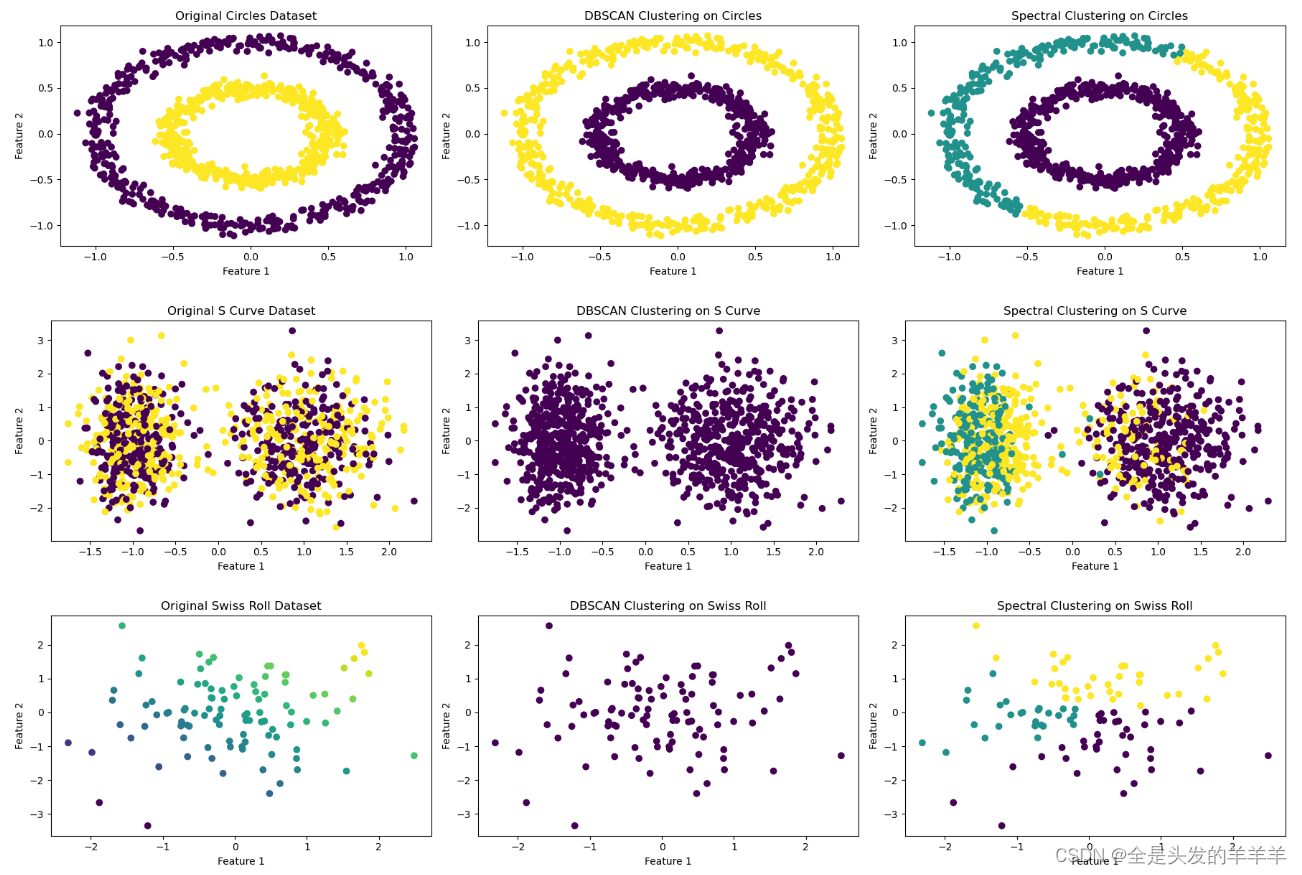 可以看出聚类存在很大的问题,特别是谱聚类,因为按理说谱聚类是对kmeans的一个方面的改进,应该可以比较好地对圆形数据和月牙形地数据做一个比较好的分类,但上述结果明显没有做到,于是下面将五个数据集分开,对不同的数据集,用不一样的明显参数进行分类。
可以看出聚类存在很大的问题,特别是谱聚类,因为按理说谱聚类是对kmeans的一个方面的改进,应该可以比较好地对圆形数据和月牙形地数据做一个比较好的分类,但上述结果明显没有做到,于是下面将五个数据集分开,对不同的数据集,用不一样的明显参数进行分类。
此外,在对数据生成的函数的进一步的了解,发现make_classification和make_swiss_roll生成的数据,不适用于对聚类算法的研究,于是下面只研究Blobs、Moons和Circle的数据。
Blobs数据:
修改了DBscan参数
# DBSCAN 和谱聚类参数设置
dbscan = DBSCAN(eps=0.5, min_samples=5)
spectral = SpectralClustering(n_clusters=3, affinity='nearest_neighbors', random_state=42)下面是三种不同的Blobs数据?

?这个数据集上,DBscan和谱聚类的分类效果都挺好的

?在上述这个数据集上的聚类,可以明显看出DBSCAN没有正确地进行分类,进行过多次调参,都没有办法达到更好的分类效果
????????在给定的合成数据集中,既有 DBSCAN 及谱聚类未能很好地对数据进行有效聚类。DBSCAN 未能有效识别形状复杂且交错的数据结构,这是因为 DBSCAN 对于类别之间的密度差异和噪声数据敏感,对于密度相近的区域难以分辨。而对于谱聚类,尽管它能够在某些情况下处理非凸形状的聚类结构,但它仍然基于样本之间的相似度图进行聚类,难以很好地解决数据交错在一起的情况。
????????对于交错在一起的数据,即使是 DBSCAN 和谱聚类这样的聚类算法也可能遇到困难。DBSCAN 依赖于密度可达性的概念,因此无法很好地处理密度相似的区域,谱聚类虽然不受凸形状的限制,但也是基于样本之间的相似性度量,因此在处理交错在一起的数据时也存在挑战。
????????对于类似这种交错在一起的数据结构,可能需要更复杂或更适合此类情况的聚类方法,或者在应用传统聚类算法之前,对数据进行特征工程或预处理,以更好地凸显数据内部的聚类结构,比如使用核方法、降维技术等。另外,使用可视化工具对数据进行分析,能够帮助更好地理解数据的特性及算法的表现,从而为选择适当的聚类算法提供指导。
Moons数据:?
修改了谱聚类的参数
# DBSCAN 和谱聚类参数设置
dbscan = DBSCAN(eps=0.1, min_samples=5)
spectral = SpectralClustering(n_clusters=2, affinity='rbf', random_state=42,gamma=500)?
Circles数据:
使用和上面这个月牙形一样的参数
# DBSCAN 和谱聚类参数设置
dbscan = DBSCAN(eps=0.1, min_samples=5)
spectral = SpectralClustering(n_clusters=2, affinity='rbf', random_state=42,gamma=500)?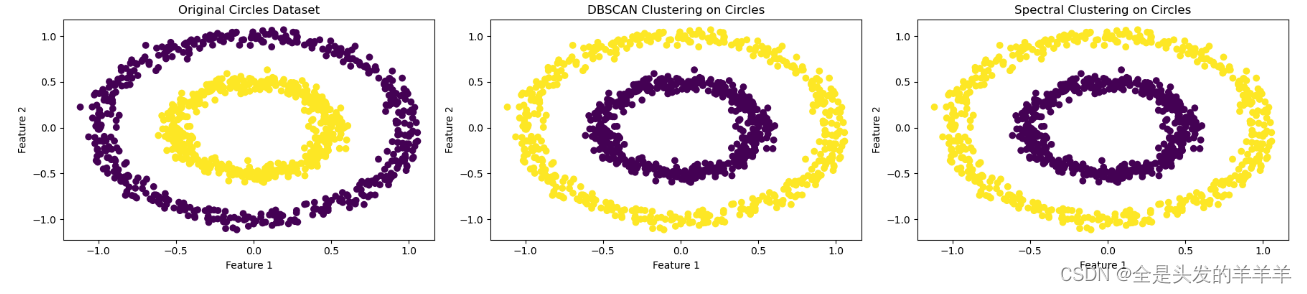
?
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:chenni525@qq.com进行投诉反馈,一经查实,立即删除!