基于YOLOv8的农作物水稻病害检测系统,优化SPPF提升检测精度
💡💡💡本文摘要:农作物水稻病害首先进行数据处理到训练模型,最好优化SPPF提升检测精度,map0.5从原始的0.807提升至0.821
1.YOLOv8介绍
?????????Ultralytics YOLOv8是Ultralytics公司开发的YOLO目标检测和图像分割模型的最新版本。YOLOv8是一种尖端的、最先进的(SOTA)模型,它建立在先前YOLO成功基础上,并引入了新功能和改进,以进一步提升性能和灵活性。它可以在大型数据集上进行训练,并且能够在各种硬件平台上运行,从CPU到GPU。
具体改进如下:
-
Backbone:使用的依旧是CSP的思想,不过YOLOv5中的C3模块被替换成了C2f模块,实现了进一步的轻量化,同时YOLOv8依旧使用了YOLOv5等架构中使用的SPPF模块;
-
PAN-FPN:毫无疑问YOLOv8依旧使用了PAN的思想,不过通过对比YOLOv5与YOLOv8的结构图可以看到,YOLOv8将YOLOv5中PAN-FPN上采样阶段中的卷积结构删除了,同时也将C3模块替换为了C2f模块;
-
Decoupled-Head:是不是嗅到了不一样的味道?是的,YOLOv8走向了Decoupled-Head;
-
Anchor-Free:YOLOv8抛弃了以往的Anchor-Base,使用了Anchor-Free的思想;
-
损失函数:YOLOv8使用VFL Loss作为分类损失,使用DFL Loss+CIOU Loss作为分类损失;
-
样本匹配:YOLOv8抛弃了以往的IOU匹配或者单边比例的分配方式,而是使用了Task-Aligned Assigner匹配方式

框架图提供见链接:Brief summary of YOLOv8 model structure · Issue #189 · ultralytics/ultralytics · GitHub
2.农作物水稻病害数据集介绍
数据集大小一共1448张,类别如下
# class names
names:
0: BrownSpot
1: RiceBlast
2: BacterialBlight2.1数据集划分
通过split_train_val.py得到trainval.txt、val.txt、test.txt??
# coding:utf-8
import os
import random
import argparse
parser = argparse.ArgumentParser()
#xml文件的地址,根据自己的数据进行修改 xml一般存放在Annotations下
parser.add_argument('--xml_path', default='Annotations', type=str, help='input xml label path')
#数据集的划分,地址选择自己数据下的ImageSets/Main
parser.add_argument('--txt_path', default='ImageSets/Main', type=str, help='output txt label path')
opt = parser.parse_args()
trainval_percent = 0.9
train_percent = 0.7
xmlfilepath = opt.xml_path
txtsavepath = opt.txt_path
total_xml = os.listdir(xmlfilepath)
if not os.path.exists(txtsavepath):
os.makedirs(txtsavepath)
num = len(total_xml)
list_index = range(num)
tv = int(num * trainval_percent)
tr = int(tv * train_percent)
trainval = random.sample(list_index, tv)
train = random.sample(trainval, tr)
file_trainval = open(txtsavepath + '/trainval.txt', 'w')
file_test = open(txtsavepath + '/test.txt', 'w')
file_train = open(txtsavepath + '/train.txt', 'w')
file_val = open(txtsavepath + '/val.txt', 'w')
for i in list_index:
name = total_xml[i][:-4] + '\n'
if i in trainval:
file_trainval.write(name)
if i in train:
file_train.write(name)
else:
file_val.write(name)
else:
file_test.write(name)
file_trainval.close()
file_train.close()
file_val.close()
file_test.close()?2.2 通过voc_label.py得到适合yolov8训练需要的
# -*- coding: utf-8 -*-
import xml.etree.ElementTree as ET
import os
from os import getcwd
sets = ['train', 'val', 'test']
classes = ["0 BrownSpot","1 RiceBlast","2 BacterialBlight"] # 改成自己的类别
abs_path = os.getcwd()
print(abs_path)
def convert(size, box):
dw = 1. / (size[0])
dh = 1. / (size[1])
x = (box[0] + box[1]) / 2.0 - 1
y = (box[2] + box[3]) / 2.0 - 1
w = box[1] - box[0]
h = box[3] - box[2]
x = x * dw
w = w * dw
y = y * dh
h = h * dh
return x, y, w, h
def convert_annotation(image_id):
in_file = open('Annotations/%s.xml' % (image_id), encoding='UTF-8')
out_file = open('labels/%s.txt' % (image_id), 'w')
tree = ET.parse(in_file)
root = tree.getroot()
size = root.find('size')
w = int(size.find('width').text)
h = int(size.find('height').text)
for obj in root.iter('object'):
difficult = obj.find('difficult').text
#difficult = obj.find('Difficult').text
cls = obj.find('name').text
if cls not in classes or int(difficult) == 1:
continue
cls_id = classes.index(cls)
xmlbox = obj.find('bndbox')
b = (float(xmlbox.find('xmin').text), float(xmlbox.find('xmax').text), float(xmlbox.find('ymin').text),
float(xmlbox.find('ymax').text))
b1, b2, b3, b4 = b
# 标注越界修正
if b2 > w:
b2 = w
if b4 > h:
b4 = h
b = (b1, b2, b3, b4)
bb = convert((w, h), b)
out_file.write(str(cls_id) + " " + " ".join([str(a) for a in bb]) + '\n')
wd = getcwd()
for image_set in sets:
if not os.path.exists('labels/'):
os.makedirs('labels/')
image_ids = open('ImageSets/Main/%s.txt' % (image_set)).read().strip().split()
list_file = open('%s.txt' % (image_set), 'w')
for image_id in image_ids:
list_file.write(abs_path + '/images/%s.jpg\n' % (image_id))
convert_annotation(image_id)
list_file.close()3.训练结果分析
confusion_matrix.png :列代表预测的类别,行代表实际的类别。其对角线上的值表示预测正确的数量比例,非对角线元素则是预测错误的部分。混淆矩阵的对角线值越高越好,这表明许多预测是正确的。

?上图是水稻病害检测训练,有图可以看出 ,分别是BrownSpot、RiceBlast、BacterialBlight和background FP。该图在每列上进行归一化处理。
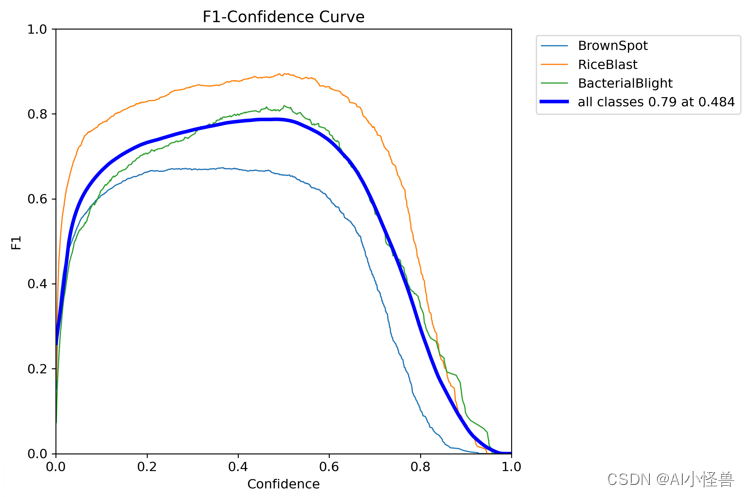
F1_curve.png:F1分数与置信度(x轴)之间的关系。F1分数是分类的一个衡量标准,是精确率和召回率的调和平均函数,介于0,1之间。越大越好。
TP:真实为真,预测为真;
FN:真实为真,预测为假;
FP:真实为假,预测为真;
TN:真实为假,预测为假;
精确率(precision)=TP/(TP+FP)
召回率(Recall)=TP/(TP+FN)
F1=2*(精确率*召回率)/(精确率+召回率)
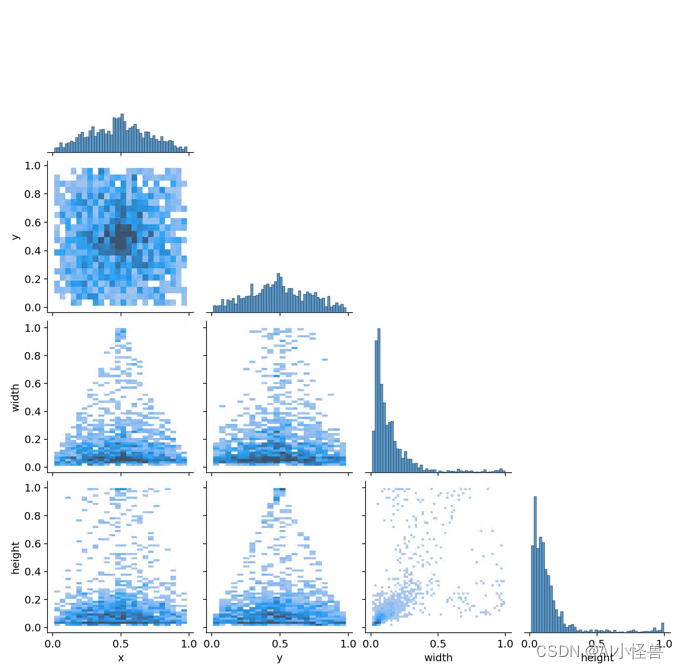
?labels_correlogram.jpg :显示数据的每个轴与其他轴之间的对比。图像中的标签位于 xywh 空间。
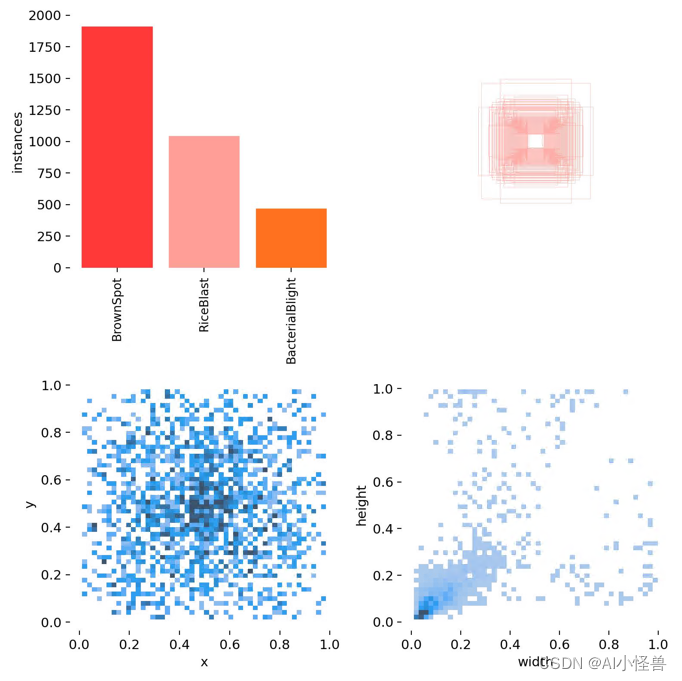
?labels.jpg :
(1,1)表示每个类别的数据量
(1,2)真实标注的?bounding_box
(2,1) 真实标注的中心点坐标
(2,2)真实标注的矩阵宽高
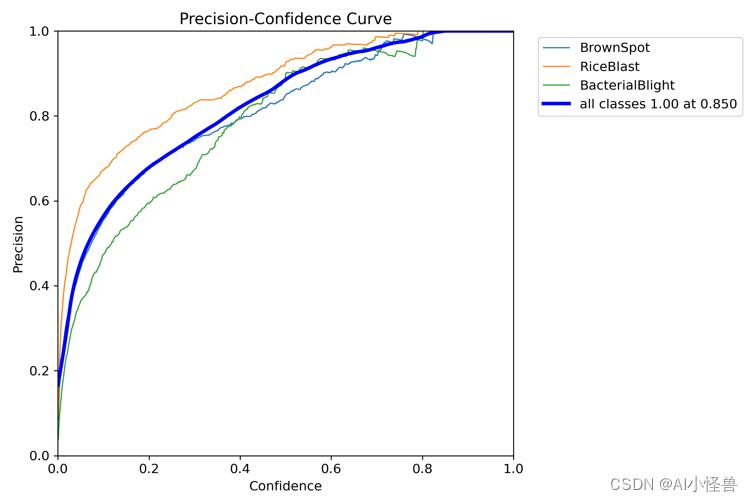
?P_curve.png:表示准确率与置信度的关系图线,横坐标置信度。由下图可以看出置信度越高,准确率越高。
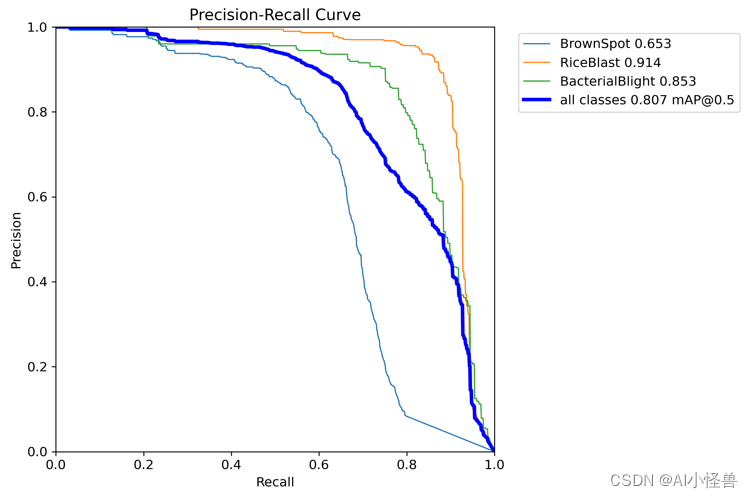
?PR_curve.png :PR曲线中的P代表的是precision(精准率),R代表的是recall(召回率),其代表的是精准率与召回率的关系。
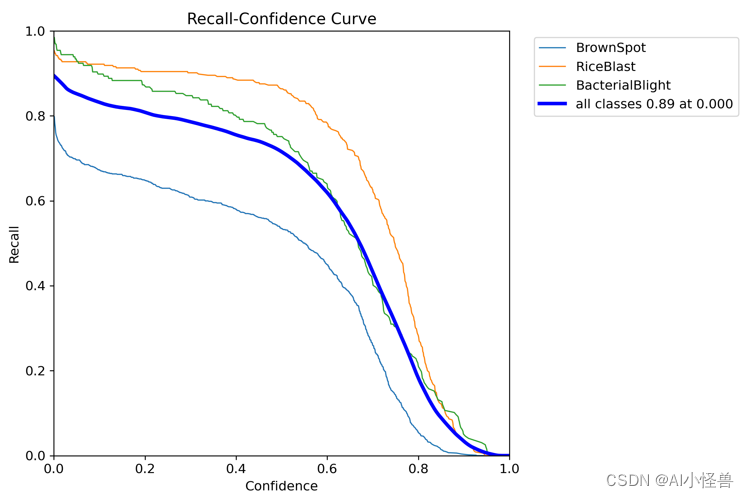
?R_curve.png :召回率与置信度之间关系

?预测结果:

YOLOv8n summary (fused): 168 layers, 3006233 parameters, 0 gradients, 8.1 GFLOPs
Class Images Instances Box(P R mAP50 mAP50-95): 100%|██████████| 7/7 [00:11<00:00, 1.58s/it]
all 391 1549 0.861 0.733 0.807 0.448
BrownSpot 391 1007 0.83 0.557 0.653 0.288
RiceBlast 391 345 0.899 0.873 0.914 0.564
BacterialBlight 391 197 0.854 0.77 0.853 0.4924.创新优化
加入SPPF自研创新 | SPPF与感知大内核卷积UniRepLK结合,大kernel+非膨胀卷积提升感受野
YOLOv8独家原创改进:SPPF自研创新 | SPPF与感知大内核卷积UniRepLK结合,大kernel+非膨胀卷积提升感受野-CSDN博客
4.1性能分析
map0.5从原始的0.807提升至0.821
YOLOv8n_SPPF_UniRepLK summary (fused): 186 layers, 5256601 parameters, 0 gradients, 9.9 GFLOPs
Class Images Instances Box(P R mAP50 mAP50-95): 100%|██████████| 7/7 [00:10<00:00, 1.57s/it]
all 391 1549 0.877 0.744 0.821 0.463
BrownSpot 391 1007 0.846 0.558 0.652 0.297
RiceBlast 391 345 0.918 0.873 0.935 0.581
BacterialBlight 391 197 0.868 0.802 0.875 0.51本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:chenni525@qq.com进行投诉反馈,一经查实,立即删除!
- Python教程
- 深入理解 MySQL 中的 HAVING 关键字和聚合函数
- Qt之QChar编码(1)
- MyBatis入门基础篇
- 用Python脚本实现FFmpeg批量转换
- RIS 辅助无线网络:基于模型、启发式和机器学习优化方法
- Java web汽车销售管理系统JavaServelet Jsp(Java毕业设计)
- centrifuge5.0.1版本请求websocket实例
- 2024 前端高频面试题之 JS 篇
- Ubuntu Desktop 死机处理
- 给零基础朋友的编程课07 - 代码
- Python学习之路——模块
- 什么?我被 Docker 一条龙服务了!
- MySQL用户管理
- 新算法!直接写: EVO-CNN-LSTM-Attention能量谷优化卷积、长短期记忆网络融合注意力机制的多变量回归预测程序