U-Mamba: Enhancing Long-range Dependency for Biomedical Image Segmentation
Abstract
卷积神经网络(Convolutional Neural Networks, cnn)和transformer是生物医学图像分割中最流行的架构,但由于固有的局部性或计算复杂性,它们处理远程依赖关系的能力有限。为了解决这一挑战,我们引入了U-Mamba,一个通用的生物医学图像分割网络。受状态空间序列模型(ssm)的启发,我们设计了一个混合CNN-SSM块,它将卷积层的局部特征提取能力与ssm捕获远程依赖的能力相结合。状态空间序列模型是一种以处理长序列的强大能力而闻名的新型深度序列模型。此外,U-Mamba具有自配置机制,允许它自动适应各种数据集,而无需人工干预。我们对四种不同的任务进行了广泛的实验,包括CT和MR图像中的三维腹部器官分割,内窥镜图像中的仪器分割和显微镜图像中的细胞分割。结果表明,U-Mamba在所有任务中都优于最先进的基于cnn和基于transformer的分割网络。这为生物医学图像分析中有效的远程依赖建模开辟了新的途径。代码、模型和数据可以在https://wanglab.ai/u-mamba.html上公开获得。
关键词:图像分割·骨干网·状态空间模型·变压器
1 Introduction
在本文中,我们提出了U-Mamba,一个通用的网络,用于三维和二维生物医学图像分割。基于创新的混合CNNSSM架构,U-Mamba可以捕获图像中的局部细粒度特征和远程依赖关系。该网络通过提供特征尺寸的线性缩放,而不是通常与变压器相关的二次复杂度,从流行的基于变压器的体系结构中脱颖而出。此外,U-Mamba的自配置功能可以无缝适应各种数据集,增强其在一系列生物医学细分任务中的可扩展性和灵活性。在四个不同的数据集上的定量和定性结果表明,U-Mamba取得了卓越的性能,大大超过了基于transformer的网络。这为未来网络设计的进步铺平了道路,有效地模拟生物医学成像中的远程依赖关系。
2 Method
U-Mamba遵循编码器-解码器网络结构,以有效的方式捕获本地特征和远程上下文。图1显示了U-Mamba区块的概况和完整的网络结构。
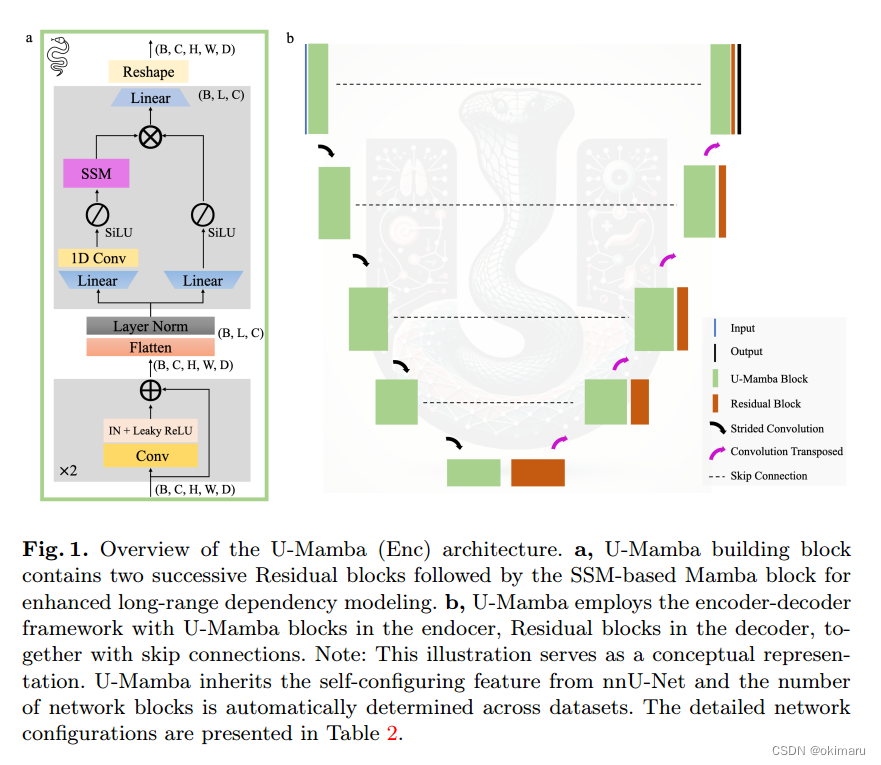
a, U-Mamba构建块包含两个连续的残余块,后面是基于ssm的Mamba块,用于增强的远程依赖关系建模。
b, U-Mamba采用编码器-解码器框架,其中U-Mamba块在编码器中,剩余块在解码器中,以及跳过连接。
注:此图作为概念性表示。U-Mamba继承了nnU-Net的自配置特性,并且在数据集之间自动确定网络块的数量。详细的网络配置如表2所示。
2.1 曼巴:选择性结构化状态空间序列模型扫描(S6)
状态空间序列模型(State Space Sequence Models, SSMs)[11]是一类映射一维函数或序列u(t)→y(t)∈R的系统,可以表示为线性常微分方程(ODE):

其中状态矩阵A∈RN×N, B, C∈RN为其参数,x(t)∈RN为隐潜状态。ssm提供了一些理想的特性,例如每个时间步的线性计算复杂度和用于高效训练的并行计算。然而,na?ve ssm通常比等价的cnn需要更多的内存,并且在训练过程中经常遇到梯度消失,阻碍了它们在一般序列建模中的更广泛应用。
结构化状态空间序列模型(S4)[14]通过在状态矩阵A上施加结构化形式并引入有效算法,显著改进了na?ve ssm。具体来说,使用高阶多项式投影算子(High-Order Polynomial Projection Operator, HIPPO)对状态矩阵进行制作和初始化[13],可以构建具有丰富能力和高效远程推理能力的深度序列模型。S4作为一种新型的网络架构,在具有挑战性的Long Range Arena Benchmark[44]上已经明显超越了Transformers[46]。
最近,Mamba[12]通过两个关键改进进一步推进了ssm在离散数据建模(例如文本和基因组)方面的应用。首先,Mamba采用了一种依赖于输入的选择机制,这与传统的时间和输入不变的ssm不同,它允许从输入中有效地过滤信息。
这是通过基于输入数据参数化SSM参数来实现的。其次,开发了一种硬件感知算法,在序列长度上线性缩放,通过扫描循环计算模型,使Mamba比现代硬件上的先前方法更快。此外,曼巴架构将SSM块与线性层合并,明显更简单,并在包括语言和基因组学在内的各种长序列领域展示了最先进的性能,在训练和推理过程中都展示了显著的计算效率。
2.2 U-Mamba: Marry Mamba with U-Net
图1b显示了完整的U-Mamba网络架构,其中编码器是用上述模块构建的,以捕获本地特征和远程依赖关系。该解码器由残差块和转置卷积组成,专注于详细的局部信息和分辨率恢复。此外,我们继承了U-Net中的跳过连接,将编码器和解码器的分层特征连接起来。最终的解码器特征与Softmax层一起传递到1 × 1 × 1卷积层,生成最终的分割概率图。此外,我们还实现了一个U-Mamba变体,其中只有瓶颈使用U-Mamba块,其他都是普通的残余块。我们使用“U-Mamba_Bot”和“U-Mamba_Enc”来区分这两种网络变体,它们分别在瓶颈和所有编码器块中使用U-Mamba块。
3 Experiments and results
3.1 Datasets

3.2 Implementation and training protocols
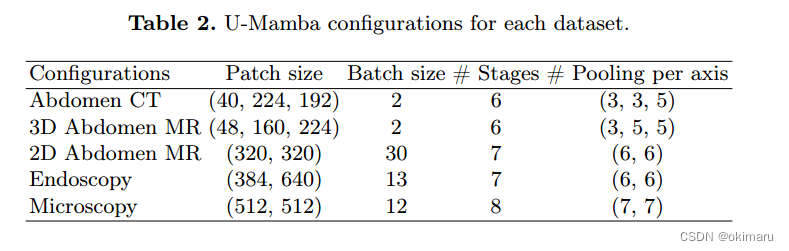
我们在流行的nnU-Net[22]框架中实现了U-Mamba,该框架由两个期望的特性驱动。首先,nnU-Net的模块化设计完全符合我们对引入新网络架构的关注。这种设计使我们能够专注于网络的实现,同时控制其他变量,如图像预处理和数据增强。这样的设置使U-Mamba在统一条件下与各种方法进行公平比较,网络架构是唯一不同的因素。其次,nnU-Net引人注目的自配置框架,能够为不同的分割数据集自动配置超参数,这是我们选择的另一个令人信服的原因。我们也保留这一功能,允许u -曼巴很容易适应广泛的分割任务。在训练过程中,补丁大小、批大小和网络配置(例如,分辨率状态的数量和沿不同轴的下采样操作的数量)与nnU-Net保持一致(表2)。U-Mamba也通过随机梯度下降进行了优化,损失函数是Dice损失和交叉熵的未加权和,因为复合损失已被证明在不同任务中具有鲁棒性[31]。在推理过程中,所有实验都禁用了测试时间增强(TTA),以简化和高效的评估过程,因为TTA对2D和3D数据集的计算负担分别增加了4倍和8倍。
3.3 Benchmarking
我们将U-Mamba与两种基于cnn的分割网络(nnUNet[22]和SegResNet[39])和两种基于transformer的网络(UNETR[17]和SwinUNETR[16])进行了比较,这两种网络在医学图像分割竞赛中被广泛使用。为了公平比较,我们还将SegResNet、UNETR和SwinUNETR实现到nnU-Net框架中,并使用他们建议的优化器(例如Adam[26]和AdamW[29])进行模型训练。我们使用nnU-Net中的默认图像预处理[22]。所有网络在同一批处理大小的NVIDIA A100 GPU上从头开始训练1000次(表2)。
根据Metrics Reloaded[37]中的建议,我们使用Dice Similarity Coefficient (DSC)和Normalized Surface Distance (NSD)进行三个语义分割任务,包括CT和MRI扫描中的器官分割,以及内镜图像中的仪器分割。由于单元格分割是一个实例分割任务,因此采用F1分数来评价单元格分割的质量。
3.4 Quantitative and qualitative segmentation results
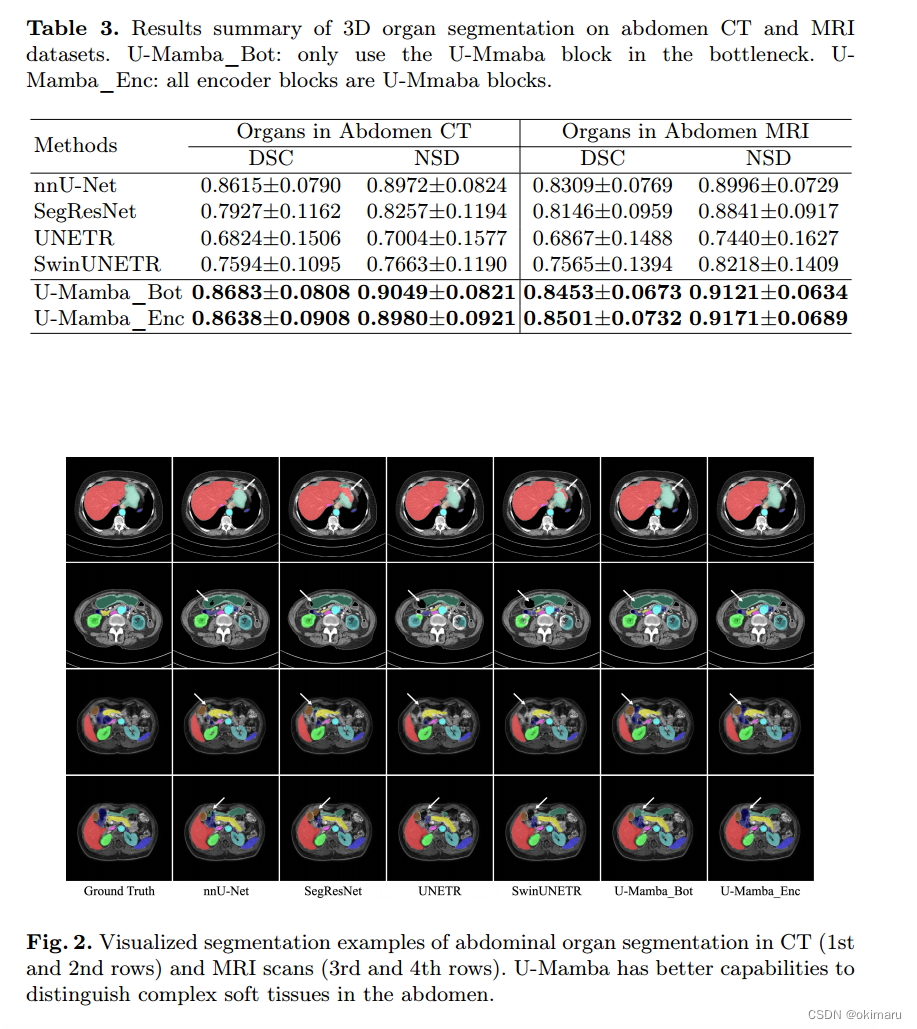
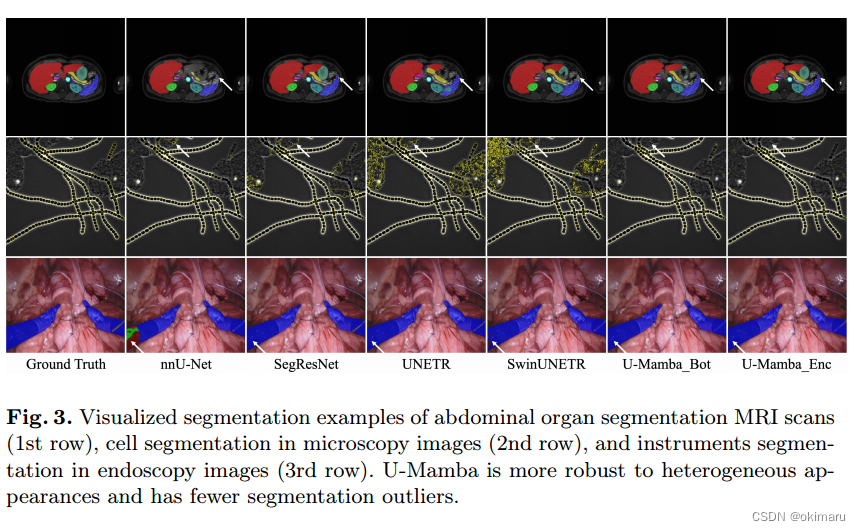
表3为CT和MRI扫描腹部脏器的定量三维分割结果。U-Mamba优于CNN和基于transformer的分割网络,在腹部CT和MR数据集上的平均DSC得分分别为0.8683 (U-Mamba_Bot)和0.8501 (U-Mamba_Enc)。nnU-Net具有竞争力,比SegResNet、UNETR和SwinUNETR要好得多,但U-Mamba仍然具有优势,如图2所示。从六种网络的可视化分割结果来看,UMamba在分割结果中的异常值较少。例如,U-Mama可以生成更准确的肝脏和胃分割面具,而其他的在腹部CT扫描中肝脏面具存在过度分割错误,胃面具缺失区域。同样,对于MRI扫描中的胆囊分割,U-Mamba成功地描绘了其边界,而其他方法会产生各种分割错误。这些观察结果也反映在器官方面的DSC和NSD评分中(附录表5和表6)。
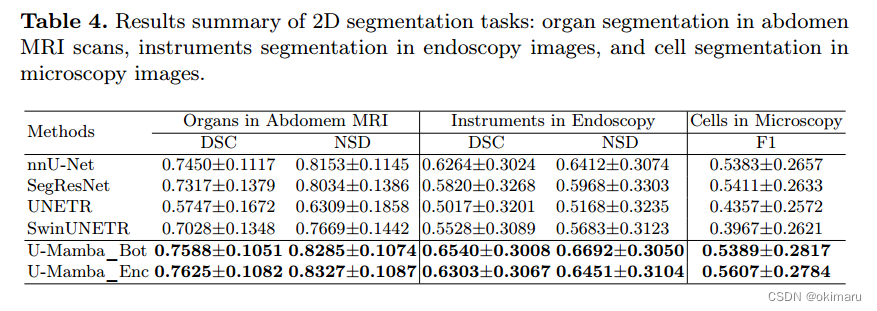
表4为定量二维分割结果。MRI扫描通常比CT扫描具有更多的非均匀间隔,二维分割网络也是实践中常见的选择[4]。因此,我们也比较了腹部器官分割的2D设置下的所有网络,其中每个3D MRI扫描被转换成多个2D切片。U-Mamba在三个2D分割任务上再次证明了其优于现有方法的优势,在器官分割、仪器分割和细胞分割上,U-Mamba分别获得了0.7625、0.6504和0.5607的最佳平均DSC和F1分数。图2的定性结果显示,nnU-Net、UNETR和SwinUNETR因其异质外观而混淆了脾脏。UNETR和SwinUNETR在显微镜图像中也产生了许多细胞异常值,nnU-Net未能成功识别内窥镜图像中的大针头驱动器。相比之下,U-Mamba在这些场景中表现出明显的优势,表明它有更好的捕捉全球环境的能力。
4 Discussion and conclusion
在本文中,我们引入U-Mamba来解决由于cnn的固有局部性和变压器的计算复杂性而导致的远程依赖关系建模的挑战。综合实验结果表明,在不同的模式和分割目标上,U-Mamaba优于现有的基于CNN和transformer的分割网络。特别是,U-Mamba在处理具有异质外观的对象方面表现出了卓越的能力,从而减少了分割异常值。性能的提高很大程度上归功于U-Mamba的架构设计,它可以同时提取多尺度的局部特征和捕获远程依赖关系。
变形金刚在许多自然图像分割任务中表现出更强的性能[28]。然而,它们并没有改变医学图像分割。这一点很明显,因为在医学图像分割竞赛中获胜的解决方案仍然主要依赖于CNNs6,如nnU-Net[22]和SegResNet[39]。有趣的是,我们还观察到,在我们所有的测试场景中,基于transformer的网络相对于基于cnn的网络表现不佳,尽管使用了它们的原始网络实现并使用推荐的优化器对它们进行了训练。这可能是用多个GPU而不是一个GPU来训练这些网络的要求。然而,这将导致不公平的比较,因为所有其他网络只使用一个GPU进行训练。另一个原因可能是,变形金刚应该用于大规模的预训练和微调范例。为了保持评估的一致性和公平性,我们从头开始训练所有网络,并在所有实验中使用相同的预处理和数据增强技术。我们相信社区协作和透明度的价值,因此,我们已经公开了我们的比较方法的实现,供研究社区进一步检查和使用。
此外,nnU-Net在大多数任务上显示出非常有竞争力的结果,但UMamba的总体得分更高。我们注意到,在CT和MRI扫描中,nnU-Net和U-Mamba对某些器官具有互补的分割性能,如主动脉、肾脏和下腔静脉,如附录表5-7所示。这表明通过模型集成进一步整合它们的潜力,这是一种经常用于提高医学图像分割比赛性能的策略[30]。
虽然这项工作的主要焦点是新的分割架构,但U-Mamba还有许多进一步增强和扩展的途径。
一个直接的方向是利用U-Mamba列车的大规模数据集,旨在创建易于部署的分割工具或为数据有限的任务提供预训练的模型权重,如TotalSegmentator[47]和STU-Net[21]等类似计划的例子。此外,U-Mamba的设计本质上支持与先进技术的集成,例如针对小数据集的强数据增强,针对高度不平衡目标的损失函数,以及针对嵌套对象的基于区域的训练[23]。此外,U-Mamba区块为分类和检测网络提供了一个新的应用机会,以获得更好的远程依赖建模。我们把这些方向作为近期的工作。
综上所述,本文提出了一种新的通用生物医学图像分割架构U-Mamba,该架构融合了cnn的局部模式识别和Mamba的全局上下文理解的优点。
U-Mamba可以自动配置不同的数据集,使其成为生物医学成像中各种分割任务的多功能和灵活的工具。结果表明,作为下一代生物医学图像分割网络的骨干,U-Mamba是一个有希望的候选者。
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:chenni525@qq.com进行投诉反馈,一经查实,立即删除!
- Python教程
- 深入理解 MySQL 中的 HAVING 关键字和聚合函数
- Qt之QChar编码(1)
- MyBatis入门基础篇
- 用Python脚本实现FFmpeg批量转换
- 02.阿里Java开发手册——日期时间
- 如何获取一个德国容器
- A 股承担着一个什么功能?
- MS8311/8312精密、低噪、CMOS、轨到轨输入输出运算放大器
- Java异常处理、自定义运行和编译异常及释放资源try-with-resouce
- [漏洞复现]Apache POI < 3.10.1 XXE(CVE-2014-3529)
- 2024--Django平台开发-Redis持久化、主从复制、哨兵(十)
- 为什么静态IP是您批量创建社交媒体和账户管理必备?
- MySQL死锁
- JavaWeb笔记之JSP