【数模百科】一文讲清楚主成分分析PCA算法(附python代码)
本文摘录自PCA原理 - 数模百科,如果想了解更多有关PCA的信息,请移步?主成分分析 - 数模百科
你有一堆数据,里面信息多得让人眼花缭乱,有些信息很重要,有些可能没那么关键。你想找出最关键的信息,但是又不想丢掉太多东西。主成分分析就是为了解决这个问题而生的。
这个方法的动机很简单:它帮助我们把那些乱糟糟的数据整理得井井有条,把最重要的信息放在前面,不那么重要的可以暂时放一边。这样,我们就能用更少的数据表达原来的大意,这个过程就像是给数据做“瘦身”。
在具体使用主成分分析的时候,我们不必深究它背后复杂的数学原理,你只需要知道它能帮你把原始数据转换成一种新的形式,这种新的形式能让数据中最有价值的部分更加突出。
比如说,在数学建模比赛里,你想要快速了解数据里面有什么奥秘,PCA能帮你找到关键线索。在图像处理方面,如果你想减小图片的大小,PCA能帮你压缩图像,让它占用更少的空间,但信息还是保留得差不多。在金融分析中,如果你想知道投资风险在哪里,PCA能帮你把那些复杂的金融数据简化,找出最重要的风险因素。
总之,无论是在数据分析、图像处理,还是金融评估,只要你手上有一大堆数据,需要找出里面最重要的部分,主成分分析都能帮到你。它就像是一个帮你理清思路的好帮手,让你在数据的海洋里游刃有余。
白话文——游戏人物测评
这是一个打怪游戏,每次打怪我们需要挑选合适的角色上阵,帮助我们赢得关卡。那么我们如何根据关卡去选择合适的角色呢?我们可以拉一个表格,把所有游戏角色的属性列出来。
这里面每个属性的含义是:
-
生命值:代表角色进入关卡的初始生命值。
-
攻击力:代表角色对怪物造成的攻击指数。
-
减伤力度:代表角色受到怪物的实际攻击=怪物攻击力 *(1-减伤力度)。
-
增伤力度:代表角色每次攻击力都会在上一次攻击力的基础上增加一定百分比数值。例如角色增伤力度为 y,第一次攻击怪物时造成的攻击力为初始攻击力的值 x,第二次攻击怪物时实际攻击力变为
,第二次实际攻击力为
。
| 生命值 | 攻击力 | 减伤力度 | 增伤力度 | |
|---|---|---|---|---|
| 角色A | 100 | 30 | 20% | 0% |
| 角色B | 60 | 40 | 40% | 5% |
| 角色C | 50 | 80 | 10% | 20% |
这些数值看得让人眼花缭乱,我相信你在玩游戏的时候一定不会为了通关而把这个表格拿着翻来覆去地研究,因为游戏里的角色肯定不止三个。现在让我们尝试简化这张表格,也就是简化每个角色的属性。
首先我们看看,哪些属性有比较强的关联。你可能会发现,"生命值"和"减伤力度"这两个属性共同表现了这个角色是否禁得住被怪物打,也就是"生命力"是否顽强。同时,"攻击力"和"增伤力度"体现了这个角色是否打怪物很强,也就是"战斗力"是否强。
现在,我们不再用四个不同的属性来描述每个角色,而是用两个新的维度:生命力和战斗力。
其实上面的工作就相当于主成分分析,我们从多个相关属性中提取最核心的特征,简化了数据,是我们能够更容易地理解和比较游戏中的角色。如果现在这个关卡的怪物打人很疼,我们可能考虑派生命力强的角色上场;如果怪物的血量很厚,我们就会优先考虑战斗力强的角色。
定义与详解
定义
PCA的数学定义是:一个正交化线性变换,把数据变换到一个新的坐标系统中,使得这一数据的任何投影的第一大方差在第一个坐标(称为第一主成分)上,第二大方差在第二个坐标(第二主成分)上,依次类推。
更具体地,新坐标系统的每一个坐标轴(主成分)都是原始数据空间中的一个方向,方差在第一个坐标轴(第一主成分)上最大,数据投影到第一主成分上的方差是最大的。第二个坐标轴(第二主成分)是与第一主成分正交且在剩余方向上具有最大方差的方向。以此类推,可以得到和数据维度一样多的主成分。
换句话说,PCA的目标是找到一个新的坐标系统,使得数据在这个系统中的方差(即数据的分散程度)被最大化地展现出来。在这个新坐标系统中,每个轴都是原始数据中方差最大的方向,且这些轴是相互正交(或者说垂直)的。
这么做的目标是通过降维(选择前k个主成分),在保留数据集中大部分变异性(信息)的同时,减少数据的复杂性或者噪声。
协方差
协方差(Covariance)是衡量两个变量之间变化趋势是否相同的统计指标。直白一点说,如果我们有两组数据,协方差可以告诉我们,这两组数据是不是"一荣俱荣,一损俱损"的关系。
假设我们有两个随机变量,分别叫做 X 和 Y。它们的协方差,记作 ,可以通过以下的公式计算得到:
这里, 和
分别是 X 和 Y 的平均值(或者说是期望值),而 E 代表的是期望值的操作,你可以简单理解为对括号里面内容的平均值。
如果这个计算出来的数是正的,说明当 X 大于它的平均值的时候,Y 通常也会大于它的平均值,它们是同步的。如果是负的,那就表示 X 和 Y 是反着走的,一个大于平均值的时候,另一个通常小于平均值。如果协方差接近于零,那就说明 X 和 Y 没有明显的同步或反向趋势。
协方差矩阵(Covariance Matrix)是一个矩阵,其中每个元素是各个向量元素之间的协方差。当数据不止两个变量时,我们可以构造一个协方差矩阵来描述这些变量之间的关系。如果我们有 n 个变量,那么协方差矩阵就会是一个 的矩阵,记为 C。在这个矩阵中,第 i 行第 j 列的元素表示的是第 i 个变量和第 j 个变量的协方差,计算公式如下:
这里 X[i] 和 X[j] 分别表示第 i 个和第 j 个随机变量。
在主成分分析(PCA)中,协方差矩阵发挥着至关重要的作用。我们通过计算数据的协方差矩阵,然后分析这个矩阵,可以找出数据的主成分。主成分其实就是那些有着最大协方差(即方差)的方向,它们代表了数据中变化最大的部分。简单来说,PCA就是通过协方差矩阵来找出哪些方向上的变化对整体数据影响最大。
推导过程
基于最小投影距离
假设我们有一个数据集,包含了 m 个 n 维的数据点 ,这些数据点已经进行了中心化处理,即数据点的均值为零(
)。中心化是为了简化后续计算,它确保了数据是围绕原点分布的。
我们的目标是将这些数据点投影到一个低维的空间中,这个空间由一组新的标准正交基 定义,其中 n' < n 。这个过程会减少数据的维度,同时尽量保留原始数据中的重要信息。
每个数据点 在新的坐标系中的投影表示为
其中 是数据点在新坐标系第 j 维上的坐标。
如果我们想将投影后的点 转换回原始的坐标系中,我们会得到一个"恢复"的数据点
,这里 W 是一个包含了 n' 个标准正交基向量的矩阵。
为了尽可能多地保留原始数据的信息,我们希望最小化所有数据点到投影平面的距离的平方和,即最小化以下目标函数:
这个公式表示的是所有数据点与它们的投影之间距离的平方和,我们想要这个和尽可能小。
我们可以使用拉格朗日乘数法来解决这个最小化问题,同时需要满足约束条件(即 W 的列向量是标准正交的)。我们构建拉格朗日函数:
其中 X 是一个 的矩阵,其第 i 行是
,
表示矩阵的迹,
是拉格朗日乘数,作为一个对角矩阵。
为了找到最小化函数的解,我们需要对 W 求导然后设为零:
这个方程说明了 W 由 的特征向量组成,而
由对应的特征值构成。
最终,PCA的推导表明,为了得到最优的投影,我们需要选择与协方差矩阵 最大特征值对应的特征向量构成的投影矩阵。这些特征向量称为主成分,它们定义了数据降维后的新坐标系。
基于最大投影方差
主成分分析(PCA)是一种常见的数据降维技术,目的是在减少数据维度的同时尽可能多地保留原始数据的信息。我们可以通过最大化投影方差的方式来推导PCA。以下是详细的推导过程:
假设我们有m个n维的数据点,这些数据点已经完成了中心化,即满足 \
。我们想要将这些数据点投影到一个新的坐标系
中,这个新坐标系由 n' 个标准化的正交基向量组成,其中
且对任意的
,有
。
在新的 n' 维坐标系中,每个样本点 的投影表示为:
其中, 是
在新坐标系中第 j 维的坐标。
对于每一个样本 ,它在新坐标系中的投影可以表示为
,其投影的方差是
。我们的目标是使得所有样本的投影方差之和最大。换句话说,我们想要最大化:
这等价于最大化迹(trace):
我们可以使用拉格朗日乘子法来求解这个问题,定义拉格朗日函数:
其中, 是拉格朗日乘子矩阵,它是一个对角矩阵,
是第 i 个拉格朗日乘子。
对 W 求导,并令导数为零,我们得到:
整理后我们得到:
这表明,W 的列向量是 的特征向量,而
是对应的特征值。当我们从 n 维降维到 n' 维时,我们需要选择最大的 n' 个特征值对应的特征向量。这 n' 个特征向量组成的矩阵 W 就是我们需要的新坐标系。
最后,对于原始数据集,我们可以通过计算 来将原始数据投影到新的 n' 维空间中,这样就实现了数据的降维,同时保留了最大的投影方差,即保留了数据中最重要的结构信息。
算法流程
-
数据中心化(去均值化): 首先,我们需要将数据集中的每个特征维度都进行中心化处理,即每个特征值都要减去该特征的平均值。这样做的目的是为了让数据在每个维度上的均值变为0。设我们有一个数据矩阵 X ,它的大小为
,其中 m 是样本数量,n 是特征数量。中心化后的数据矩阵记为
。
-
计算协方差矩阵: 接下来,我们要计算数据的协方差矩阵。协方差矩阵展示了各个特征之间的相关性,它的计算公式如下:
其中,
?是中心化数据矩阵
的转置。
-
计算特征值和特征向量: 协方差矩阵的特征值和特征向量给我们提供了数据的主成分(principal components)信息,也就是数据中变化最大的方向。通过求解特征方程:
我们可以得到协方差矩阵 C 的特征值
和对应的特征向量 W。
-
特征值排序及选择主成分: 将所有特征值从大到小排序,并选择前 k 个最大的特征值,以及与这些特征值对应的特征向量。这 k 个特征向量构成了投影矩阵
。
-
数据降维: 现在,我们可以使用投影矩阵
将原始的高维数据转换到 k 维的低维空间,这个新的低维数据记为 Z ,计算公式如下:
-
数据重构(可选): 如果我们想要将降维后的数据还原回原始的高维空间,我们可以使用以下公式进行数据重构:
其中,
是投影矩阵
的转置,
是原始数据每个特征的均值向量。这样就可以得到一个近似的原始数据集
。
通过上述步骤,我们可以用PCA算法对数据进行降维处理。
代码
以下是一个用Python实现PCA以及对结果的可视化的例子,数据点包括Iris数据集,这是一个经常用于演示机器学习算法的数据集,包括了150朵不同种类鸢尾花的四个特征:花萼长度、花萼宽度、花瓣长度、花瓣宽度。
import matplotlib.pyplot as plt
from sklearn.decomposition import PCA
from sklearn.datasets import load_iris
# 载入数据集
iris = load_iris()
X = iris.data
y = iris.target
# 执行PCA
pca = PCA(n_components=2)
X_r = pca.fit_transform(X)
print("explained variance ratio: ", pca.explained_variance_ratio_)
print("explained variance: ", pca.explained_variance_)
# 可视化结果
plt.figure()
colors = ['navy', 'turquoise', 'darkorange']
target_names = iris.target_names
lw = 2
for color, i, target_name in zip(colors, [0, 1, 2], target_names):
plt.scatter(X_r[y == i, 0], X_r[y == i, 1], color=color, alpha=.8,
lw=lw, label=target_name)
plt.legend(loc='best', shadow=False, scatterpoints=1)
plt.title('PCA of IRIS dataset')
plt.show()在以上代码中,首先加载了Iris数据集。然后使用了PCA,将数据集中的四个特征压缩成两个主成分,参数n_components=2意味着PCA会找出数据的前两个主成分。
explained_variance_ratio_给出了每个主成分的方差量占总方差量的比例,这可以看作是每个主成分的重要性。explained_variance_给出了每个主成分的方差量,这是一个绝对数字,也可以看作是每个主成分的重要性。
最后一部分的代码是进行可视化。新的数据(即主成分)被绘制在散点图上,不同颜色的点表示不同种类的花。尽管我们丢弃了一些数据(从4个特征压缩到2个主成分),不同种类的鸢尾花依然可以在二维空间上清楚地区分开来。
输出结果:
explained variance ratio: [0.92461872 0.05306648]
explained variance: [4.22824171 0.24267075]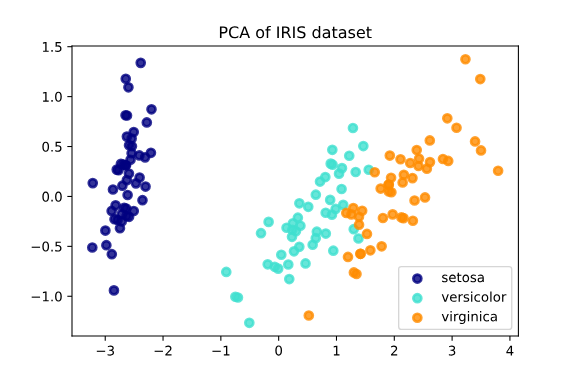
优缺点和应用
参照数模百科。
本文摘录自 数模百科 ——?主成分分析 - 数模百科
数模百科是一个由一群数模爱好者搭建的数学建模知识平台。我们想让大家只通过一个网站,就能解决自己在数学建模上的难题,把搜索和筛选的时间节省下来,投入到真正的学习当中。
我们团队目前正在努力为大家创建最好的信息集合,从用最简单易懂的话语和生动形象的例子帮助大家理解模型,到用科学严谨的语言讲解模型原理,再到提供参考代码。我们努力为数学建模的学习者和参赛者提供一站式学习平台,目前网站已上线,期待大家的反馈。
如果你想和我们的团队成员进行更深入的学习和交流,你可以通过公众号数模百科找到我们,我们会在这里发布更多资讯,也欢迎你来找我们唠嗑。
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:chenni525@qq.com进行投诉反馈,一经查实,立即删除!
- Python教程
- 深入理解 MySQL 中的 HAVING 关键字和聚合函数
- Qt之QChar编码(1)
- MyBatis入门基础篇
- 用Python脚本实现FFmpeg批量转换
- 什么是软件需求?以及需求的最佳实践?
- 【刷题日志】深度理解除(/)与取模(%)附水仙花数以及变种水仙花数题解
- 简述一种内部函数特征码识别定位方法
- 服务器中E5和I9的区别是什么,如何选择合适的配置
- 推荐!3D模型格式转换HOOPS Exchange轻松解决AutoVue多年数据难题!
- Oracle按日周月年自动分区
- python/c++ Leetcode题解——2744. 最大字符串配对数目
- 若依之自带@RateLimiter注解接口限流异常解决方案
- pandas缺失值查看及处理方法(后附源数据供大家练习)
- 自助式可视化开发,ETLCloud的集成之路