miRMaker
Introduction
除了miRNA表达数据,各种miRNA相关的知识也强有力地支持了对miRNA功能相互作用的理解。
那些具有许多共同调控靶基因或疾病的miRNAs可能具有相似的功能
一些方法通过考虑实验验证的miRNA-靶标关系来评估miRNA相互作用,评估miRNA功能相互作用的直观方法是通过Jaccard指数(Jaccard index)计算共同靶标的比例。但是,它丢失了大量的相似信息
杰卡德系数(Jaccard Index)
杰卡德系数,又称为杰卡德相似系数,用于比较两个样本之间的差异性和相似性。杰卡德系数越高,则两个样本相似度越高。
一些方法尝试使用基因本体注释或者)蛋白质-蛋白质相互作用(PPI)作为推断miRNA相互作用的靶基因的额外信息。
然而,GO数据库对大量靶基因的注释信息有限,不利于准确评价miRNA相互作用。同时,PPI数据库中存在大量不符合实验有效性的假阳性,这可能会给计算miRNA功能相似性带来偏差。
"Gene Ontology Annotations"(基因本体注释)是一种用于描述基因和蛋白质功能的标准化方法。基因本体是一个系统化的生物学术语表,它将基因和蛋白质的功能划分为三个主要的本体(ontology):分子功能(molecular function)、细胞组分(cellular component)和生物过程(biological process)。
总的来说,Gene Ontology Annotations 提供了一种标准化的方式来描述基因和蛋白质的功能,使得科研人员能够更好地理解生物体内各种生物学过程。
"Protein–protein interactions"(PPIs,蛋白质-蛋白质相互作用)是指在生物体内,两个或多个蛋白质之间发生的相互作用或结合。这种相互作用对于维持细胞结构、调控信号传导、执行代谢途径等生物学过程至关重要。?
总结:
miRNA与疾病的关系提供了miRNA参与疾病发生发展的直接证据,成为评价miRNA功能相互作用的有力选择。?
一种miRNA通常调控多种疾病的发生发展过程,一种疾病与多种miRNA相关。基于疾病的miRNA功能相似度计算的关键是准确评估疾病语义相似度。
Wang等人[23]提出了一种基于疾病有向无环图(DAG)的疾病语义相似度图推理方法。然而,基于DAG的疾病相似性通过平等地评估与根病具有相同距离的疾病的重要性而忽略了特定疾病的语义意义[24]。
因此,要获得高质量的miRNA功能相互作用,必须合理评估和充分利用miRNA-疾病关系中的疾病语义信息。
同时,利用miRNA表达数据和知识库中的信息,有利于构建稳健的miRNA相互作用网络。miRNA表达数据包含了不同样本组之间miRNA协同调控的变化信息。已知的miRNA与疾病的关系为miRNA功能相似性的评价提供了有力的支持。因此,将miRNA协同调控与miRNA功能相似性相结合,可以促进下游网络分析任务的开展,包括潜在的疾病生物标志物的鉴定。
定义基于分子相互作用网络的信息子网络是生物网络分析中的一个重要课题。一些方法采用聚类技术来划分生物网络。一般来说,需要预定义的簇号,但很难确定。一些方法采用启发式策略来识别重要模块。
Zhang等人[26]提出了基于网络的博弈论方法(NGTM),通过在基于博弈论的模块扩展中使用合作博弈论度量(Shapley值)评估特征贡献来识别潜在的癌症子网络生物标志物。但基于启发式的模块识别方法所使用的启发式信息有限,容易陷入局部最优。
与监督学习和无监督学习不同,强化学习(RL)旨在做出最大化长期回报的决策[27]。因此,RL策略可以通过充分探索解空间以获得全局最优结果来为模块生物标志物识别带来更多可能性。
Paim等人[28]尝试使用RL来检测复杂网络中的社区,并提出了Q-Learning [29] for Community Detection(QLCD)方法。网络中的每个节点充当一个代理,从其最近的邻居节点(动作空间)中选择一个节点组成集群。代理节点学习的行动策略,以最大限度地提高网络的模块化。然而,由于QLCD固有的简单动作空间和学习策略不足,在疾病研究中可能无法找到竞争模块。有必要进一步探索强化学习在定义疾病网络生物标志物方面的潜力。
为了有效识别潜在的miRNA疾病生物标志物,提出了基于多视图网络和强化学习的miRNA数据分析方法miRMarker。
- 基于表达数据构建miRNA协同调控网络。
- 利用公共知识库中已知的miRNA-疾病关系构建miRNA功能相似性网络。
- 然后,miRMarker整合两个miRNA网络,并通过强化学习策略定义关键的miRNA模块。
我们通过在9个转录组学数据集上与8种有效的数据分析方法进行比较,验证了miRMarker在疾病样本区分方面的有效性。
此外,我们检查了由miRMarker定义的结直肠癌的潜在miRNA模块生物标志物。实验结果表明miRMarker在确定疾病诊断和预后的重要模块生物标志物方面具有巨大的潜力。
材料和方法
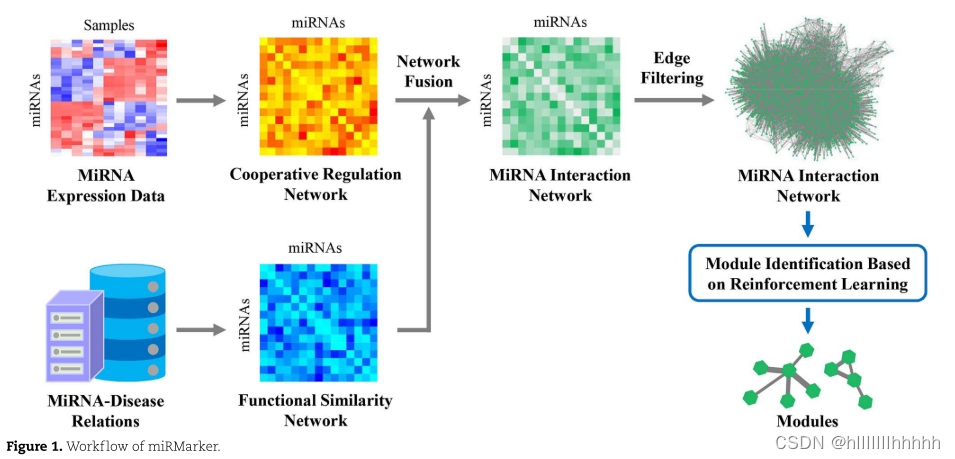
miRMarker由两个主要部分组成:
(i)分别基于miRNA表达数据和miRNA-疾病关系构建miRNA网络,整合两个网络;
(ii)通过强化学习策略定义关键的miRNA模块。图1显示了miRMarker的工作流程。
miRNA表达数据集
在这项研究中,收集了9个miRNA表达数据集,以评估miRMarker的有效性。所有数据集均来自公共数据库Gene Expression Omnibus(GEO),涉及多种疾病,如肝细胞癌、血小板增多症和结直肠癌。
表1给出了9个miRNA表达数据集的详细信息。数据集GSE 41574、GSE 67139、GSE 32273、GSE 34496、GSE 41282和GSE 108153是两类数据集。数据集GSE 31164、GSE 39046和GSE 35834是多类数据集。
我们使用miRBase v22.0将所有人类成熟miRNA名称映射到标准miRNA编号。将代表相同miRNA的探针的表达值平均。
因为同一个miRNA可能会被多个探针检测到,而这些探针的测量结果可能会有一些变化。通过取平均值,可以得到更为稳定和可靠的miRNA表达量。
miRNA与疾病的关系?
从两个最大的知识库miRCancer [20]和miR 2Disease [21]中提取手动策划的miRNA-疾病关系。
miRCancer使用文本挖掘技术从PubMed数据库中的医学文献中提取miRNA-癌症关联,然后手动修改关联。
"miR2Disease提供了从已发表的论文中整理出的miRNA与人类疾病之间的全面调控关联信息。
我们下载了miRCancer(9080个条目,于2022年6月下载)和miR2Disease(2877个条目,于2022年7月下载)的最新版本。
所有人类成熟miRNA的名称都通过miRBase映射到标准miRNA存取号。将关系中的疾病名称映射到MeSH中的规范疾病术语(于2022年7月下载)。
我们整合了miRCancer和miR 2Disease的miRNA-疾病相互作用,并消除了重复条目。最终获得了6099个miRNA与疾病的关系,涉及163种人类疾病。
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:chenni525@qq.com进行投诉反馈,一经查实,立即删除!