【Python机器学习系列】建立随机森林模型预测心脏疾病(完整实现过程)
这是Python机器学习系列原创文章,我的第200篇原创文章。
一、引言
????????对于表格数据,一套完整的机器学习建模流程如下:
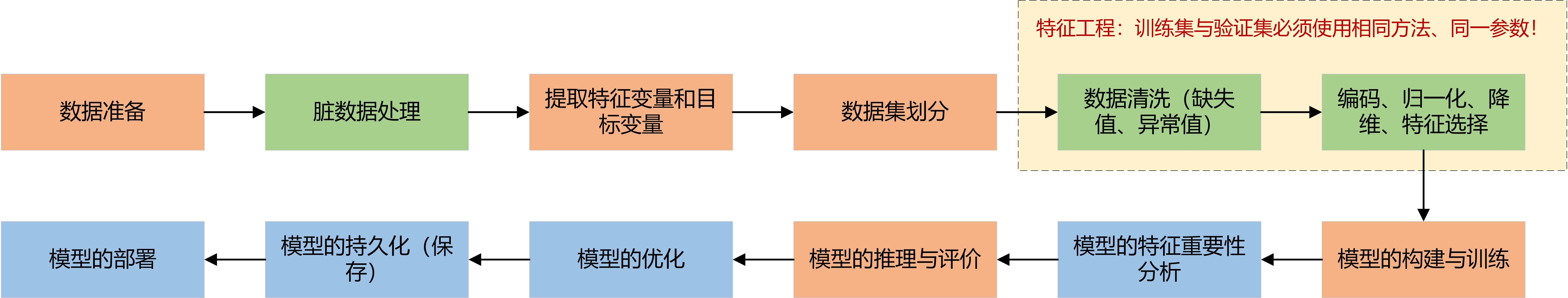
????? ? 针对不同的数据集,有些步骤不适用即不需要做,其中橘红色框为必要步骤,由于数据质量较高,本文有些步骤跳过了,跳过的步骤将单独出文章总结!同时欢迎大家关注翻看我之前的一些相关文章。
【Python机器学习系列】一文彻底搞懂机器学习中表格数据的输入形式(理论+源码)
【Python机器学习系列】一文带你了解机器学习中的Pipeline管道机制(理论+源码)
【Python机器学习系列】一文搞懂机器学习中的转换器和估计器(附案例)
【Python机器学习系列】一文讲透机器学习中的K折交叉验证(源码)
【Python特征工程系列】利用随机森林模型分析特征重要性(源码)
【Python特征工程系列】8步教你用决策树模型分析特征重要性(源码)
【Python特征工程系列】教你利用逻辑回归模型分析特征重要性(源码)
【Python特征工程系列】教你利用AdaBoost模型分析特征重要性(源码)
【Python特征工程系列】教你利用XGBoost模型分析特征重要性(源码)
【Python特征工程系列】利用梯度提升(GradientBoosting)模型分析特征重要性(源码)
【Python机器学习系列】拟合和回归傻傻分不清?一文带你彻底搞懂它
【Python机器学习系列】建立决策树模型预测心脏疾病(完整实现过程)
【Python机器学习系列】建立支持向量机模型预测心脏疾病(完整实现过程)
【Python机器学习系列】建立逻辑回归模型预测心脏疾病(完整实现过程)
【Python机器学习系列】建立KNN模型预测心脏疾病(完整实现过程)
????????随机森林(Random Forest)是一种集成学习(Ensemble Learning)方法,它结合了多个决策树(Decision Tree)来进行分类和回归任务。随机森林通过对训练数据进行自助采样(Bootstrap Sampling)和随机特征选择,构建多个决策树,并通过投票或平均的方式进行预测。
本文将实现基于心脏疾病数据集建立随机森林模型对心脏疾病患者进行分类预测的完整过程。
二、实现过程
导入必要的库
import pandas as pd
import matplotlib.pyplot as plt
from sklearn.utils import shuffle
from sklearn.model_selection import train_test_split
from sklearn.ensemble import RandomForestClassifier
from sklearn.metrics import accuracy_score
from sklearn.metrics import roc_curve
from sklearn.metrics import auc
from sklearn.metrics import confusion_matrix
from sklearn.metrics import classification_report1、准备数据
data = pd.read_csv(r'Dataset.csv')
df = pd.DataFrame(data)df:
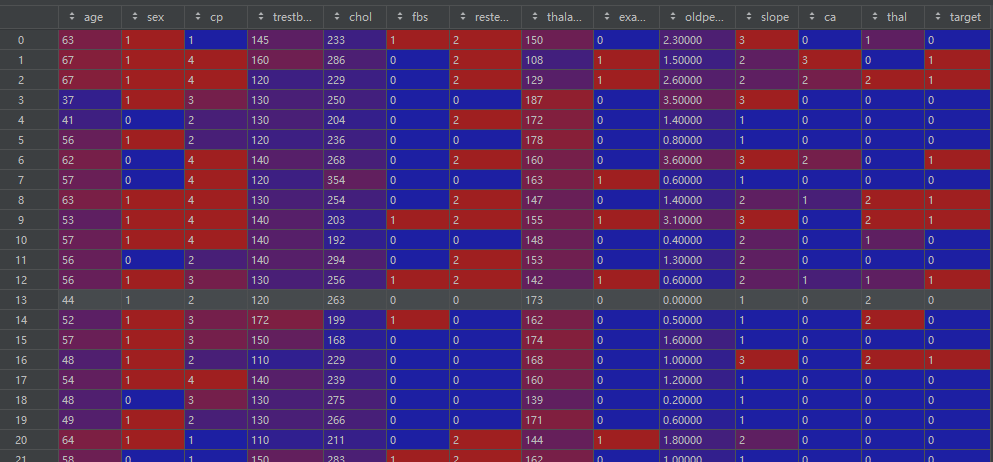
数据基本信息:
print(df.head())
print(df.info())
print(df.shape)
print(df.columns)
print(df.dtypes)
cat_cols = [col for col in df.columns if df[col].dtype == "object"] # 类别型变量名
num_cols = [col for col in df.columns if df[col].dtype != "object"] # 数值型变量名2、提取特征变量和目标变量
target = 'target'
features = df.columns.drop(target)
print(data["target"].value_counts())?#?顺便查看一下样本是否平衡3、数据集划分
# df = shuffle(df)
X_train, X_test, y_train, y_test = train_test_split(df[features], df[target], test_size=0.2, random_state=0)4、模型的构建与训练
# 模型的构建与训练
model = RandomForestClassifier()
model.fit(X_train, y_train)参数详解:
from sklearn.ensemble import RandomForestClassifier
# 全部参数
RandomForestClassifier(bootstrap=True, ccp_alpha=0.0, class_weight=None,
criterion='gini', max_depth=None, max_features='auto',
max_leaf_nodes=None, max_samples=None,
min_impurity_decrease=0.0, min_impurity_split=None,
min_samples_leaf=1, min_samples_split=2,
min_weight_fraction_leaf=0.0, n_estimators=100,
n_jobs=None, oob_score=False, random_state=None,
???????????????????????verbose=0,?warm_start=False)-
n_estimators:森林中决策树的数量。默认100。。
-
criterion:分裂节点所用的标准,可选“gini”, “entropy”,默认“gini”。
-
max_depth:树的最大深度。
-
min_samples_split:拆分内部节点所需的最少样本数。
-
min_samples_leaf:在叶节点处需要的最小样本数。
-
min_weight_fraction_leaf:在所有叶节点处(所有输入样本)的权重总和中的最小加权分数。
-
max_features:寻找最佳分割时要考虑的特征数量。
-
max_leaf_nodes:最大叶子节点数,整数,默认为None
-
min_impurity_decrease:如果分裂指标的减少量大于该值,则进行分裂。
-
min_impurity_split:决策树生长的最小纯净度。默认是0。自版本0.19起不推荐使用。
-
bootstrap:是否进行bootstrap操作,bool。默认True。
-
oob_score:是否使用袋外样本来估计泛化精度。默认False。
-
n_jobs:并行计算数。默认是None。
-
random_state:控制bootstrap的随机性以及选择样本的随机性。
-
verbose:在拟合和预测时控制详细程度。默认是0。
-
class_weight:每个类的权重,可以用字典的形式传入{class_label: weight}。
-
ccp_alpha:将选择成本复杂度最大且小于ccp_alpha的子树。默认情况下,不执行修剪。
-
max_samples:如果bootstrap为True,则从X抽取以训练每个基本分类器的样本数。。
5、模型的推理与评价
y_pred = model.predict(X_test)
y_scores = model.predict_proba(X_test)
acc = accuracy_score(y_test, y_pred) # 准确率acc
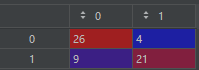
cm = confusion_matrix(y_test, y_pred) # 混淆矩阵
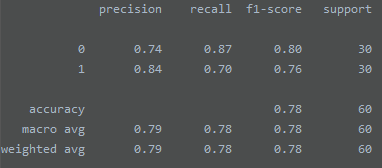
cr = classification_report(y_test, y_pred) # 分类报告
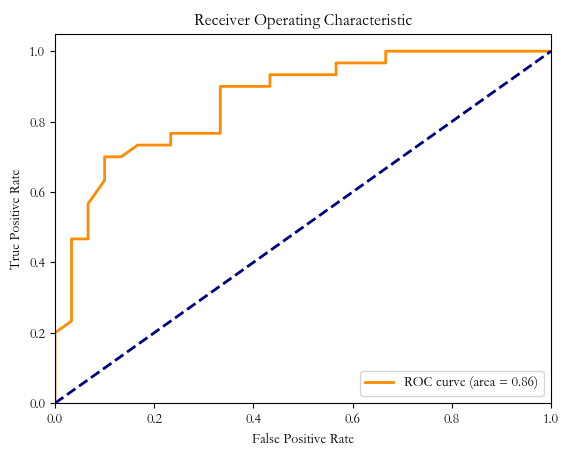
fpr, tpr, thresholds = roc_curve(y_test, y_scores[:, 1], pos_label=1) # 计算ROC曲线和AUC值,绘制ROC曲线
roc_auc = auc(fpr, tpr)
plt.figure()
plt.plot(fpr, tpr, color='darkorange', lw=2, label='ROC curve (area = %0.2f)' % roc_auc)
plt.plot([0, 1], [0, 1], color='navy', lw=2, linestyle='--')
plt.xlim([0.0, 1.0])
plt.ylim([0.0, 1.05])
plt.xlabel('False Positive Rate')
plt.ylabel('True Positive Rate')
plt.title('Receiver Operating Characteristic')
plt.legend(loc="lower right")
plt.show()cm:
cr:
ROC:
三、小结
本文利用scikit-learn(一个常用的机器学习库)实现了基于心脏疾病数据集建立随机森林模型对心脏疾病患者进行分类预测的完整过程。
作者简介:
读研期间发表6篇SCI数据挖掘相关论文,现在某研究院从事数据算法相关科研工作,结合自身科研实践经历不定期分享关于Python、机器学习、深度学习、人工智能系列基础知识与应用案例。致力于只做原创,以最简单的方式理解和学习,关注我一起交流成长。需要数据集和源码的小伙伴可以关注底部公众号添加作者微信! ??
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:chenni525@qq.com进行投诉反馈,一经查实,立即删除!