欠拟合与过拟合

欠拟合:
????????模型在训练集上表现不好,在测试集上也表现不好。模型过于简单
????????欠拟合在训练集和测试集上的误差都较大
通过代码展示欠拟合
import numpy as np
import matplotlib.pyplot as plt
from sklearn.linear_model import LinearRegression
from sklearn.metrics import mean_squared_error # 计算均方误差
from sklearn.model_selection import train_test_split
#%%
# 欠拟合
np.random.seed(666) # 设置随机数种子 如果写了一样的种子保证每次生成的随机数都是一样的
x = np.random.uniform(-3,3,size=100)
y =0.5*x**2 +x+2+np.random.normal(0,1,size=100)
# np.random.uniform 生成均匀分布的随机数
# np.random.normal 生成正态分布随机数 均值为0 , 标准差1
estimator = LinearRegression()
X = x.reshape(-1,1)
estimator.fit(X,y)
y_pred = estimator.predict(X)
print('均方误差:',mean_squared_error(y_pred,y))
plt.scatter(x, y)
# plt.plot(x, y_pred,color='r')
# 画图时输入的x数据: 要求是从小到大
plt.plot(np.sort(x), y_pred[np.argsort(x)], color='r')
plt.show()
?数据是抛物线非线性的, 用线性模型去拟合.。 模型过于简单,出现欠拟合
欠拟合原因
学习到数据的特征过少
解决方法
添加其他特征
????????有时出现欠拟合是因为特征项不够导致的,可以添加其他特征项来解决
????????“组合”、“泛化”、“相关性”三类特征是特征添加的重要手段
添加多项式特征项
????????模型过于简单时的常用套路,例如将线性模型通过添加二次项或三次项使模型泛化能力更强
# 欠拟合解决
np.random.seed(666) # 设置随机数种子 如果写了一样的种子保证每次生成的随机数都是一样的
x = np.random.uniform(-3,3,size=100)
y =0.5*x**2 +x+2+np.random.normal(0,1,size=100)
# np.random.uniform 生成均匀分布的随机数
# np.random.normal 生成正态分布随机数 均值为0 , 标准差1
estimator = LinearRegression()
X = x.reshape(-1,1)
X2 = np.hstack([X,X**2])
estimator.fit(X2,y)
y_pred2 = estimator.predict(X2)
print('均方误差:',mean_squared_error(y_pred2,y))
plt.scatter(x, y)
# plt.plot(x, y_pred2,color='r')
# 画图时输入的x数据: 要求是从小到大
plt.plot(np.sort(x), y_pred2[np.argsort(x)], color='r')
plt.show()过拟合:
????????模型在训练集上表现好,在测试集上表现不好。模型过于复杂
????????过拟合在训练集上误差较小,而测试集上误差较大
通过代码展示过拟合
import numpy as np
import matplotlib.pyplot as plt
from sklearn.linear_model import LinearRegression
from sklearn.metrics import mean_squared_error # 计算均方误差
from sklearn.model_selection import train_test_split
# 过拟合
np.random.seed(666) # 设置随机数种子 如果写了一样的种子保证每次生成的随机数都是一样的
x = np.random.uniform(-3,3,size=100)
y =0.5*x**2 +x+2+np.random.normal(0,1,size=100)
# np.random.uniform 生成均匀分布的随机数
# np.random.normal 生成正态分布随机数 均值为0 , 标准差1
estimator = LinearRegression()
X = x.reshape(-1,1)
X3 = np.hstack([X, X**2, X**3, X**4, X**5, X**6, X**7, X**8, X**9, X**10]) # 数据增加高次项
estimator.fit(X3,y)
y_pred2 = estimator.predict(X3)
print('均方误差:',mean_squared_error(y_pred2,y))
plt.scatter(x, y)
# plt.plot(x, y_pred2,color='r')
# 画图时输入的x数据: 要求是从小到大
plt.plot(np.sort(x), y_pred2[np.argsort(x)], color='r')
plt.show()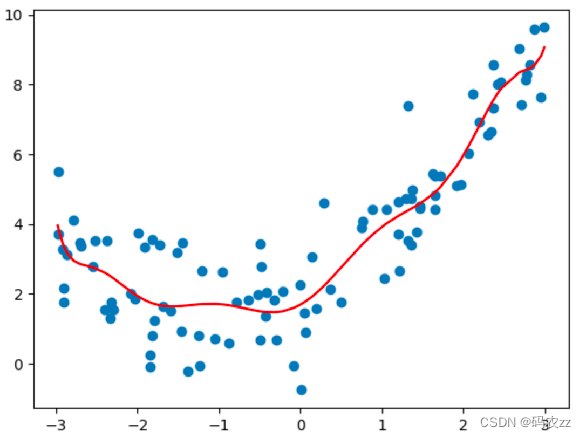
?过拟合原因
原始特征过多,存在一些嘈杂特征, 模型过于复杂是因为模型尝试去兼顾各个测试数据点
解决方法
重新清洗数据
????????对于过多异常点数据、数据不纯的地方再处理
增大数据的训练量
???????? 对原来的数据训练的太过了,增加数据量的情况下,会缓解
正则化
????????解决模型过拟合的方法,在机器学习、深度学习中大量使用
减少特征维度,防止维灾难
????????由于特征多,样本数量少,导致学习不充分,泛化能力差。
正则化
在模型训练时,数据中有些特征影响模型复杂度、或者某个特征的异常值较多, ? ? ? 所以要尽量减少这个特征的影响(甚至删除某个特征的影响),这就是正则化
在损失函数中增加正则化项 分为L1正则化、L2正则化, 消除异常点带来的w值过大过小的影响
解决方式1:
L1正则化,在损失函数中添加L1正则化项

α 叫做惩罚系数,该值越大则权重调整的幅度就越大,即:表示对特征权重惩罚力度就越大
# 过拟合解决L1
from sklearn.linear_model import Lasso
np.random.seed(666) # 设置随机数种子 如果写了一样的种子保证每次生成的随机数都是一样的
x = np.random.uniform(-3,3,size=100)
y =0.5*x**2 +x+2+np.random.normal(0,1,size=100)
# np.random.uniform 生成均匀分布的随机数
# np.random.normal 生成正态分布随机数 均值为0 , 标准差1
estimator = Lasso(alpha=0.005,normalize=True)
X = x.reshape(-1,1)
X3 = np.hstack([X, X**2, X**3, X**4, X**5, X**6, X**7, X**8, X**9, X**10]) # 数据增加高次项
estimator.fit(X3,y)
y_pred2 = estimator.predict(X3)
print('均方误差:',mean_squared_error(y_pred2,y))
plt.scatter(x, y)
# plt.plot(x, y_pred2,color='r')
# 画图时输入的x数据: 要求是从小到大
plt.plot(np.sort(x), y_pred2[np.argsort(x)], color='r')
plt.show()数据是抛物线形状的, 给模型送入的数据,增加x^2、x^3、x^4 …高次项特征, 再用线性模型去拟合。模型过于复杂,出现过拟合
解决方式2:
L2正则化,在损失函数中添加L2正则化项
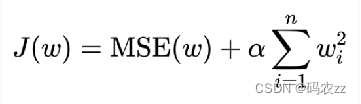
# 过拟合解决L2
from sklearn.linear_model import Ridge
np.random.seed(666) # 设置随机数种子 如果写了一样的种子保证每次生成的随机数都是一样的
x = np.random.uniform(-3,3,size=100)
y =0.5*x**2 +x+2+np.random.normal(0,1,size=100)
# np.random.uniform 生成均匀分布的随机数
# np.random.normal 生成正态分布随机数 均值为0 , 标准差1
estimator = Ridge(alpha=0.005,normalize=True)
X = x.reshape(-1,1)
X3 = np.hstack([X, X**2, X**3, X**4, X**5, X**6, X**7, X**8, X**9, X**10]) # 数据增加高次项
estimator.fit(X3,y)
y_pred2 = estimator.predict(X3)
print('均方误差:',mean_squared_error(y_pred2,y))
plt.scatter(x, y)
# plt.plot(x, y_pred2,color='r')
# 画图时输入的x数据: 要求是从小到大
plt.plot(np.sort(x), y_pred2[np.argsort(x)], color='r')
plt.show()本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:chenni525@qq.com进行投诉反馈,一经查实,立即删除!
- Python教程
- 深入理解 MySQL 中的 HAVING 关键字和聚合函数
- Qt之QChar编码(1)
- MyBatis入门基础篇
- 用Python脚本实现FFmpeg批量转换
- 配置计算云环境
- 深入探索谷歌浏览器开发者工具:提升网页开发与调试效率的终极指南(一)
- springboot/java/php/node/python银行信贷管理系统【计算机毕设】
- JavaSE变量 常量和数据类型转化
- .net6使用Sejil可视化日志
- [网络编程]UDP协议,基于UDP协议的回显服务器
- 如何彻底卸除Microsoft Edge浏览器
- linux系统使用POSIX信号量实现多线程同步sem_init sem_wait sem_post sem_destroy
- Next.js 学习笔记(三)——路由
- 此man非man的意思