基于综合特征的细菌噬菌体宿主预测工具iPHoP (Integrated Phage HOst Prediction)的介绍以及使用方法详细流程
介绍
iPHoP(Integrated Phage HOst Prediction)是一种基于综合特征的细菌噬菌体宿主预测方法。它是通过整合基因组序列、蛋白质序列和宿主基因组信息来预测细菌噬菌体的宿主范围。
iPHoP的预测过程分为三个步骤:特征提取、特征选择和宿主预测。在特征提取阶段,iPHoP会从噬菌体基因组和宿主基因组中提取一系列特征,包括基因组特征、蛋白质特征和宿主基因组特征。
在特征选择阶段,iPHoP使用机器学习算法从提取的特征中选择最具有预测能力的特征。常用的特征选择算法包括卡方检验、互信息和方差分析等。
在宿主预测阶段,iPHoP使用选择的特征来构建预测模型,通过对未知噬菌体进行预测,确定它们可能的宿主范围。
iPHoP具有以下特点:它是一种集成的预测方法,可以同时使用多个特征进行预测;它基于机器学习算法,可以根据不同的数据集进行预测;它能够预测细菌噬菌体的宿主范围,并提供预测的可靠性评估。
iPHoP已经在一些实验证明了其预测的准确性和可靠性,并且被广泛应用于细菌噬菌体宿主的研究中。
Overview
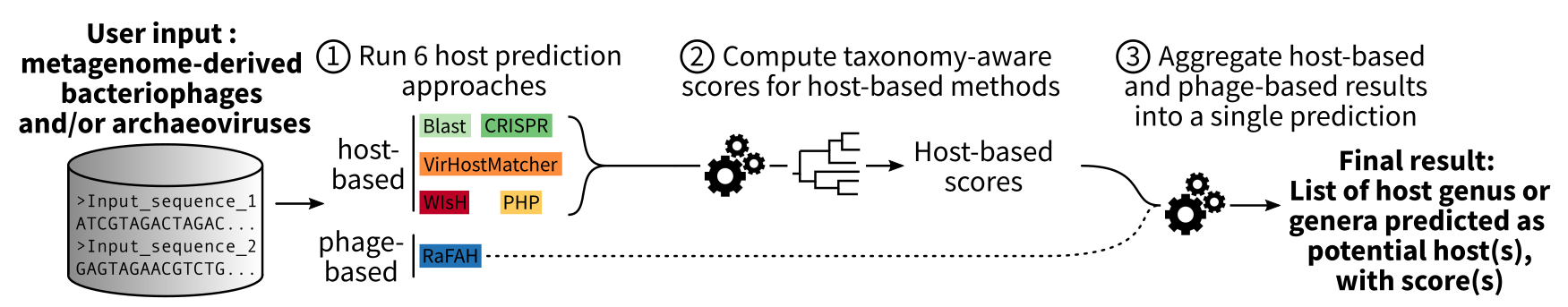
iPHoP?stands for?integrated?Phage?Host?Prediction. It is an automated command-line pipeline for predicting host genus of novel bacteriophages and archaeoviruses based on their genome sequences.
The pipeline can be broken down into 6 main steps:
仓库:srouxjgi / iphop — Bitbucket
下载代码库
git clone https://bitbucket.org/srouxjgi/iphop.git大致的使用流程
A: 第1步:运行单个宿主预测工具
基于噬菌体的工具: RaFAH(https://doi.org/10.1016/j.patter.2021.100274):生成宿主属的预测结果及相应的评分,保存以备后续步骤5使用
宿主基础工具:
- 使用blastn对宿主基因组进行比对。所有匹配度≥80%、长度≥500bp的片段被考虑在内。若匹配片段覆盖了“宿主”contig长度的≥50%,则忽略这些片段,因为它们往往源自几乎完全为病毒的contigs,可能源于基因组或MAGs中的污染,因此对于宿主预测不可靠。
- 使用blastn对CRISPR间隔序列数据库进行比对。所有最多允许4个错配的匹配结果都被考虑。
- WIsH(https://doi.org/10.1093/bioinformatics/btx383):基于病毒与宿主基因组k-mer组成相似性判断宿主关联
- VHM-s2* 相似性(Alignment-free |$d_2^*$| oligonucleotide frequency dissimilarity measure improves prediction of hosts from metagenomically-derived viral sequences | Nucleic Acids Research | Oxford Academic?/?https://doi.org/10.1093/nargab/lqaa044):基于病毒与宿主基因组k-mer组成相似性判断宿主关联
- PHP(https://doi.org/10.1186/s12915-020-00938-6):基于病毒与宿主基因组k-mer组成相似性判断宿主关联
B: 第2步:收集所有得分和基于宿主工具的所有命中之间的距离 * 对于两个潜在宿主(即,给定工具和查询病毒的两个匹配结果),距离是基于GTDB树(https://doi.org/10.1093/nar/gkab776)计算得出的。
C: 步骤3和4:为每种病毒 - 工具 - 候选宿主组合编制组织有序的命中列表 * 对于每个命中结果,将从同一病毒且使用相同工具获取的其他顶级命中结果进行汇总,并根据基线宿主与其他命中宿主之间的距离进行排序(参见步骤2)。 * 这些系列命中的结果被用作自动化分类器的输入,以推导出给定病毒 - 候选宿主对的评分。 * 这使得在评估每一个潜在宿主(每个命中结果)时,能够考虑到该病毒所获得的顶级命中结果的上下文信息。
D: 第5步:为每种病毒 - 候选宿主组合得出宿主基础工具的3个评分 * 仅基于blast或crispr匹配的顶级评分会被保留,因为这些方法本身在宿主预测上就足够可靠。 * 第三个评分通过考虑来自所有单独分类器的所有得分(参见步骤4)得出,即同时考虑所有5种宿主基础方法。
E: 第6步:计算每种病毒 - 候选宿主属组合的综合评分,整合宿主基础信号和噬菌体基础信号 * 将3个宿主基础评分(参见步骤5)与噬菌体基础评分(RaFAH - https://doi.org/10.1016/j.patter.2021.100274)结合,为所有病毒 - 候选宿主属对获得一个单一的综合评分。
conda安装
conda create -c conda-forge -n iphop_env python=3.8
conda activate iphop_env
mamba install -c conda-forge -c bioconda iphopmamba没有的大家自己使用conda?安装一下就行,怎么配置conda基础环境可以参考:
轻快小miniconda3在linux下的安装配置-centos9stream-Miniconda3 Linux 64-bit_离线安装miniconde linux-CSDN博客
数据库下载
iphop download --db_dir path_to_iPHoP_db
# 验证
iphop download --db_dir path_to_iPHoP_db --full_verify手动下载:
wget https://portal.nersc.gov/cfs/m342/iphop/db/iPHoP.latest_rw.tar.gz
tar -zxvf iPHoP.latest_rw.tar.gz开始使用
超级简单直接运行
iphop predict --fa_file my_input_phages.fasta --db_dir path/to/iphop_db/Sept_2021_pub/ --out_dir iphop_output/主要输出结果文件:
Main output files
Host_prediction_to_genus_mXX.csv, where XX is the minimum score cutoff selected (default: Host_prediction_to_genus_m90.csv)
This contains integrated results from host-based and phage-based tools at the host genus level:
| Virus | AAI to closest RaFAH reference | Host genus | Confidence score | List of methods |
|---|---|---|---|---|
| IMGVR_UViG_3300029435_000002 | 48.49 | d__Bacteria;p__Bacteroidota;c__Bacteroidia;o__Bacteroidales;f__Bacteroidaceae;g__Prevotella | 98.50 | RaFAH;91.30 iPHoP-RF;89.50 CRISPR;70.20 |
| IMGVR_UViG_3300029435_000003 | 53.00 | d__Bacteria;p__Firmicutes_A;c__Clostridia;o__Lachnospirales;f__Lachnospiraceae;g__Agathobacter | 92.20 | blast;94.40 |
| IMGVR_UViG_3300029435_000003 | 53.00 | d__Bacteria;p__Firmicutes_A;c__Clostridia;o__Lachnospirales;f__Lachnospiraceae;g__Bacteroides_F | 90.90 | CRISPR;93.30 iPHoP-RF;51.70 |
| IMGVR_UViG_3300029435_000005 | 42.95 | d__Bacteria;p__Firmicutes_A;c__Clostridia;o__Oscillospirales;f__Ruminococcaceae;g__Gemmiger | 95.30 | blast;96.70 CRISPR;92.70 iPHoP-RF;82.50 |
| IMGVR_UViG_3300029435_000007 | 35.09 | d__Bacteria;p__Bacteroidota;c__Bacteroidia;o__Bacteroidales;f__Bacteroidaceae;g__Prevotella | 98.40 | CRISPR;98.80 iPHoP-RF;95.40 blast;93.60 |
| IMGVR_UViG_3300029435_000009 | 99.62 | d__Bacteria;p__Firmicutes_A;c__Clostridia;o__Lachnospirales;f__Lachnospiraceae;g__Lachnospira | 99.00 | CRISPR;98.80 blast;92.60 iPHoP-RF;70.90 RaFAH;65.80 |
| IMGVR_UViG_3300029435_000009 | 99.62 | d__Bacteria;p__Firmicutes_A;c__Clostridia;o__Lachnospirales;f__Lachnospiraceae;g__Roseburia | 95.70 | CRISPR;97.00 iPHoP-RF;56.80 |
| IMGVR_UViG_3300029435_000010 | 22.47 | d__Bacteria;p__Proteobacteria;c__Gammaproteobacteria;o__Burkholderiales;f__Burkholderiaceae;g__Sutterella | 97.60 | blast;98.30 CRISPR;80.00 iPHoP-RF;78.30 |
- This output file lists for each prediction the virus sequence ID, the level of amino-acid similarity (AAI) between the query and the genomes in the RaFAH phage database, the predicted host genus, the confidence score calculated from all tools, and the list of scores for individual classifiers obtained for this virus-host pair.
- For the detailed score by classifier, "RaFAH" represents the score derived from RaFAH (https://www.sciencedirect.com/science/article/pii/S2666389921001008), iPHoP-RF is the score derived from all host-based tools, CRISPR the score derived only from CRISPR hits, and blast the score derived only from blastn hits
- All virus-host pairs for which the confidence score is higher than the selected cutoff (default = 90) are included, so each virus may be associated with multiple predictions (e.g. IMGVR_UViG_3300029435_000003 and IMGVR_UViG_3300029435_000009).
其他注意事项:
注意事项:我们建议所有用户首先使用标准数据库对相同的病毒序列运行iPHoP。同时,强烈建议仔细筛查所有MAGs(宏基因组组装基因组)以剔除污染,因为微生物MAGs中错误地归类的病毒contig可能导致高置信度的错误宿主预测。
注意事项:对于版本小于1.2.0的iPHoP,在添加自定义MAGs时,需要的是GTDB-tk v1.5.0的输出结果,目前与GTDB-tk v2的输出不兼容。但在1.2及更高版本中,这一问题应该已经得到了修复。
用户可以将他们自己的MAGs添加到宿主数据库中,例如从获取输入噬菌体的相同数据集或采样地点获得的MAGs。iPHoP中的"add_to_db"模块可用于此目的,需要为每个MAG提供fasta文件以及应用在这些相同MAG上的"gtdb-tk infer"功能的结果。示例文件集可在https://bitbucket.org/srouxjgi/iphop/downloads/Data_test_add_to_db.tar.gz 获取,基于Dalcin Martins等人发表的研究“Viral and metabolic controls on high rates of microbial sulfur and carbon cycling in wetland ecosystems”的数据。
要将MAGs添加到宿主数据库的完整过程如下:
使用wget下载示例数据包:
wget https://bitbucket.org/srouxjgi/iphop/downloads/Data_test_add_to_db.tar.gz解压下载的数据包:
tar -xvf Data_test_add_to_db.tar.gz?查看解压后的目录内容:
ls Data_test_add_to_db其中,“Expected_results/”文件夹包含了使用Sept_2021_pub数据库或包含额外MAGs的新数据库时iPHoP的预期结果文件。“Input_viral_contigs.fasta”是输入文件。“Wetland_MAGs/”文件夹包含了所有MAGs的fasta文件。“Wetland_MAGs_GTDB-tk_results/”文件夹则包含了iPHoP将使用的gtdb-tk结果文件。
生成gtdb-tk结果文件
gtdbtk de_novo_wf --genome_dir Wetland_MAGs/ --bacteria --outgroup_taxon p__Patescibacteria --out_dir Wetland_MAGs_GTDB-tk_results/ --cpus 32 --force --extension fa
gtdbtk de_novo_wf --genome_dir Wetland_MAGs/ --archaea --outgroup_taxon p__Altarchaeota --out_dir Wetland_MAGs_GTDB-tk_results/ --cpus 32 --force --extension fa创建一个新的iPHoP数据库,该数据库将包括GTDB基因组和用户提供的额外MAGs,但不包括GEM或IMG基因组
cd Data_test_add_to_db
iphop add_to_db --fna_dir Wetland_MAGs/ --gtdb_dir Wetland_MAGs_GTDB-tk_results/ --out_dir Sept_2021_pub_rw_w_Wetland_hosts --db_dir /path/to/iphop_db/Sept_2021_pub_rw/?注意:为了避免复制大量文件,新数据库部分基于原始数据库的符号链接。这意味着如果原始数据库(此处为 "iphop_db/Sept_2021_pub/")被修改或删除,新的数据库也将无法正常工作。这也意味着应提供原始数据库的完整路径作为 "db_dir" 参数。
然后,可以使用"Sept_2021_pub_w_Wetland_hosts"文件夹作为iPHoP数据库进行宿主预测操作,例如:
iphop predict --fa_file Input_viral_contigs.fasta --db_dir Sept_2021_pub_rw_w_Wetland_hosts/ --out_dir test_add_db -t 4引用信息
@article{roux_iphop_2023,
abstract = {The extraordinary diversity of viruses infecting bacteria and archaea is now primarily studied through metagenomics. While metagenomes enable high-throughput exploration of the viral sequence space, metagenome-derived sequences lack key information compared to isolated viruses, in particular host association. Different computational approaches are available to predict the host(s) of uncultivated viruses based on their genome sequences, but thus far individual approaches are limited either in precision or in recall, i.e., for a number of viruses they yield erroneous predictions or no prediction at all. Here, we describe iPHoP, a two-step framework that integrates multiple methods to reliably predict host taxonomy at the genus rank for a broad range of viruses infecting bacteria and archaea, while retaining a low false discovery rate. Based on a large dataset of metagenome-derived virus genomes from the IMG/VR database, we illustrate how iPHoP can provide extensive host prediction and guide further characterization of uncultivated viruses.},
author = {Roux, Simon and Camargo, Antonio Pedro and Coutinho, Felipe H. and Dabdoub, Shareef M. and Dutilh, Bas E. and Nayfach, Stephen and Tritt, Andrew},
doi = {10.1371/journal.pbio.3002083},
issn = {1545-7885},
journal = {PLOS Biology},
number = {4},
title = {{iPHoP}: {An} integrated machine learning framework to maximize host prediction for metagenome-derived viruses of archaea and bacteria},
volume = {21},
year = {2023},
}
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:chenni525@qq.com进行投诉反馈,一经查实,立即删除!