Pytorch项目,肺癌检测项目之二
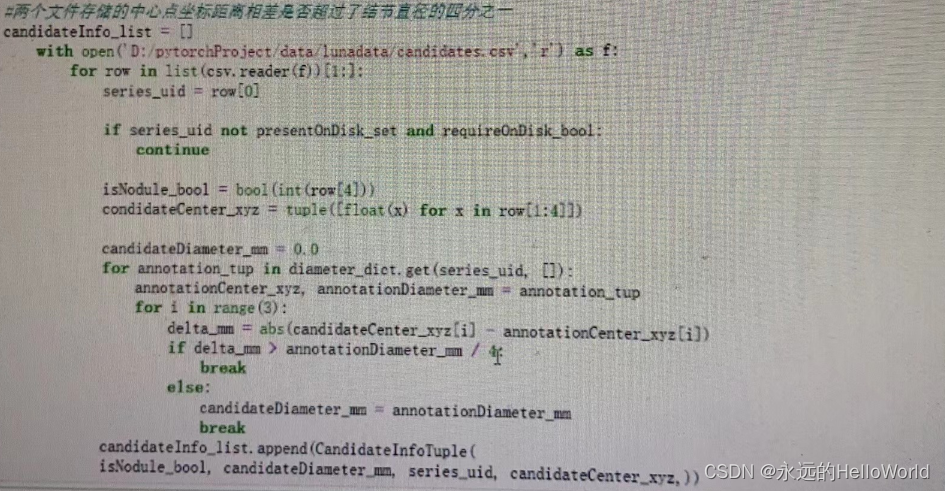
diameter_dict={}
? ? ?with open('/xunlian/annotations.csv' ,‘r’) as f:
? ? ? ? ? ? for? row in list(csv.reader(f)[1:]):
? ? ? ? ? ? ? ? ?series_uid = row[0]
? ? ? ? ? ? ? ? annotationCenter_xyz = tuple([float(x) for x in row[1:4]])
? ? ? ? ? ? ? ? annotationDiameter_mm = float(row[4])
? ? ? ? ? ? ? ? diameter_dict.setdefault(series_uid,[]).append(annotationCenter_xyz ,annotationDiameter_mm )
#两个文件存储的中心点坐标距离相差是否超过了结节直径的四分之一
candidateInfo_list.sort(reverse=True)
return?candidateInfo_list
# 导入SimpleITK
import??SimpleITK as sitk
class Ct:
? ? ? def _init_(self,series_uid):
? ? ? ? ? ? mhd_path = glob.glob('/xunlian/subset*/{}.mhd'.format(series_uid))[0]
? ? ? ? ? ? ct_mhd = sitk.ReadImage(mhd_path)
? ? ? ? ? ? ct_a = np.array(sitk.GetArrayFromImage(ct_mhd),dtype=np.float32
? ? ? ? ? ? ct_a.clip(-1000,1000,ct_a)
? ? ? ? ? ? self.series_uid = series_uid
? ? ? ? ? ? self.hu_a = ct_a? ?# HU
? ? ? ? ? ? self.origin_xyz = XyzTuple(*ct_mhd.GetOrigin())
? ? ? ? ? ? self.vxSize_xyz = XyzTuple(*ct_mhd.GetSpacing())
? ? ? ? ? ? self.direction_a = np.array(ct_mhd.GetDirection().reshape(3,3))
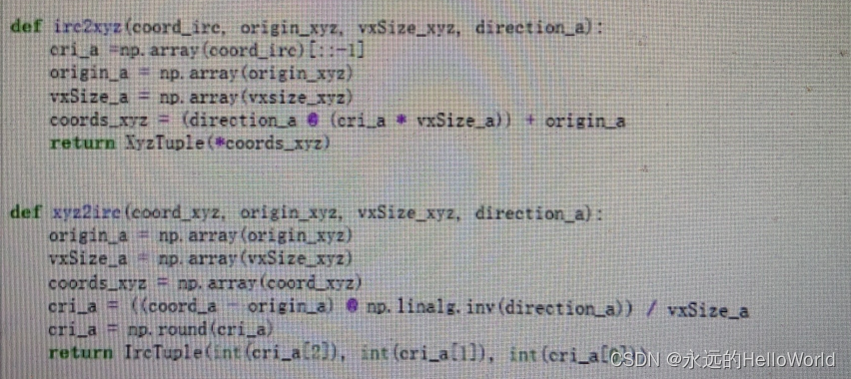
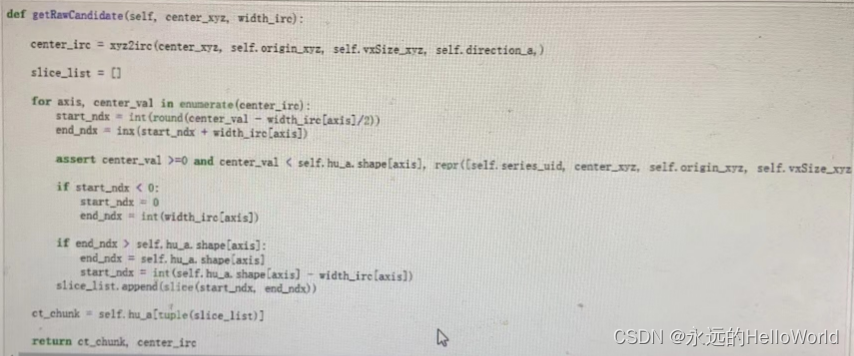
# 毫米为单位的坐标称为 (X,Y,Z)坐标,以体素为单位的坐标称为(I,R,C)
数据坐标系的转化的代码实现
IrcTuple = collections.namedtuple('IrcTuple',['index','row','col'])
XyzTuple? =?collections.namedtuple('XyzTuple',['x','y','z'])
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:chenni525@qq.com进行投诉反馈,一经查实,立即删除!
- Python教程
- 深入理解 MySQL 中的 HAVING 关键字和聚合函数
- Qt之QChar编码(1)
- MyBatis入门基础篇
- 用Python脚本实现FFmpeg批量转换
- 第一章 FPGA开发环境安装
- node 版本管理(windows/linux)
- 4D打印何时能走出实验室,获得真正的应用
- 【重点问题回顾】攻击面发现揭秘直播
- 【Java面试题】在Java中String,Stringbuffer,StringBuilder的区别?
- 基于电商场景的高并发RocketMQ实战-NameServer内存中核心数据模型分析、内核级Producer发送消息流程
- 05 SpringBoot之跨域
- python dash 的学习笔记1
- ubuntu安装kibana
- 位运算 总结