100个GEO基因表达芯片或转录组数据处理之GSE27342(007)
发布时间:2024年01月19日
写在前边
虽然现在是高通量测序的时代,但是GEO、ArrayExpress等数据库储存并公开大量的基因表达芯片数据,还是会有大量的需求去处理芯片数据,并且建模或验证自己所研究基因的表达情况,芯片数据的处理也可能是大部分刚学生信的道友入门R语言数据处理的第一次实战,因此准备更新100个基因表达芯片或转录组高通量数据的处理。
数据信息检索
可以看到GSE27342是基因表达芯片数据,因此可以使用GEOquery包下载数据
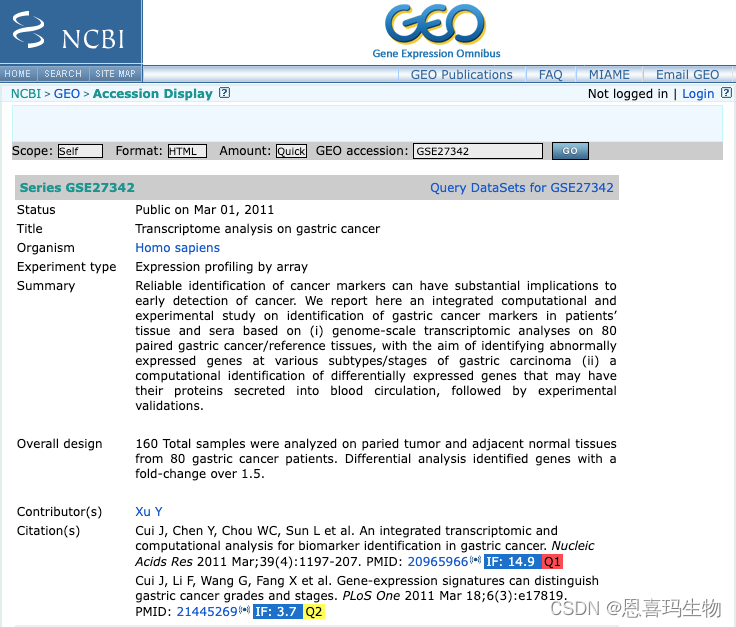
使用GEOquery包下载数据
using(tidyverse, GEOquery, magrittr, data.table, AnnoProbe, clusterProfiler, org.Hs.eg.db, org.Mm.eg.db)
注:using是我写的函数,作用是一次性加载多个R包,不用写双引号,并且不在屏幕上打印包的加载信息,可以参考之前的推文《如何优雅地管理R包》using的定义;函数名字using是在模仿Julia语言中的包加载函数
因为文件太大,在R内下载失败,可通过图片中的方法下载文件,并且把下载后的文件放在destdir = "./"即当前工作目录,GEOquery::getGEO便能跳过下载,直接使用本地的文件。
geo_accession <- "GSE27342"
gset <- GEOquery::getGEO(geo_accession, destdir = "./", AnnotGPL = F, getGPL = F)
eSet <- gset[[1]]
gpl <- eSet@annotation
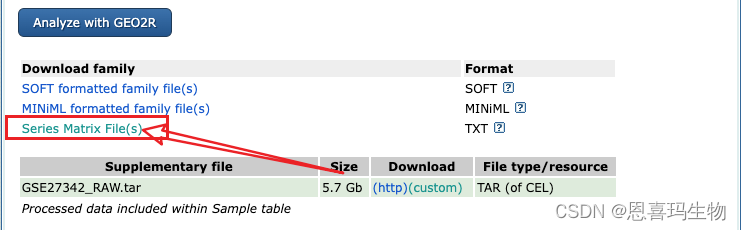

处理表型数据
这部分是很关键的,可以筛选一下分组表型信息,只保留自己需要的样本,在这里只保留tissue:ch1中 normal gastric tissue和gastric cancer tissue的样本,作为后续分析的样本(根据自己的研究目的筛选符合要求的样本)
pdata <- pData(eSet)
| geo_accession | age:ch1 | gender:ch1 | grade:ch1 | Stage:ch1 | tissue:ch1 |
|---|---|---|---|---|---|
| GSM675890 | 41 | F | unknown | IV | normal gastric tissue |
| GSM675891 | 41 | F | unknown | IV | gastric cancer tissue |
| GSM675892 | 62 | F | unknown | III | normal gastric tissue |
| GSM675893 | 62 | F | unknown | III | gastric cancer tissue |
| GSM675894 | 54 | F | G2 | III | normal gastric tissue |
| GSM675895 | 54 | F | G2 | III | gastric cancer tissue |
pdata %<>%
dplyr::mutate(
Sample = geo_accession,
Group = dplyr::case_when(
stringr::str_detect(`tissue:ch1`, "normal") ~ "Control",
stringr::str_detect(`tissue:ch1`, "cancer") ~ "Cancer",
TRUE ~ NA)
) %>%
dplyr::filter(!is.na(Group)) %>%
dplyr::rename(Age=`age:ch1`,Gender=`gender:ch1`,Grade=`grade:ch1`,Stage=`Stage:ch1`) %>%
dplyr::select(Sample, Group, Gender, Grade, Stage)
处理表达谱数据
数据大小大于50需要取log
exprs_mtx <- exprs(eSet)
range(exprs_mtx, na.rm = TRUE)
# 5e-05 1957353.939
exprs_mtx <- log2(exprs_mtx+1)
probe_exprs <- as.data.table(exprs_mtx, keep.rownames = "ProbeID")
探针与基因Symbol对应关系
下载GPL18990注释信息https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GPL5175,需要ID和Gene Symbol列
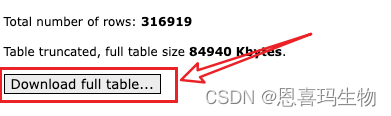
从GEO网站的平台注释文件中获取探针与GeneID对应关系
idmaps2 <- function(ann_file, ProbeID = "ID", GeneID = "gene_assignment", skip = 12, pattern = "control") {
temp <- fread(ann_file, skip = skip, nThread = 8)
vars <- c(ProbeID, GeneID)
temp <- temp[, ..vars]
data.table::setnames(temp, c("ProbeID", "GeneID"))
genes <- stringr::str_split(temp$GeneID," // ",simplify = TRUE)[,2]
temp %<>% dplyr::mutate(GeneID=stringr::str_split(string=GeneID,pattern = " // ",simplify = TRUE)[,2] %>%
stringr::str_remove_all(" ")) %>%
data.table::as.data.table()
temp <- temp[!is.null(GeneID), ][!is.na(GeneID), ][GeneID != "", ][GeneID != "---", ][!stringr::str_detect(string = GeneID, pattern = pattern), ]
return(as.data.frame(temp))
}
probe2symbol <- idmaps2("GPL5175-3188.txt", GeneID = "gene_assignment", skip = 12)
ID转换
把表达矩阵中的探针名转换为基因名;transid是我写的一个R函数,有需要可以联系我的公众号@恩喜玛生物,加入交流群
fdata <- transid(probe2symbol, probe_exprs)
保存数据
common_samples <- base::intersect(colnames(fdata),pdata$Sample)
fdata %<>% select(all_of(c("GeneID",common_samples)))
fwrite(fdata, file = stringr::str_glue("{geo_accession}_{gpl}_fdata.csv.gz"))
pdata %<>% dplyr::filter(Sample %in% common_samples)
fwrite(pdata, file = stringr::str_glue("{geo_accession}_{gpl}_pdata.csv"))

文章来源:https://blog.csdn.net/weixin_44493991/article/details/135692821
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:chenni525@qq.com进行投诉反馈,一经查实,立即删除!
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:chenni525@qq.com进行投诉反馈,一经查实,立即删除!
最新文章
- Python教程
- 深入理解 MySQL 中的 HAVING 关键字和聚合函数
- Qt之QChar编码(1)
- MyBatis入门基础篇
- 用Python脚本实现FFmpeg批量转换
- P33鱼和熊掌可以兼得的机器学习-why deep
- java.lang.Exception: Method a() should be public
- 共话 AI for Science | 中国自然资源航空物探遥感中心于峻川:“AI+遥感”技术地学应用实践与展望
- 德思特应用 | 革新MIMO无线电测试,精准测量10 MHz-8 GHz复杂射频信号!(二)
- 【Spring实战】26 使用Spring Security 保护 Spring Boot Admin
- linux 设备驱动之tty 线路设置
- 强化学习的数学原理学习笔记 - 值函数近似(Value Function Approximation)
- 计算机毕业设计----SSH高校科研管理系统平台
- 最全程序员职业发展路线,这13个工作岗位越早看到越好!
- Repo代码仓库搭建