多标签节点分类

Multi-Label Node Classification on Graph-Structured Data,TMLR’23
Code
学习笔记
图结构数据的多标签分类
-
节点表示或嵌入方法
通常会生成查找表,以便将相似的节点嵌入的更近。学习到的表示用作各种下游预测模块的输入特征。
表现突出的方法是基于随机游走(random walk)的方法:通过节点在随机游走中的共现频率(co-occurrence frequency)来定义节点之间的相似性。[在随机游走的过程中,如果两个节点经常在一起出现,即它们在游走序列中共现的频率较高。]比如DeepWalk -
CNN.其他方法比如说使用卷积神经网络,首先通过聚合局部邻域的特征信息来提取节点表示。然后将提取的特征向量与标签嵌入融合以生成最终的节点嵌入。比如LANC
-
图神经网络(GNNs)
通过递归聚合和转换其邻居的特征表示来计算节点表示,然后将其传递到分类模块。图卷积运算的第k层可以描述为:
z i ( k ) = A G G R E G A T E ( { x i ( k ? 1 ) , { x j ( k ? 1 ) ∣ j ∈ N ( i ) } } ) z_i^{(k)}=AGGREGATE(\left \{ \mathbf{x}_i^{(k-1)},\left \{ {\mathbf{x}_j^{(k-1)}|j\in N(i)} \right \}\right \}) zi(k)?=AGGREGATE({xi(k?1)?,{xj(k?1)?∣j∈N(i)}})
对于多标签节点分类,采用sigmoid层作为最后一层来预测类别概率: y ← ( sigmoid ( z i ( L ) θ ) ) \mathbf{y}\gets (\text{sigmoid}(z_i^{(L)}\theta )) y←(sigmoid(zi(L)?θ)) θ \theta θ对应于分类模块中的可学习权重矩阵。
GNN模型的主要区别在于聚合层的实现。最简单的模型是GCN:对邻域特征采用度加权(degree-weighted)聚合;
GAT采用了多个堆叠的图注意力层,它允许节点关注其邻域的特征;
GraphSAGE仅使用邻域的随机样本进行特征聚合步骤。
一般来说,GNNs在高**同配图(连接的节点往往共享相同的标签)**表现出更好的性能。H2GCN显示了在异配图上(多类设置)的改进:将邻居聚合的信息与自我节点的信息分开。此外,它利用高阶邻域信息来学习信息丰富的节点表示。 -
标签传播(label propagation)
LPA算法和GNNs都基于消息传递。GNNs传播和变换节点特征,而LPA沿着图的边缘传播节点标签信息,以预测未标记节点的标签分布。 最近的一些工作将他们结合起来,比如GCN-LPA利用LPA作为正则项来帮助GCN学习适当的边权重,从而提高分类性能。
多标签数据集的特性
标签同配性label homophily
本文提出多标签图数据集的同配性定义
GNNs的性能通常根据标签同配性来讨论,标签同配性量化了图中相似节点之间的相似性,特别是,标签同配性在文(Beyond homophily in graph neural networks: Current limitations and effective designs)中被定义为图中同配边的分数:其中如果一条边连接具有相同标签的两个节点,则该边被认为是同配的。
这一定义不能直接用于多标签图数据集,因为每个节点可以有多个标签,并且在多标签dataset中两个连接节点的整个标签集相同的情况很少见。通常,两个节点共享其部分标签。
给定一个多标签图
G
G
G,其同配性
h
h
h定义为图中所有连通节点的标签集的杰卡德(Jaccard)相似度的平均值:
h
=
1
∣
ε
∣
∑
(
i
,
j
)
∈
ε
∣
l
(
i
)
∩
l
(
j
)
∣
∣
l
(
i
)
∪
l
(
j
)
∣
.
h=\frac{1}{|\varepsilon |}\sum _{(i,j)\in \varepsilon }\frac{|l(i)\cap l(j)|}{|l(i)\cup l(j)|} .
h=∣ε∣1?(i,j)∈ε∑?∣l(i)∪l(j)∣∣l(i)∩l(j)∣?.
标签同配性是一阶标签引起的相似性,因为它根据相邻节点的标签分布来量化相邻节点之间的相似性。
跨类邻域相似度CCNS
二阶标签诱导度量,量化任何两个节点的邻域之间的相似性。

异配图的CCNS较低,同配图的CCNS更高
常用多标签节点分类数据集
- BlogCat
nodes博客作者,edges表示他们的关系,labels表示所属社会群体 - Yelp
nodes客户评论,edges对应他们的friendship,labels代表企业类型 - OGB-Proteins
nodes蛋白质,edges表示蛋白质之间不同类型的生物学意义关联,labels对应于蛋白质功能 - DBLP
nodes作者,edges合著关系,labels表示作者的研究领域
‘25%’、'50%'和’75%'对应于节点的标签数量的排序列表的第25、第50和第75位。即,有 25%、'50%‘或’75%’ 的节点的标签数目小于或等于这个值。
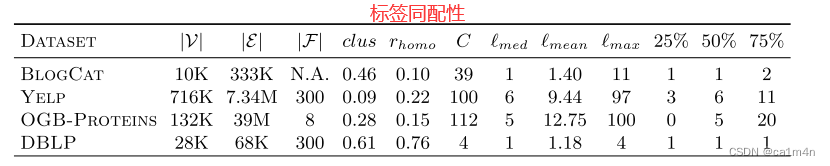
- 标签分布不对称
- 高标签稀疏性下使用AUROC评分进行评估存在问题
(ROC曲线是以召回率为纵轴,FPR为横轴的曲线,其面积代表AUROC分数
FPR是被错误地预测为正类别地负样本占所有负样本的比例,召回率是预测为true positive样本占所有实际positive样本的比例)
因此存在,通过增加训练使其的数量来增加AUROC分数,即鼓励了模型通过预测为negative样本来减少损失。
NEW生物数据集
- PCG:蛋白质表型预测(phenotype:疾病可观察到的特征或形状)
- HumLoc:人类蛋白质亚细胞位置预测数据集(预测蛋白质亚细胞位置可以帮助识别药物靶点)标签是位置信息,14个维度one-hot编码
- EukLoc:真核生物蛋白质亚细胞位置预测数据集
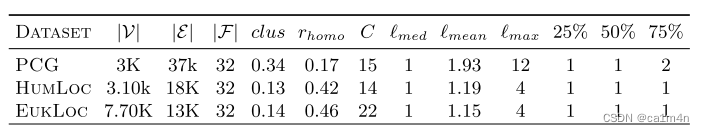
HUMLOC、EUKLOC有着较高的同配性
Results
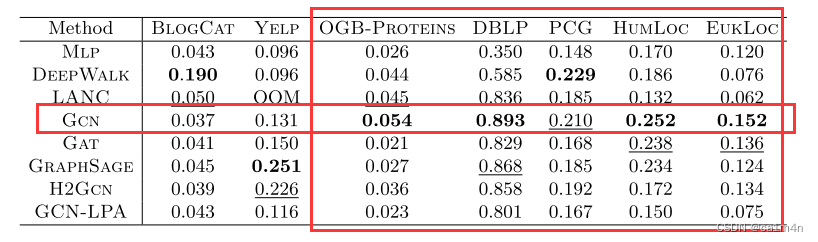
- DBLP
- EukLoc
- HumLoc
- PCG
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:chenni525@qq.com进行投诉反馈,一经查实,立即删除!