喜讯!ChIP-seq再立功,组蛋白去甲基化酶在水稻种子的表观调控机制见刊The Plant Journal

-
发表单位:广东省农业科学院农业生物基因研究中心
-
发表日期:2023年12月20日
-
期刊:The Plant Journal
-
影响因子:7.2
2023年12月20日,广东省农业科学院农业生物基因研究中心种子表观遗传刘军研究团队在The Plant Journal上发表题为“OsJMJ718, a histone demethylase gene, positively regulates seed germination in rice”的研究论文。研究揭示了组蛋白去甲基化酶OsJMJ718介导靶基因上的H3K9me3去甲基化影响ABA和乙烯信号转导途径调节水稻种子萌发。爱基百客为该研究提供了ChIP-seq的技术支持。

01 研究背景
水稻是重要的粮食作物,对粮食安全至关重要。种子活力对水稻出苗率、出苗均匀性、幼苗生长、作物产量和播种后的质量有显著影响。研究种子活力的调节机制对解决种子活力遗传改良、促进水稻直接种植生产等实际问题具有重要的理论和实践意义。表观遗传学对于染色质结构和动态基因表达的调节十分重要,一些表观遗传基因被证明可以调节种子萌发和抗逆性。组蛋白甲基化是一种重要的表观遗传修饰机制,对植物生长、开花、果实成熟和胁迫反应至关重要。在水稻种子萌发中,是否存在组蛋白甲基化的调节以及相关的表观遗传因子尚不是十分清楚。
02 研究思路

03 研究结果
1、OsJMJ718基因调控水稻种子萌发
JmjC家族蛋白是一类重要的组蛋白赖氨酸去甲基化酶,在调控基因表达和水稻发育中具有一定的功能。此研究利用RT-qPCR检测种子萌发过程中的JmjC基因表达水平,发现OsJMJ718可能参与了种子萌发。OsJMJ718序列为6468bp,包含7个外显子和6个内含子(图1a),其ORF长1140bp,编码一个380aa的预测蛋白。进化分析表明,OsJMJ718与拟南芥中的AtJMJ25、AtJMJ26、AtJMJ29和AtJMJ27蛋白序列一致。已知AtJMJ25和AtJMJ27调控拟南芥发育和非生物胁迫应答,推测OsJMJ718也有类似的功能。为了研究OsJMJ718的功能,作者构建了敲除突变体和过表达(OE)系。
基于CRISPR-Cas9和35S启动子控制的过表达技术,获得了4个OsJMJ718敲除系(CR)(t图1b)和10个OE系(图1c),2个CR系(CR-18和CR-25)和2个OE系(OE-4和OE-8)被选择进行表型分析(图1c,d)。从三种基因型材料上采集的种子在保存2个月后,CR系表现出种子活力较低的情况,萌发势和幼苗活力指数较低(图1e-h),这说明OsJMJ718正调节种子萌发。

图1. 野生型(WT)、OsJMJ718敲除系(OsJMJ718-CR)和过表达系(OsJMJ718-OE)的萌发分析
2、OsJMJ718的表达模式及亚细胞定位
采用qRT-PCR分析了OsJMJ718基因在根、茎、叶、幼穗、旗叶及开花后3、6、9、12、15、18和21 d的发育种子中的表达情况。OsJMJ718在叶片、旗叶和开花后9和21d的发育种子中表达量较高,但在根和茎中表达量较低(图2a,c)。此外,以吸胀0、12、24和48h的萌发种子进行分析,发现OsJMJ718的转录水平在种子萌发期间趋向于下调(图2b)。在各种非生物胁迫处理下,ABA和盐处理3h能显著诱导OsJMJ718高表达,低温和PEG处理也有一定的诱导作用,而ACC处理未能诱导OsJMJ718表达(图2d)。OsJMJ718启动子GUS (pOsJMJ718:GUS)转基因系显示OsJMJ718在胚胎和胚囊内均有表达(图2e)。在水稻原生质体和烟草叶片中,激光共聚焦显微镜显示OsJMJ718-GFP的绿色荧光信号主要分布在细胞核内(图2f、g),这说明OsJMJ718可能是细胞核定位蛋白。

图2. OsJMJ718的表达模式分析及其在水稻原生质体和烟草表皮细胞中的亚细胞定位
3、转基因材料的组蛋白修饰分析
为了研究OsJMJ718在组蛋白修饰中的功能,从各系材料的幼苗中提取组蛋白进行Western blot分析。与WT相比,CR系中的H3K9me1和H3K9me3水平升高。然而,OsJMJ718过表达导致H3K9me1和H3K9me3水平降低。这表明OsJMJ718可能是一种组蛋白H3K9me 1/3去甲基化酶(图3)。

图3. OsJMJ718是H3K9me1/3去甲基化酶
4、OsJMJ718活性的功能丧失导致数千个基因的H3K9me3增加
作者进一步研究OsJMJ718是否通过靶基因的H3K9me3水平调节其表达,为此进行了ChIP-seq,以分析WT和OsJMJ718-CR萌发种子中的全基因组H3K9me3谱。OsJMJ718介导的H3K9me3去除位点分布在被注释基因的不同区域,包括外显子、启动子、内含子、3’UTR、5’UTR和基因间区域,其中基因间区域占很大比例(67.96%,图4a)。在OsJMJ718-CR中,与WT相比,观察到8235个H3K9me3超甲基化峰(FDR <0.05),对应5867个基因。相比之下,H3K9me3低甲基化峰有151个,对应129个基因(图4b)。
通过整合ChIP-seq和RNA-seq的分析,在OsJMJ718-CR系中分别获得了因H3K9me3水平升高或降低造成表达变化的1217个基因和19个基因。KEGG和GO富集分析显示,这1236个基因与植物激素信号途径密切相关(图4c)。3个ABA信号转导途径基因(OsPP2C30,OsWRKY24和OsEMP1)、3个GA相关基因(OsWRKY53,OsGA3和OsGASR9)和2个乙烯相关基因(OsERF34和OsERF48)的可视化图进一步确认了H3K9me3水平的变化(图4d)。基于RNA-seq数据,这些选定基因中的大部分在OsJMJ718-CR中都表达降低了(图4e)。这些结果说明OsJMJ718-CR中H3K9me3水平的升高与基因表达的降低呈正相关。

图4. 抗H3K9me3的ChIP-seq和RNA-seq综合分析
5、OsJMJ718调节ABA和ACC水平
为了研究与OsJMJ718相关的激素,分别采用高效液相色谱-串联质谱(LC-MS /MS)和气相色谱-串联质谱法(GC-MS/MS)技术测定了萌发0、12、24、48和72 h的三种基因型水稻种子的ABA和ACC含量。萌发后0 ~ 48 h,OsJMJ718-CR的ABA含量高于WT,而OsJMJ718-OE则相反(图5a)。由于ABA的生物合成和分解代谢决定了细胞内ABA的水平,作者确定了OsJMJ718-CR和OsJMJ718-OE中ABA合成和分解代谢基因的表达是否受到影响。ABA分解代谢基因OsABA8ox1在OsJMJ718-OE中升高,在OsJMJ718-CR中降低。有趣的是,在吸胀后12-72 h,?OsJMJ718-CR中的ACC含量低于WT(图5b)。研究确定了OsJMJ718-OE和OsJMJ718-CR中乙烯应答基因的表达是否发生了变化。与WT相比,OsJMJ718-CR中许多乙烯应答转录因子基因,如OsERF3、OsERF104、OsEATB和OsERF109,在OsJMJ718-CR中显著减少。
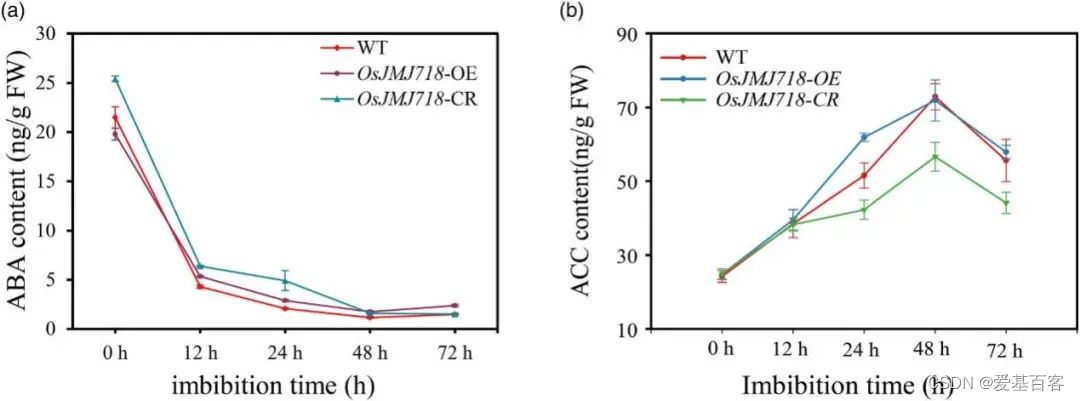
图5. OsJMJ718能改变水稻萌发种子中的ABA和ACC的含量
6、OsJMJ718通过调控ABA和乙烯信号影响水稻种子萌发
RNA-seq、ChIP-seq和表达模式分析表明,OsJMJ718可能参与水稻内ABA和乙烯信号转导。为了证实这一点,研究在萌发阶段评估了WT和转基因系对ABA的敏感性。在对照条件下,OE系在72 h发芽率达到57-70%,CR系则是28-30%,而WT的发芽率为58%,说明OsJMJ718正调控萌发(图6b)。然而,在ABA处理下,所有种子都出现了不可避免的发芽延迟(图6a)。与WT种子相比,OE系种子对ABA浓度的增加更为敏感,这表明OsJMJ718可能对ABA信号转导组分具有负调控作用(图6b-d)。为了确定转基因植物对ABA的生长敏感性,作者将在对照条件下发芽的种子转移到含有不同浓度 ABA的1/2 MS液体培养基中,种子经过2天的吸胀,在3周后测量幼苗高度。在3和5 μmol/L ABA处理下,与WT相比,OE植株对ABA的敏感性增加,生长速率显著降低(图6e,f)。此外,3个ABA应答基因,OsbZIP72在转基因系和WT中在有无ABA处理的情况下表达水平没有显著变化,而OsPP2C30和OsLEA3在ABA处理下,与WT相比,在OE系中显著上调。
此外,为了确定OsJMJ718在萌发阶段是否与乙烯信号转导有关,作者分析了WT、OE系和CR系在外源乙烯存在下的种子萌发情况。当种子萌发时添加30μM ACC或10μM乙烯利时,CR系的促苗率高于WT,而OE系的促苗率与WT无显著差异。

图6. OsJMJ718正调节种子萌发和萌发后生长过程中对ABA的敏感性
04 结? ?论?
总之,这项研究结果表明OsJMJ718在调节种子萌发和活力方面发挥积极作用。OsJMJ718敲除系的低发芽率和种子活力与种子萌发过程中游离ABA的水平升高、ACC水平降低有关。OsJMJ718可以通过去除OsPP2C和OsERF中H3K9me3的甲基化激活它们的表达,分别抑制ABA信号通路转导和促进乙烯信号转导,从而促进种子萌发(图7)。

图7. OsJMJ718调控水稻种子萌发的潜在作用模型
关于我们
爱基百客提供专业的表观遗传技术服务检测,在ChIP-seq上有着丰富经验,协助客户以组蛋白修饰酶为切入点的文章发表在New Phytologist、Plant Communications、Plant Physiology、Plant Biotechnology Journal、PLos Genetics、Virulence等刊物上。
-
组蛋白修饰酶相关的部分项目文章
-
DING, Xiaochun, et al. SlJMJ7 orchestrates tomato fruit ripening via crosstalk between H3K4me3 and DML2‐mediated DNA demethylation. New Phytologist, 2022, 233.3: 1202-1219.
-
CHEN, Xiaoyang, et al. A secreted fungal effector suppresses rice immunity through host histone hypoacetylation. New Phytologist, 2022, 235.5: 1977-1994.
-
ZHENG, Yangyi, et al. Rice OsUBR7 modulates plant height by regulating histone H2B monoubiquitination and cell proliferation. Plant Communications, 2022, 3.6.
-
CHEN, Xiaoyang, et al. Ustilaginoidea virens‐secreted effector Uv1809 suppresses rice immunity by enhancing OsSRT 2‐mediated histone deacetylation. Plant Biotechnology Journal, 2023.
-
JIANG, Hang, et al. Opposing functions of Fng1 and the Rpd3 HDAC complex in H4 acetylation in Fusarium graminearum. PLoS Genetics, 2020, 16.11: e1009185.
-
ZHUANG, Zhenhong, et al. Set2 family regulates mycotoxin metabolism and virulence via H3K36 methylation in pathogenic fungus Aspergillus flavus. Virulence, 2022, 13.1: 1358-1378.
-
XU, Huaijian, et al. The Fng3 ING protein regulates H3 acetylation and H4 deacetylation by interacting with two distinct histone‐modifying complexes. New Phytologist, 2022, 235.6: 2350-2364.
-
LU, Jingqin, et al. SET DOMAIN GROUP 711-mediated H3K27me3 methylation of cytokinin metabolism genes regulates organ size in rice. Plant Physiology, 2023, kiad568.
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:chenni525@qq.com进行投诉反馈,一经查实,立即删除!