工具系列:TensorFlow决策森林_(3)使用dtreeviz可视化
文章目录
介绍
之前的教程演示了如何使用TensorFlow的决策森林(随机森林、梯度提升树和CART)分类器和回归器来准备数据、训练和评估。 (我们将TensorFlow决策森林缩写为TF-DF。)您还学会了如何使用内置的plot_model_in_colab()函数可视化树,并显示特征重要性度量。
本教程的目标是通过可视化更深入地解释分类器和回归器决策树。我们将查看详细的树结构图示,以及决策树如何划分特征空间以做出决策的描绘。树结构图帮助我们理解模型的行为,特征空间图帮助我们通过展示特征和目标变量之间的关系来理解数据。
我们将使用的可视化库称为dtreeviz,为了保持一致性,我们将重复使用初学者教程中的企鹅和鲍鱼数据
在本教程中,您将学习如何:
- 显示TF-DF森林中决策树的结构
- 更改dtreeviz树结构图的大小和样式
- 绘制叶子信息,例如每个叶子中的实例数、每个叶子中目标值的分布以及关于叶子的各种统计信息
- 跟踪树对特定实例的解释,并显示从根到叶子的路径,以进行预测
- 打印树如何解释实例的英文解释
- 查看一维和二维特征空间,以了解模型如何将它们划分为相似实例的区域
设置
安装 TF-DF 和 dtreeviz
# 安装tensorflow_decision_forests库
!pip install -q -U tensorflow_decision_forests
# 安装 dtreeviz 库
!pip install -q -U dtreeviz
导入库
import tensorflow_decision_forests as tfdf
import tensorflow as tf
import os
import numpy as np
import pandas as pd
import tensorflow as tf
import math
import dtreeviz
from matplotlib import pyplot as plt
from IPython import display
# 避免“Arial字体未找到”的警告
import logging
logging.getLogger('matplotlib.font_manager').setLevel(level=logging.CRITICAL)
display.set_matplotlib_formats('retina') # 生成高分辨率的图形
np.random.seed(1234) # 为了可重现的图形/数据解释的目的
2023-03-07 12:10:56.998585: W tensorflow/compiler/xla/stream_executor/platform/default/dso_loader.cc:64] Could not load dynamic library 'libnvinfer.so.7'; dlerror: libnvinfer.so.7: cannot open shared object file: No such file or directory
2023-03-07 12:10:56.998704: W tensorflow/compiler/xla/stream_executor/platform/default/dso_loader.cc:64] Could not load dynamic library 'libnvinfer_plugin.so.7'; dlerror: libnvinfer_plugin.so.7: cannot open shared object file: No such file or directory
2023-03-07 12:10:56.998714: W tensorflow/compiler/tf2tensorrt/utils/py_utils.cc:38] TF-TRT Warning: Cannot dlopen some TensorRT libraries. If you would like to use Nvidia GPU with TensorRT, please make sure the missing libraries mentioned above are installed properly.
/tmpfs/tmp/ipykernel_9236/31193553.py:20: DeprecationWarning: `set_matplotlib_formats` is deprecated since IPython 7.23, directly use `matplotlib_inline.backend_inline.set_matplotlib_formats()`
# 打印库的版本信息
tfdf.__version__, dtreeviz.__version__ # 希望 dtreeviz 的版本大于等于 2.2.0
('1.2.0', '2.2.0')
为了方便起见,我们需要定义一个函数来将数据集分为训练集和测试集:
# 定义一个函数split_dataset,用于将一个panda dataframe分成两部分,通常用于训练集和测试集的划分。
# 使用相同的随机种子确保我们得到相同的划分,以便本教程中的描述与生成的图像相对应。
def split_dataset(dataset, test_ratio=0.30, seed=1234):
"""
将一个panda dataframe分成两部分,通常用于训练集和测试集的划分。
使用相同的随机种子确保我们得到相同的划分,以便本教程中的描述与生成的图像相对应。
参数:
dataset:要划分的数据集,panda dataframe类型
test_ratio:测试集所占比例,默认为0.30
seed:随机种子,默认为1234
返回值:
划分后的训练集和测试集,均为panda dataframe类型
"""
# 设置随机种子
np.random.seed(seed)
# 生成一个与dataset长度相同的随机数数组,元素值在0到1之间
# 若随机数小于test_ratio,则对应位置为True,否则为False
test_indices = np.random.rand(len(dataset)) < test_ratio
# 返回划分后的训练集和测试集
# 通过~test_indices可以得到test_indices的逻辑反,即对应位置为False的元素
# 通过test_indices可以得到test_indices的逻辑值,即对应位置为True的元素
return dataset[~test_indices], dataset[test_indices]
可视化分类树
 使用企鹅数据,让我们构建一个分类器来预测其他7列中的
使用企鹅数据,让我们构建一个分类器来预测其他7列中的species(Adelie,Gentoo或Chinstrap)。然后,我们可以使用dtreeviz来显示树并询问模型以了解它如何做出决策以及了解我们的数据。
加载、清洗和准备数据
和初学者教程一样,让我们开始下载企鹅数据并将其转换为pandas数据框。
# 下载企鹅数据集
!wget -q https://storage.googleapis.com/download.tensorflow.org/data/palmer_penguins/penguins.csv -O /tmp/penguins.csv
# 将数据集加载到 Pandas Dataframe 中
df_penguins = pd.read_csv("/tmp/penguins.csv")
# 显示前三行数据
df_penguins.head(3)
| species | island | bill_length_mm | bill_depth_mm | flipper_length_mm | body_mass_g | sex | year | |
|---|---|---|---|---|---|---|---|---|
| 0 | Adelie | Torgersen | 39.1 | 18.7 | 181.0 | 3750.0 | male | 2007 |
| 1 | Adelie | Torgersen | 39.5 | 17.4 | 186.0 | 3800.0 | female | 2007 |
| 2 | Adelie | Torgersen | 40.3 | 18.0 | 195.0 | 3250.0 | female | 2007 |
快速检查显示数据集中存在缺失值:
df_penguins.columns[df_penguins.isna().any()].tolist()
['bill_length_mm', 'bill_depth_mm', 'flipper_length_mm', 'body_mass_g', 'sex']
相比于填充缺失值,让我们只是删除不完整的行,以便在本教程中专注于可视化。
# 删除包含缺失值的行
df_penguins = df_penguins.dropna() # 例如,有19行缺少性别等信息...
TF-DF要求分类标签为整数,范围在[0,num_labels)之间,因此让我们将标签列species从字符串转换为整数。
注意: TF-DF支持分类字符串输入特征。您不需要对任何特征值进行编码。
# 定义变量penguin_label,表示分类目标标签的名称
penguin_label = "species"
# 获取数据集中penguin_label列的所有唯一值,并将其转换为列表
classes = list(df_penguins[penguin_label].unique())
# 将数据集中的penguin_label列的值映射为它们在classes列表中的索引值
df_penguins[penguin_label] = df_penguins[penguin_label].map(classes.index)
# 打印输出分类目标标签的名称和对应的类别列表
print(f"Target '{penguin_label}'' classes: {classes}")
# 显示数据集的前3行
df_penguins.head(3)
Target 'species'' classes: ['Adelie', 'Gentoo', 'Chinstrap']
| species | island | bill_length_mm | bill_depth_mm | flipper_length_mm | body_mass_g | sex | year | |
|---|---|---|---|---|---|---|---|---|
| 0 | 0 | Torgersen | 39.1 | 18.7 | 181.0 | 3750.0 | male | 2007 |
| 1 | 0 | Torgersen | 39.5 | 17.4 | 186.0 | 3800.0 | female | 2007 |
| 2 | 0 | Torgersen | 40.3 | 18.0 | 195.0 | 3250.0 | female | 2007 |
现在,让我们使用上面定义的便捷函数将训练和测试数据按70-30的比例划分,并将这些数据框转换为tensorflow数据集。
分割训练/测试集并训练模型
# 将数据集分割为训练集和测试集
train_ds_pd, test_ds_pd = split_dataset(df_penguins)
print(f"{len(train_ds_pd)} 个训练样本,{len(test_ds_pd)} 个测试样本。")
# 将数据集转换为 TensorFlow 数据集
train_ds = tfdf.keras.pd_dataframe_to_tf_dataset(train_ds_pd, label=penguin_label)
test_ds = tfdf.keras.pd_dataframe_to_tf_dataset(test_ds_pd, label=penguin_label)
243 examples in training, 90 examples for testing.
训练一个随机森林分类器
# 导入所需的库和模块
# 创建一个随机森林模型对象,设置参数verbose为0表示不输出训练过程中的详细信息,random_seed为1234表示设置随机种子为1234
cmodel = tfdf.keras.RandomForestModel(verbose=0, random_seed=1234)
# 使用训练数据集train_ds对模型进行训练
cmodel.fit(train_ds)
WARNING:tensorflow:From /tmpfs/src/tf_docs_env/lib/python3.9/site-packages/tensorflow/python/autograph/pyct/static_analysis/liveness.py:83: Analyzer.lamba_check (from tensorflow.python.autograph.pyct.static_analysis.liveness) is deprecated and will be removed after 2023-09-23.
Instructions for updating:
Lambda fuctions will be no more assumed to be used in the statement where they are used, or at least in the same block. https://github.com/tensorflow/tensorflow/issues/56089
[INFO 2023-03-07T12:11:06.100795433+00:00 kernel.cc:1214] Loading model from path /tmpfs/tmp/tmpeau3pdt_/model/ with prefix 72ee2781602146e9
[INFO 2023-03-07T12:11:06.113257784+00:00 decision_forest.cc:661] Model loaded with 300 root(s), 4310 node(s), and 7 input feature(s).
[INFO 2023-03-07T12:11:06.113286363+00:00 abstract_model.cc:1311] Engine "RandomForestGeneric" built
[INFO 2023-03-07T12:11:06.113305638+00:00 kernel.cc:1046] Use fast generic engine
WARNING:tensorflow:AutoGraph could not transform <function simple_ml_inference_op_with_handle at 0x7f67957524c0> and will run it as-is.
Please report this to the TensorFlow team. When filing the bug, set the verbosity to 10 (on Linux, `export AUTOGRAPH_VERBOSITY=10`) and attach the full output.
Cause: could not get source code
To silence this warning, decorate the function with @tf.autograph.experimental.do_not_convert
WARNING: AutoGraph could not transform <function simple_ml_inference_op_with_handle at 0x7f67957524c0> and will run it as-is.
Please report this to the TensorFlow team. When filing the bug, set the verbosity to 10 (on Linux, `export AUTOGRAPH_VERBOSITY=10`) and attach the full output.
Cause: could not get source code
To silence this warning, decorate the function with @tf.autograph.experimental.do_not_convert
<keras.callbacks.History at 0x7f68310ddd90>
只是为了验证一切是否正常工作,让我们检查模型的准确率,应该约为99%:
# 对模型进行编译,使用"accuracy"作为评估指标
cmodel.compile(metrics=["accuracy"])
# 对测试数据集进行评估,返回字典形式的评估结果,verbose=0表示不输出评估过程
cmodel.evaluate(test_ds, return_dict=True, verbose=0)
{'loss': 0.0, 'accuracy': 0.9888888597488403}
是的,模型在测试集上的准确率很高。
显示决策树
现在我们有了一个模型,让我们选择随机森林中的一棵树,并查看其结构。dtreeviz库要求我们将TF-DF模型与相关的训练数据捆绑在一起,然后可以重复询问模型。
# 获取penguin数据集的特征名
penguin_features = [f.name for f in cmodel.make_inspector().features()]
# 创建一个dtreeviz的可视化模型
# 参数说明:
# - cmodel: 训练好的决策树模型
# - tree_index: 指定要可视化的决策树的索引
# - X_train: 训练集的特征数据
# - y_train: 训练集的标签数据
# - feature_names: 特征的名称列表
# - target_name: 目标变量的名称
# - class_names: 类别的名称列表
viz_cmodel = dtreeviz.model(cmodel,
tree_index=3,
X_train=train_ds_pd[penguin_features],
y_train=train_ds_pd[penguin_label],
feature_names=penguin_features,
target_name=penguin_label,
class_names=classes)
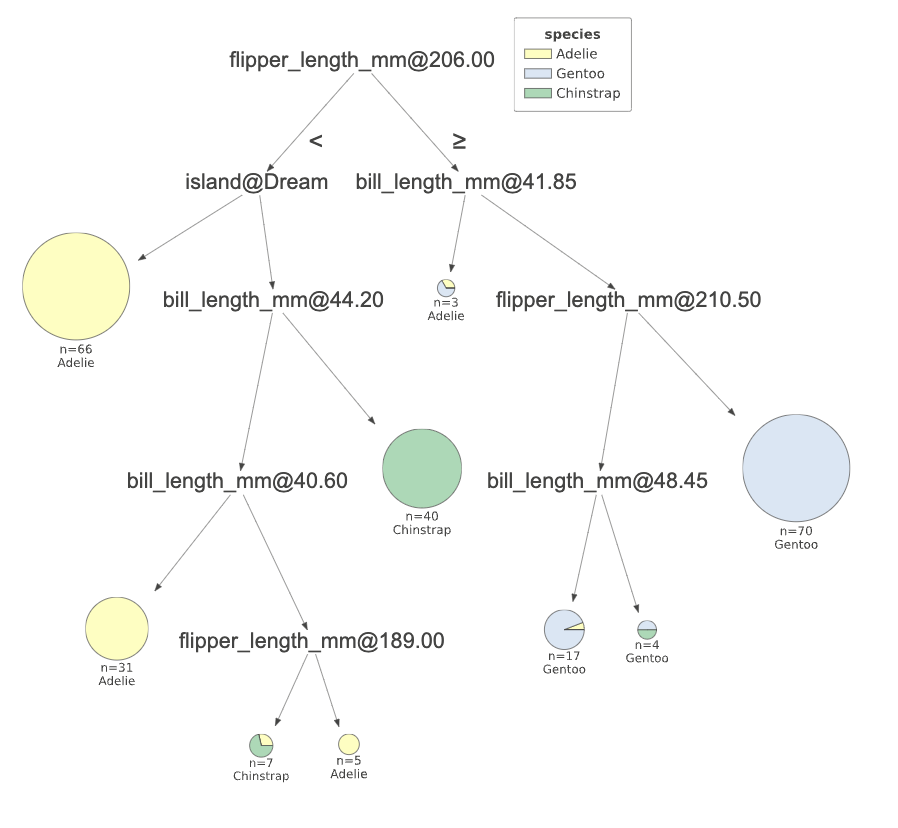
最常见的dtreeviz API函数是view(),它显示树的结构以及与每个决策节点相关联的实例的特征分布。
# 调用viz_cmodel的view方法,并设置缩放比例为1.2,用于显示模型的可视化结果。
viz_cmodel.view(scale=1.2)

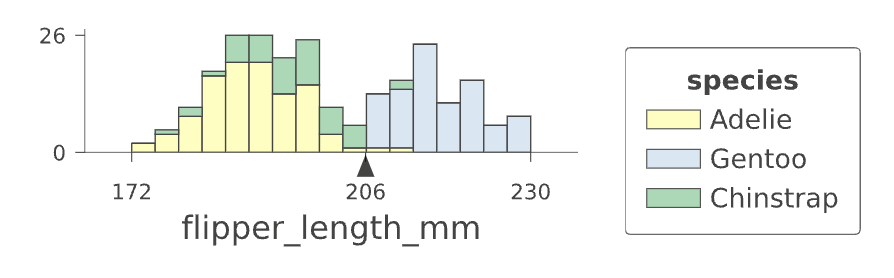
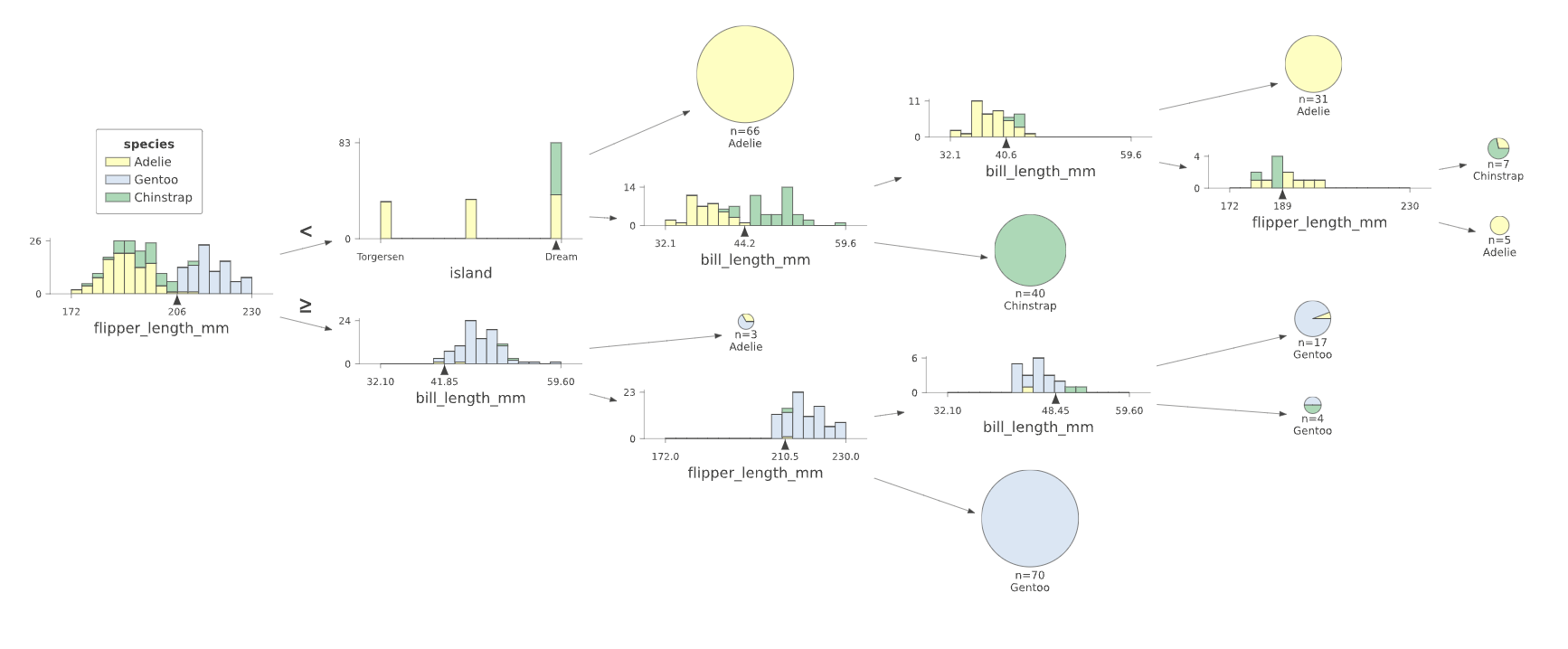
决策树的根节点表示分类开始时通过测试flipper_length_mm特征,使用分割值206。如果测试实例的flipper_length_mm特征值小于206,则决策树向左子节点下降。如果它大于或等于206,则分类通过向右子节点下降进行。
为了了解模型为什么选择在flipper_length_mm=206处分割训练数据,让我们放大根节点。
# 设置深度范围和缩放比例,并显示模型
viz_cmodel.view(depth_range_to_display=[0,0], scale=1.5)
清晰地看到,206右侧的几乎所有实例都是蓝色(Gentoo企鹅)。因此,通过一次特征比较,模型可以将训练数据分成一个相当纯净的Gentoo组和一个混合组。(模型将通过根节点以下的未来分割进一步净化子组。)
决策树还具有分类决策节点,可以测试类别子集而不是简单的数值分割。例如,让我们来看看树的第二层:
# 调用viz_cmodel的view函数,并设置参数
# depth_range_to_display参数用于指定显示的深度范围,这里设置为[1,1],表示只显示深度为1的部分
# scale参数用于指定显示的缩放比例,这里设置为1.5,表示放大1.5倍显示
viz_cmodel.view(depth_range_to_display=[1,1], scale=1.5)
节点(左侧)测试特征island,如果测试实例具有island==Dream,则分类继续向下移动到其右子节点。对于另外两个类别Torgersen和Biscoe,分类继续向下移动到其左子节点。(在这个图中,右侧的bill_length_mm节点与对分类决策节点的讨论无关。)
这种分割行为突出了决策树将特征空间划分为目标值纯度增加的区域的目标。我们将在下面更详细地查看特征空间。
决策树可能会变得非常庞大,将它们完整地绘制出来并不总是有用的。但是,我们可以查看树的简化版本、树的部分、各个叶子节点(进行预测的地方)中的训练实例数量等等… 这是一个例子,我们关闭了精美的决策节点分布图,并将整个图像缩小到75%的比例:
# 调用viz_cmodel的view函数,以可视化模型
# 参数fancy设置为False,表示不使用复杂的样式
# 参数scale设置为0.75,表示缩放比例为0.75
viz_cmodel.view(fancy=False, scale=.75)
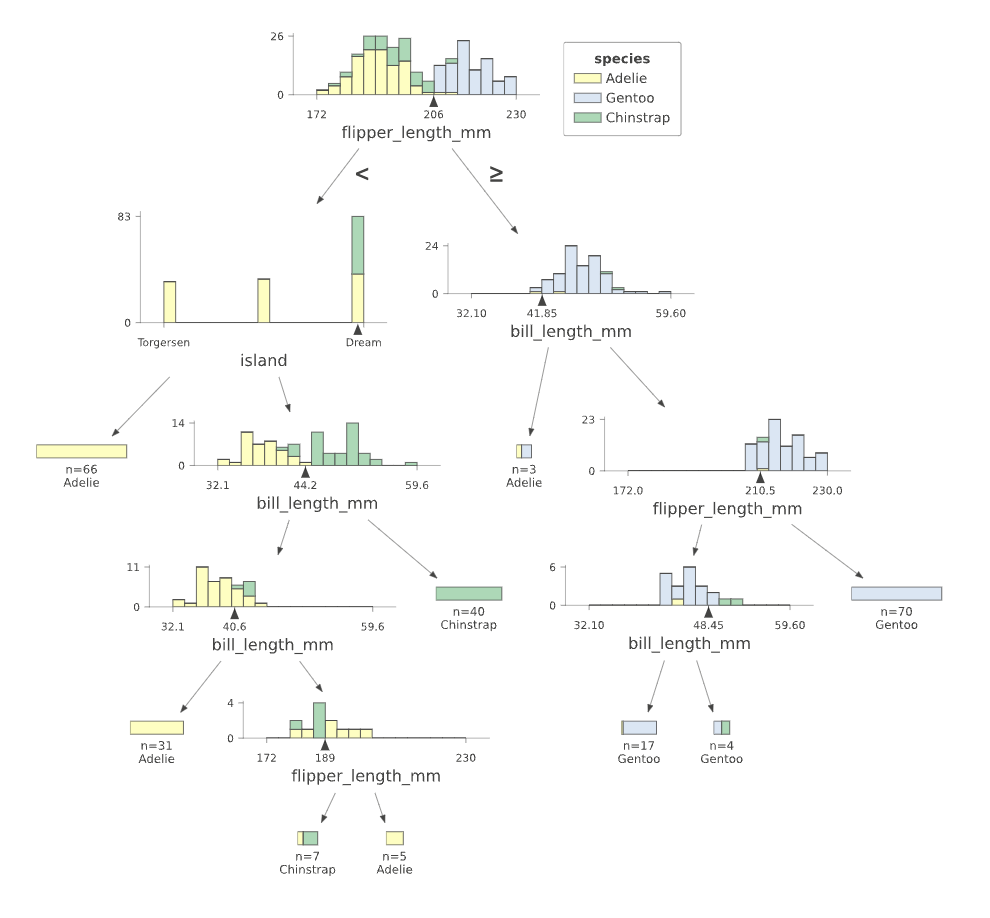
我们还可以使用从左到右的方向,这样有时会得到一个较小的图。
# 设置可视化模型的方向为从左到右,缩放比例为0.75
viz_cmodel.view(orientation='LR', scale=.75)
如果你不是饼图的粉丝,你也可以使用条形图。
# 使用viz_cmodel对象的view方法展示数据可视化结果
# leaftype参数指定使用条形图展示数据
# scale参数指定缩放比例为0.75,即将图形缩小为原来的75%大小
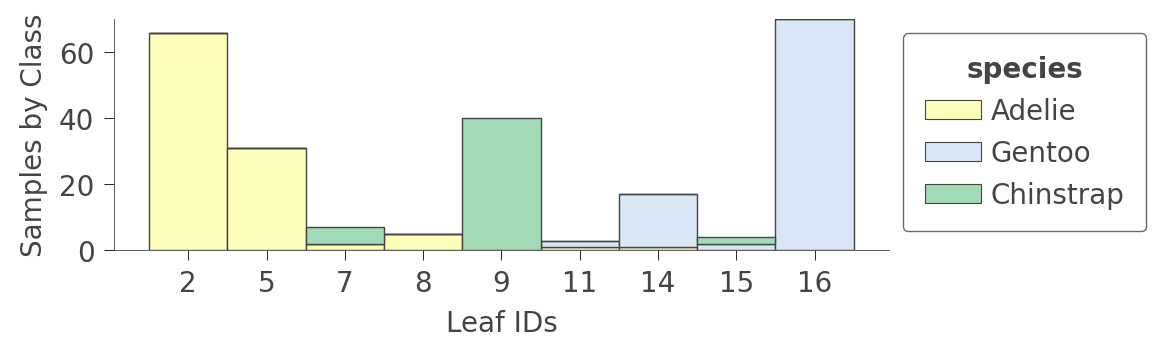
viz_cmodel.view(leaftype='barh', scale=.75)
检查叶节点统计信息
决策树在叶节点上做出决策,因此如果整个图表太大而无法一次性查看所有内容,有时候将焦点放在叶节点上是很有用的。以下是如何检查每个叶节点中分组的训练数据实例数量:
# 调用viz_cmodel的leaf_sizes方法,并设置figsize参数为(5,1.5)
viz_cmodel.leaf_sizes(figsize=(5,1.5))

也许更有趣的图表是显示各个叶子中每种训练实例的比例。训练的目标是使叶子节点具有单一颜色,因为它代表可以高度自信地预测该类别的“纯净”节点。
# 调用ctree_leaf_distributions函数,并设置图像大小为(5,1.5)
viz_cmodel.ctree_leaf_distributions(figsize=(5,1.5))
我们还可以放大特定的叶节点,查看各个实例特征的一些统计信息。例如,叶节点5包含31个实例,其中有24个实例具有唯一的bill_length_mm值:
# 调用viz_cmodel的node_stats方法,传入参数node_id=5,用于获取节点5的统计信息。
viz_cmodel.node_stats(node_id=5)
| bill_depth_mm | bill_length_mm | body_mass_g | flipper_length_mm | island | sex | year | |
|---|---|---|---|---|---|---|---|
| count | 31.0 | 31.0 | 31.0 | 31.0 | 31 | 31 | 31 |
| unique | 24.0 | 28.0 | 26.0 | 17.0 | 1 | 2 | 3 |
| top | 18.5 | 39.5 | 3300.0 | 185.0 | Dream | female | 2009 |
| freq | 4.0 | 2.0 | 2.0 | 4.0 | 31 | 19 | 11 |
决策树如何对实例进行分类
现在我们已经了解了决策树的结构和内容,让我们来弄清楚分类器如何对特定实例进行决策。通过将实例(特征向量)作为参数x传入view()函数,该函数将突出显示分类器为该实例进行预测所追求的从根到叶子的路径。
# 选择第20个样本
x = train_ds_pd[penguin_features].iloc[20]
# 调用viz_cmodel库中的view函数,可视化样本x
viz_cmodel.view(x=x, scale=.75)
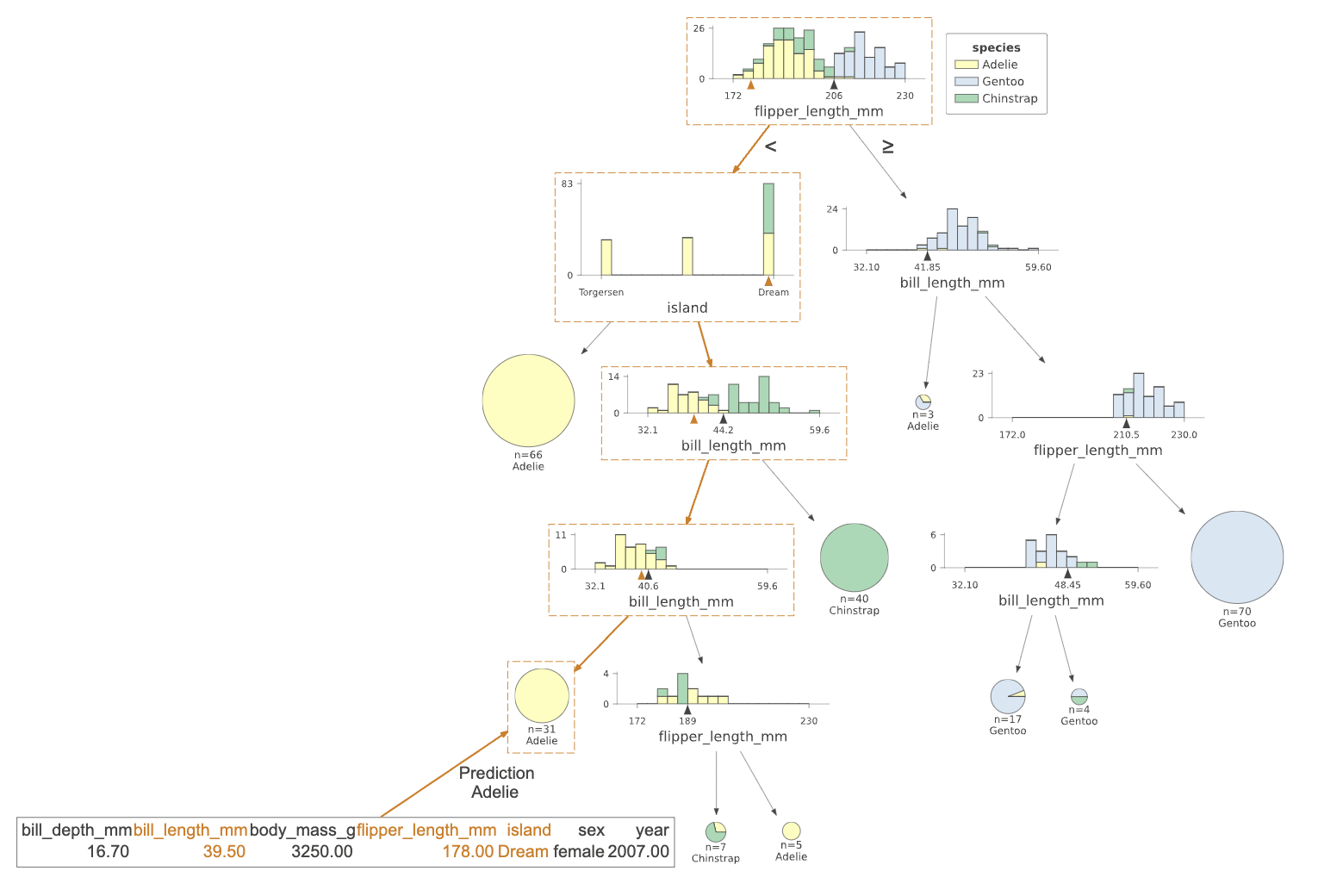
说明:
该插图突出显示了被测试的树路径和实例特征(island、bill_length_mm和flipper_length_mm)。
对于非常大的树,您还可以通过使用show_just_path参数仅查看树的路径,而不是整个树。
# 调用viz_cmodel的view方法来可视化模型
# 参数x表示输入数据
# 参数show_just_path表示只显示路径
# 参数scale表示缩放比例为0.75
viz_cmodel.view(x=x, show_just_path=True, scale=.75)
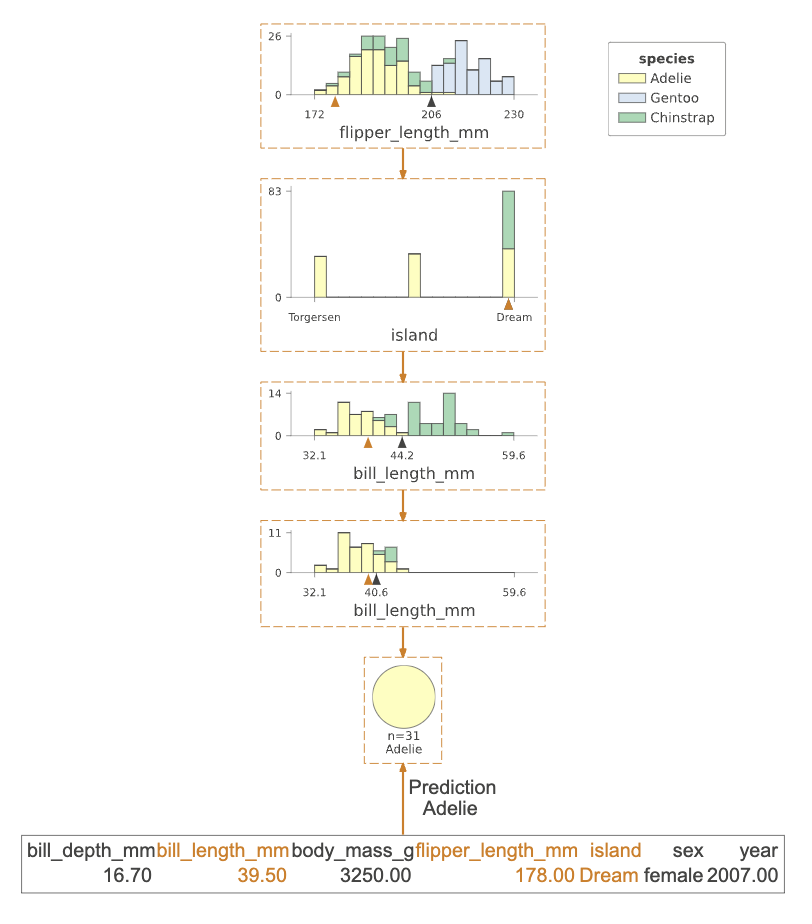
为了获得一个实例分类的英文解释,使用explain_prediction_path()函数来获取最小可能的表示。
# 打印可视化模型的解释预测路径
print(viz_cmodel.explain_prediction_path(x=x))
bill_length_mm < 40.6
flipper_length_mm < 206.0
island in {'Dream'}
模型测试 x 的 bill_length_mm、flipper_length_mm 和 island 特征,以达到叶子节点,该节点预测为 Adelie。
特征空间划分
到目前为止,我们已经了解了树的结构以及树如何解释实例以做出决策,但是决策节点到底在做什么呢?决策树将特征空间划分为一组共享相似目标值的观测值。每个叶子节点表示从根节点到该叶子节点执行的特征分裂序列所导致的分区。对于分类问题,目标是使分区共享相同或大部分相同的目标类值。
如果我们回顾一下树的结构,我们会发现变量flipper_length_mm在树中被三个节点测试。相应的决策节点分裂值为189、206和210.5,这意味着决策树将flipper_length_mm分成了四个区域,我们可以使用ctree_feature_space()来说明:
# 调用ctree_feature_space函数,并传入参数
# features参数指定要显示的特征,这里只显示'flipper_length_mm'
# show参数指定要显示的内容,这里显示'splits'和'legend'
# figsize参数指定图像的大小,这里设置为(5,1.5)
viz_cmodel.ctree_feature_space(features=['flipper_length_mm'], show={'splits','legend'}, figsize=(5,1.5))

(在这种单特征情况下,垂直轴没有意义。为了增加可见性,垂直轴只是将表示不同目标类的点分隔成不同的高度,并添加了一些噪音。)
第一个分割点在206处(在根部进行测试)将训练数据分割成了一个重叠区域,其中包含了Adelie/Gentoo Penguins,以及一个相当区域的Chinstrap Penguins。随后在210.5处的分割进一步隔离了一个纯Chinstrap区域(大于210.5的鳍长)。决策树还在189处进行了分割,但是得到的区域仍然不纯。树依靠通过其他变量进行分割来分离“混乱”的Adelie/Gentoo Penguins。因为我们只传入了一个特征名称,所以其他特征的分割没有显示出来。
让我们看看另一个具有更多分割的特征,bill_length_mm。决策树中有四个节点测试了该特征,因此我们得到了一个将特征空间分割成五个区域的结果。请注意,模型可以通过测试bill_length_mm小于40来分割出一个纯净的Adelie区域。
# 调用ctree_feature_space函数,并传入参数features=['bill_length_mm'],表示只显示bill_length_mm特征
# 参数show={'splits','legend'}表示显示决策树的分割线和图例
# 参数figsize=(5,1.5)表示设置图像的大小为5x1.5
viz_cmodel.ctree_feature_space(features=['bill_length_mm'], show={'splits','legend'}, figsize=(5,1.5))

我们还可以同时检查树如何将特征空间划分为两个特征,例如flipper_length_mm和bill_length_mm:
# 调用ctree_feature_space函数,并传入参数
# features参数指定要显示的特征,这里是'flipper_length_mm'和'bill_length_mm'
# show参数指定要显示的内容,这里是'splits'和'legend'
# figsize参数指定图像的大小,这里是(5,5)
viz_cmodel.ctree_feature_space(features=['flipper_length_mm','bill_length_mm'],
show={'splits','legend'}, figsize=(5,5))

区域的颜色表示测试实例的分类颜色,其特征落在该区域内。
通过同时考虑两个变量,决策树可以创建更加纯净(矩形)的区域,从而实现更准确的预测。例如,左上方的区域完全包含了“Chinstrap”企鹅。
根据我们选择的变量,区域的纯度会有所不同。这是另一个基于bill_depth_mm和bill_length_mm特征的二维特征空间划分,其中阴影表示不确定性。
# 使用ctree_feature_space函数绘制特征空间图
# 参数features指定要绘制的特征,这里选择了'body_mass_g'和'bill_length_mm'
# 参数show指定要显示的内容,这里选择了'splits'和'legend'
# 参数figsize指定图像的大小,这里设置为(5,5)
viz_cmodel.ctree_feature_space(features=['body_mass_g','bill_length_mm'], show={'splits','legend'}, figsize=(5,5))
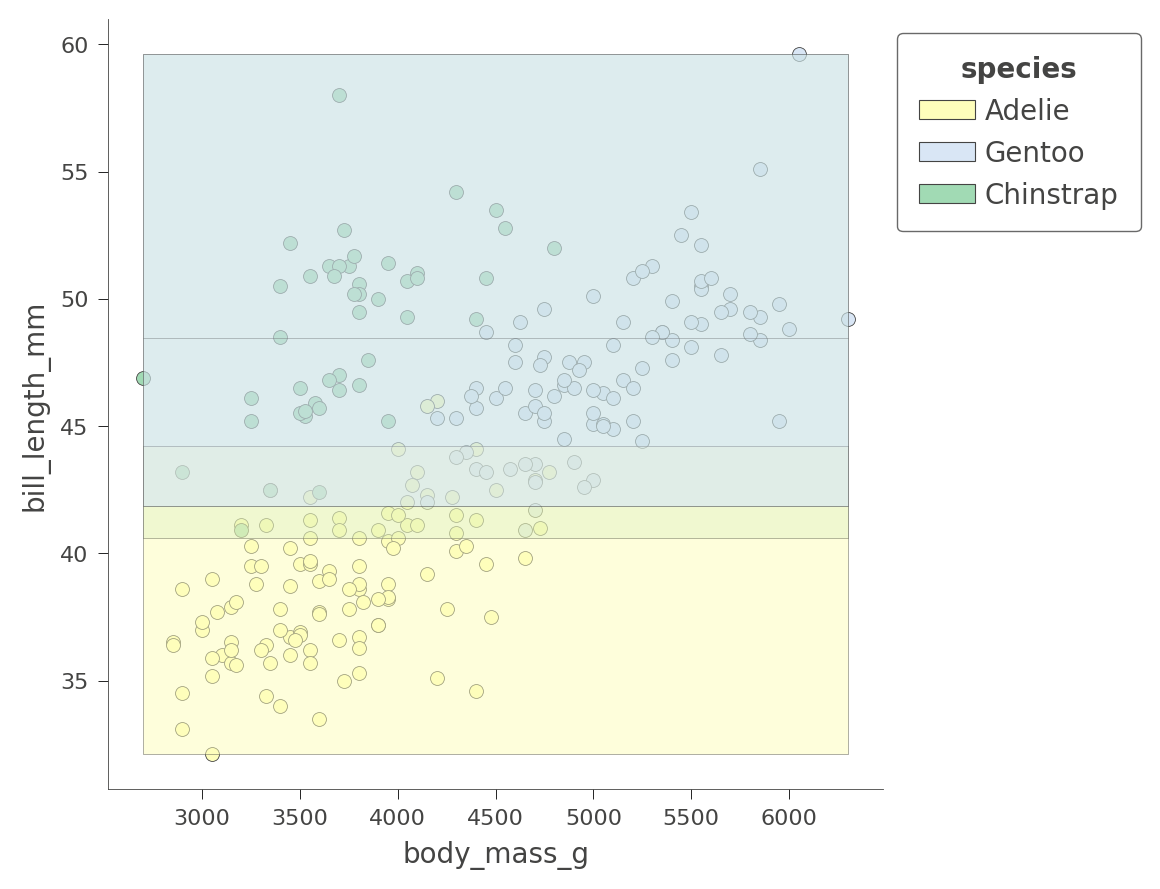
只有Adelie地区相对纯净。树依赖于其他变量来获得更好的分区,就像我们刚刚在flipper_length_mm vs bill_length_mm空间中看到的那样。
目前,dtreeviz库无法可视化超过两个特征维度的分类。
到目前为止,您已经很好地掌握了如何可视化决策树的结构,树如何分割特征空间以及树如何对测试实例进行分类。现在让我们转向回归,看看dtreeviz如何可视化回归树。
可视化回归树
 让我们使用初学者教程中使用的鲍鱼数据集来探索回归树的结构。与上面的分类相同,我们首先加载和准备训练数据。给定8个变量,我们想预测鲍鱼壳中的环数。
让我们使用初学者教程中使用的鲍鱼数据集来探索回归树的结构。与上面的分类相同,我们首先加载和准备训练数据。给定8个变量,我们想预测鲍鱼壳中的环数。
加载、清洗和准备数据
使用以下代码片段,我们可以看到除了 Type(性别)变量之外,所有特征都是数值型的。
# 下载数据集
!wget -q https://storage.googleapis.com/download.tensorflow.org/data/abalone_raw.csv -O /tmp/abalone.csv
# 读取CSV文件并将数据存储在DataFrame中
df_abalone = pd.read_csv("/tmp/abalone.csv")
# 显示DataFrame的前3行数据
df_abalone.head(3)
| Type | LongestShell | Diameter | Height | WholeWeight | ShuckedWeight | VisceraWeight | ShellWeight | Rings | |
|---|---|---|---|---|---|---|---|---|---|
| 0 | M | 0.455 | 0.365 | 0.095 | 0.5140 | 0.2245 | 0.1010 | 0.15 | 15 |
| 1 | M | 0.350 | 0.265 | 0.090 | 0.2255 | 0.0995 | 0.0485 | 0.07 | 7 |
| 2 | F | 0.530 | 0.420 | 0.135 | 0.6770 | 0.2565 | 0.1415 | 0.21 | 9 |
幸运的是,没有缺失的数据需要处理:
# 使用isna()方法检查数据集中是否存在缺失值,any()方法判断是否存在缺失值
df_abalone.isna().any()
Type False
LongestShell False
Diameter False
Height False
WholeWeight False
ShuckedWeight False
VisceraWeight False
ShellWeight False
Rings False
dtype: bool
分割训练/测试集并训练模型
# 定义分类目标标签名称为 "Rings"
abalone_label = "Rings"
# 将数据集按照 70/30 的比例分为训练集和测试集
df_train_abalone, df_test_abalone = split_dataset(df_abalone)
# 输出训练集和测试集的样本数量
print(f"{len(df_train_abalone)} examples in training, {len(df_test_abalone)} examples for testing.")
# 将数据集转换为 TensorFlow 数据集
train_ds = tfdf.keras.pd_dataframe_to_tf_dataset(df_train_abalone, label=abalone_label, task=tfdf.keras.Task.REGRESSION)
test_ds = tfdf.keras.pd_dataframe_to_tf_dataset(df_test_abalone, label=abalone_label, task=tfdf.keras.Task.REGRESSION)
2935 examples in training, 1242 examples for testing.
训练一个随机森林回归器
现在我们有了训练集和测试集,让我们来训练一个随机森林回归器。由于数据的特性,我们需要人为地限制树的高度以便进行可视化。(限制树的深度也是一种正则化的形式,用于防止过拟合。)深度为5足够准确,同时又足够小以进行可视化。
# 创建一个随机森林模型
rmodel = tfdf.keras.RandomForestModel(task=tfdf.keras.Task.REGRESSION, # 设置任务为回归
max_depth=5, # 设置树的最大深度为5,避免树过大
random_seed=1234, # 设置随机种子,确保每次创建相同的树
verbose=0) # 设置不显示训练过程中的详细信息
# 使用训练数据集进行模型训练
rmodel.fit(x=train_ds)
[INFO 2023-03-07T12:11:19.959239957+00:00 kernel.cc:1214] Loading model from path /tmpfs/tmp/tmpdts8fzxf/model/ with prefix a5115ef6d4b2486a
[INFO 2023-03-07T12:11:19.98628563+00:00 decision_forest.cc:661] Model loaded with 300 root(s), 9264 node(s), and 8 input feature(s).
[INFO 2023-03-07T12:11:19.986325053+00:00 abstract_model.cc:1311] Engine "RandomForestOptPred" built
[INFO 2023-03-07T12:11:19.986350895+00:00 kernel.cc:1046] Use fast generic engine
<keras.callbacks.History at 0x7f68310dd430>
让我们使用MAE和MSE来检查模型的准确性。Rings的范围是1-27,所以测试集上的MAE为1.66并不是很好,但对于我们的演示目的来说还可以。
# 编译模型,指定评估指标为平均绝对误差(MAE)和均方误差(MSE)
rmodel.compile(metrics=["mae","mse"])
# 在测试数据集上评估模型,并返回评估结果
evaluation = rmodel.evaluate(test_ds, return_dict=True, verbose=0)
# 打印均方误差(MSE)
print(f"MSE: {evaluation['mse']}")
# 打印平均绝对误差(MAE)
print(f"MAE: {evaluation['mae']}")
# 打印均方根误差(RMSE),通过对均方误差取平方根得到
print(f"RMSE: {math.sqrt(evaluation['mse'])}")
MSE: 5.4397759437561035
MAE: 1.6559592485427856
RMSE: 2.3323327257825164
显示决策树
要使用dtreeviz,我们需要将模型和训练数据捆绑在一起。我们还必须选择要显示的随机森林中的特定树;让我们选择树3,就像我们对分类问题所做的那样。
# 创建一个列表abalone_features,其中包含了rmodel模型的所有特征的名称
abalone_features = [f.name for f in rmodel.make_inspector().features()]
# 使用dtreeviz库中的model函数创建一个决策树可视化模型viz_rmodel
# 设置tree_index参数为3,表示选择第三棵决策树进行可视化
# 使用X_train参数传入训练集的特征数据df_train_abalone[abalone_features]
# 使用y_train参数传入训练集的目标数据df_train_abalone[abalone_label]
# 使用feature_names参数传入特征的名称列表abalone_features
# 使用target_name参数传入目标变量的名称'Rings'
viz_rmodel = dtreeviz.model(rmodel, tree_index=3,
X_train=df_train_abalone[abalone_features],
y_train=df_train_abalone[abalone_label],
feature_names=abalone_features,
target_name='Rings')
功能view()显示了树的结构,但现在决策节点是散点图而不是堆叠条形图。每个决策节点显示了指定变量与目标(Rings)的边际图。
# 调用viz_rmodel的view方法,并设置缩放比例为1.2,用于可视化rmodel模型。
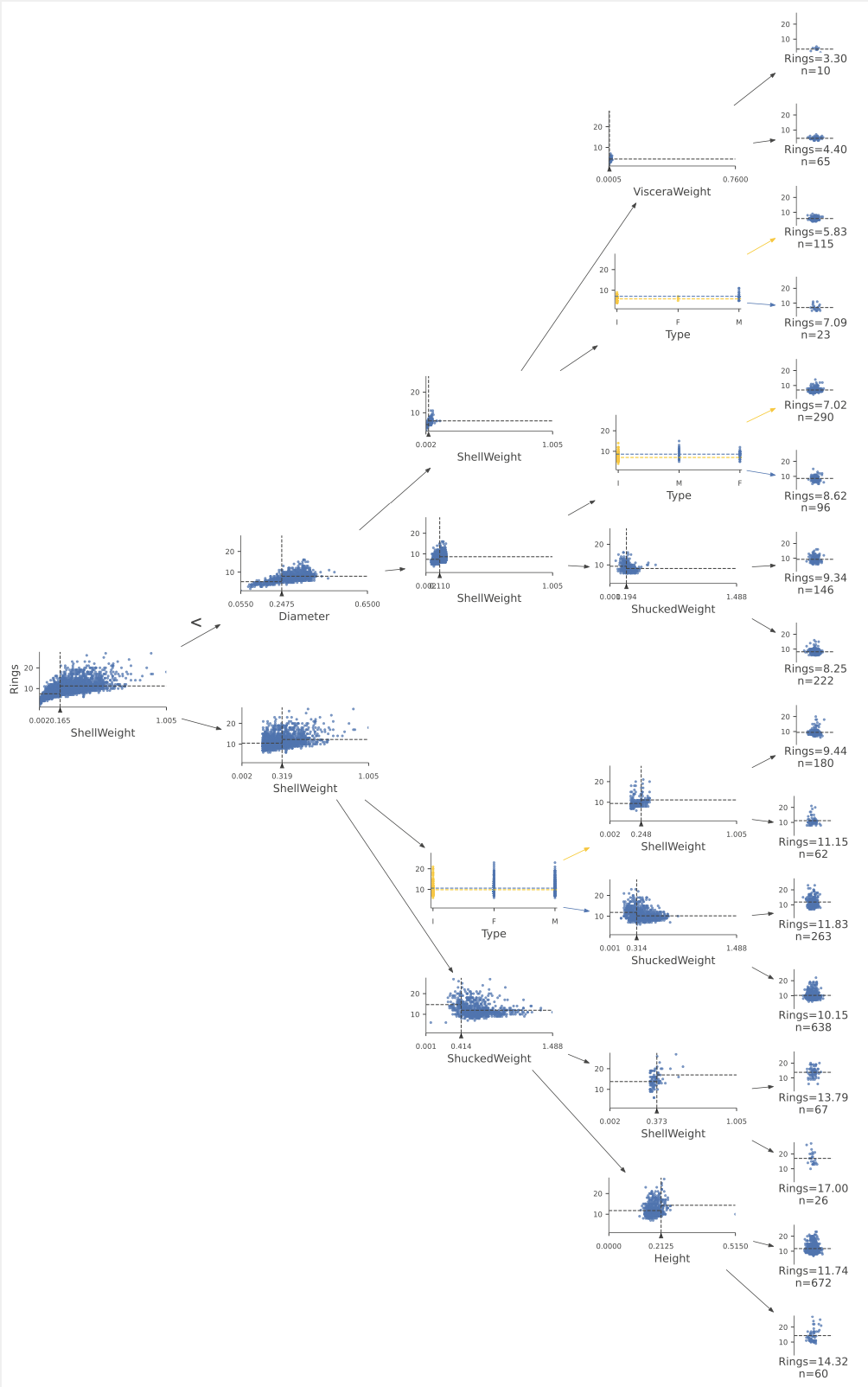
viz_rmodel.view(scale=1.2)
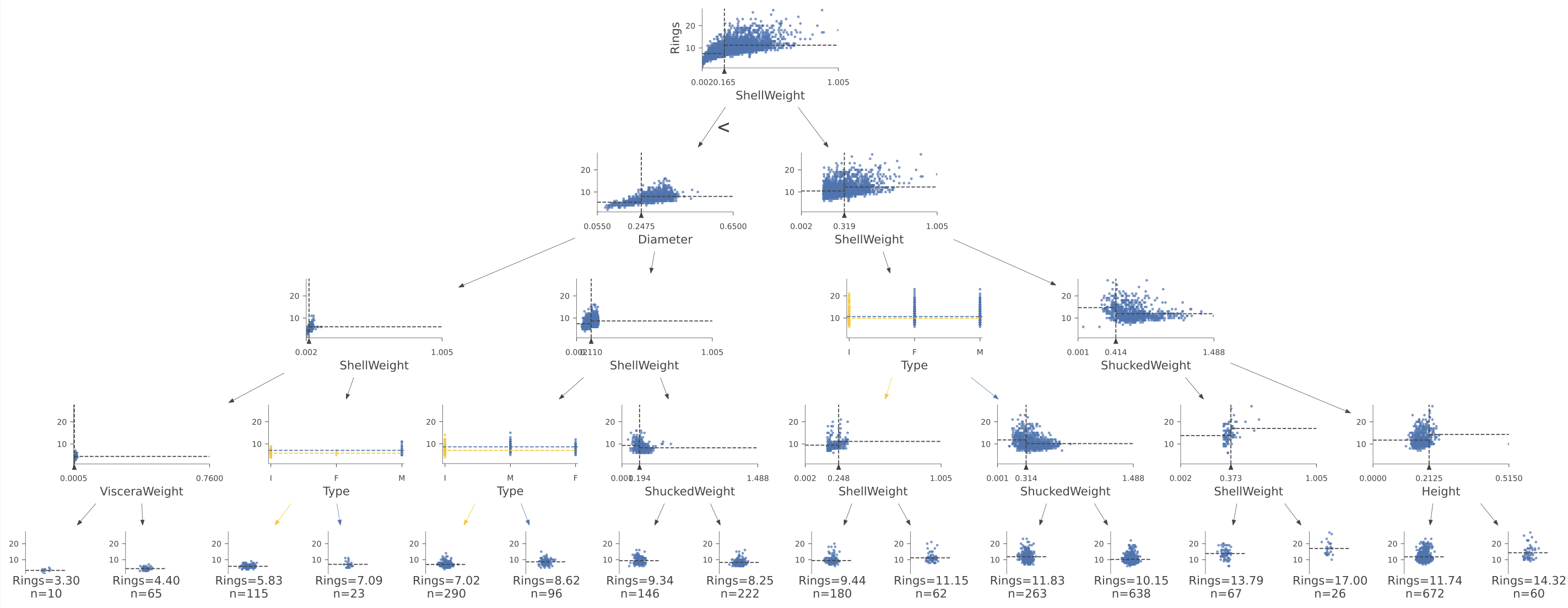
与分类一样,回归从树的根部向特定叶子前进,最终为特定的测试实例进行预测。通往叶子的路径上的节点测试数值或分类变量,将回归器引导到具有非常相似目标值的特定特征空间区域(希望如此)。
叶子是条带图,显示叶子中所有实例的目标变量“Rings”的值。水平参数没有意义,只是一点噪音,用于分隔点,以便我们可以看到密度分布在哪里。考虑左下角的叶子,n=10,Rings=3.30。这表示该叶子中10个实例的平均“Rings”值为3.30,这也是决策树对达到该叶子的任何测试实例的预测结果。
让我们放大树的根部,看看回归器如何根据变量“ShellWeight”进行分割:
# 调用viz_rmodel库中的view函数,并传入参数depth_range_to_display=[0,0]和scale=2
viz_rmodel.view(depth_range_to_display=[0,0], scale=2)
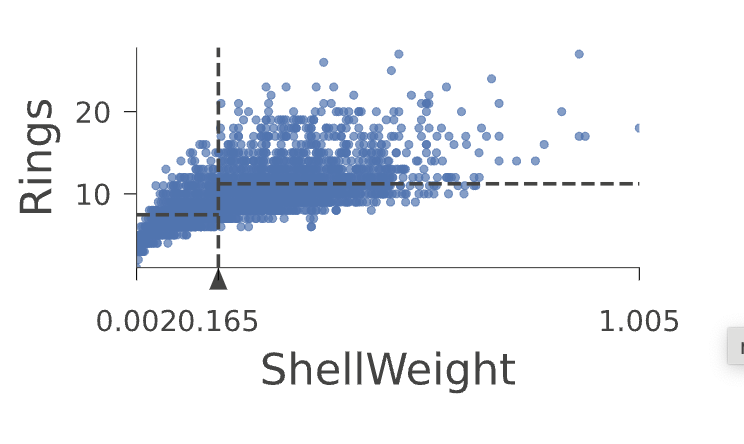
对于一个具有ShellWeight<0.164的测试实例,回归器会沿着根节点的左子节点进行处理;否则,它会沿着右子节点进行处理。水平虚线表示与ShellWeight大于或小于0.164的实例相关联的平均Rings值。
另一方面,对于分类变量,决策节点测试类别的子集,因为类别是无序的。在树的第四层中,有两个测试分类变量Type的决策节点:
# 调用viz_rmodel的view方法来显示可视化结果
# depth_range_to_display参数指定了要显示的深度范围,这里设置为[3,3],表示只显示深度为3的部分
# scale参数指定了显示的缩放比例,这里设置为1.5,表示放大1.5倍显示结果
viz_rmodel.view(depth_range_to_display=[3,3], scale=1.5)
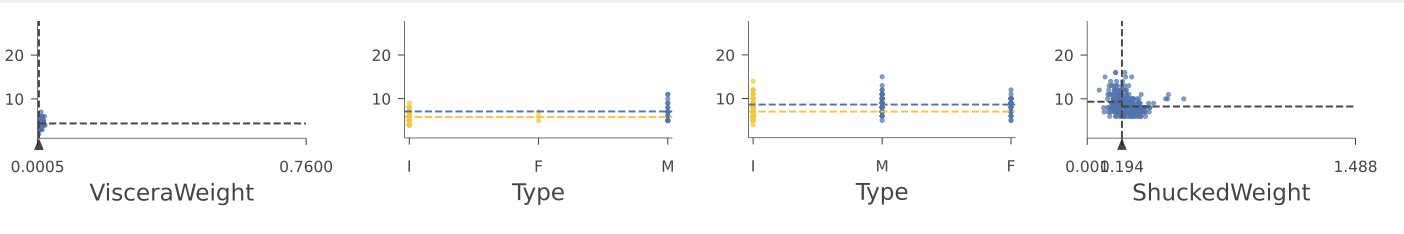
分类器节点使用颜色来指示子集。例如,第四层左侧的决策节点指示分类器在测试实例的Type=I或Type=F时向左下降;否则,分类器向右下降。黄色和蓝色表示与左右分支相关联的两个分类值子集。水平虚线表示具有相关分类值的实例的平均Rings目标值。
要显示大型树,可以使用orientation参数获得从左到右的树的版本,尽管它相当高,因此使用scale来缩小它是一个好主意。使用计算机上的屏幕缩放功能,可以放大感兴趣的区域。
# 调用view函数,设置参数orientation为'LR',表示水平方向从左到右排列;设置参数scale为0.5,表示缩放比例为0.5
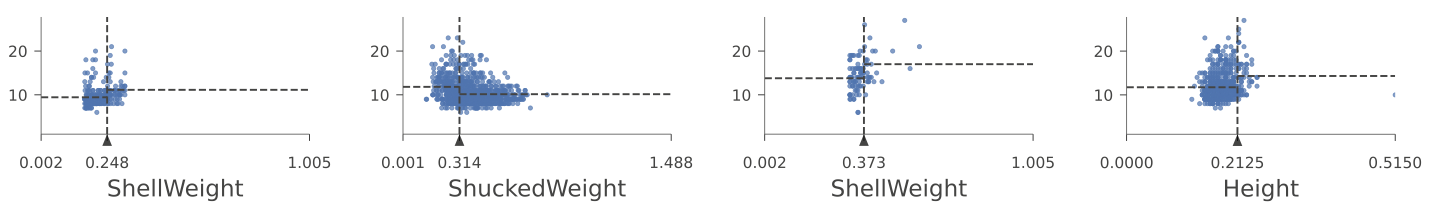
viz_rmodel.view(orientation='LR', scale=.5)
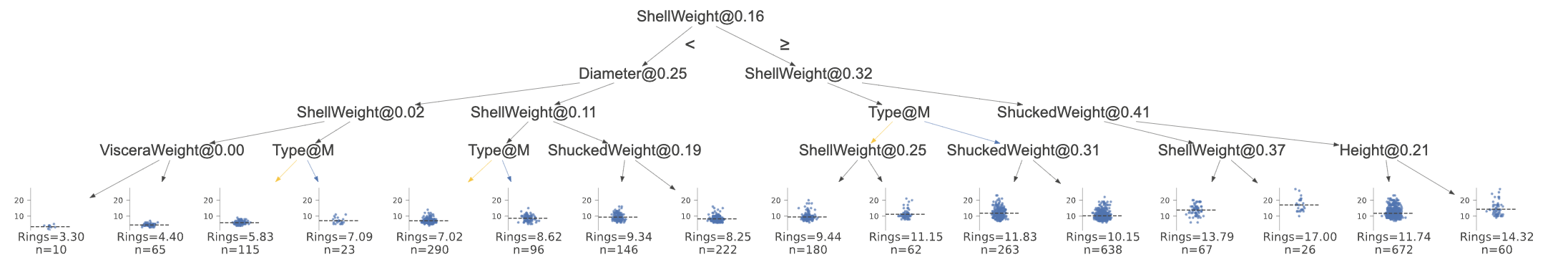
我们可以使用非花哨的图表来节省空间。它仍然显示决策节点的分裂变量和分裂点;只是不太漂亮。
# 使用viz_rmodel库中的view函数来可视化模型
# 参数fancy设置为False,表示不使用复杂的样式
# 参数scale设置为0.75,表示缩放比例为0.75
viz_rmodel.view(fancy=False, scale=.75)
检查叶子节点统计信息
当图形变得非常大时,有时候更好地关注叶子节点。函数leaf_sizes()指示每个叶子节点中找到的实例数量:
# 调用leaf_sizes函数,并设置figsize参数为(5,1.5),用于指定绘图的大小
viz_rmodel.leaf_sizes(figsize=(5,1.5))
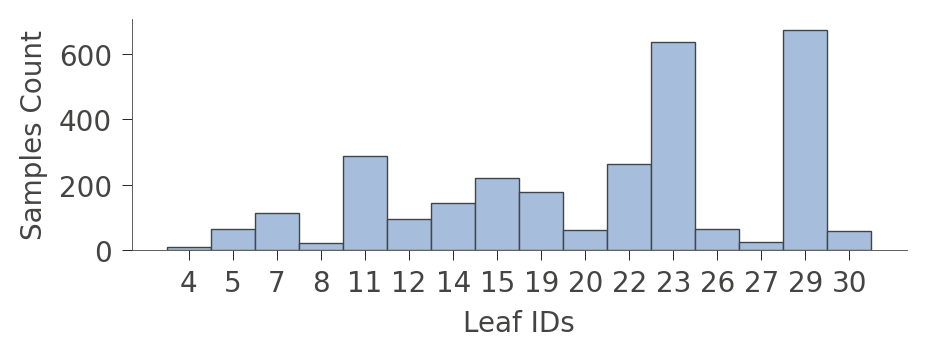
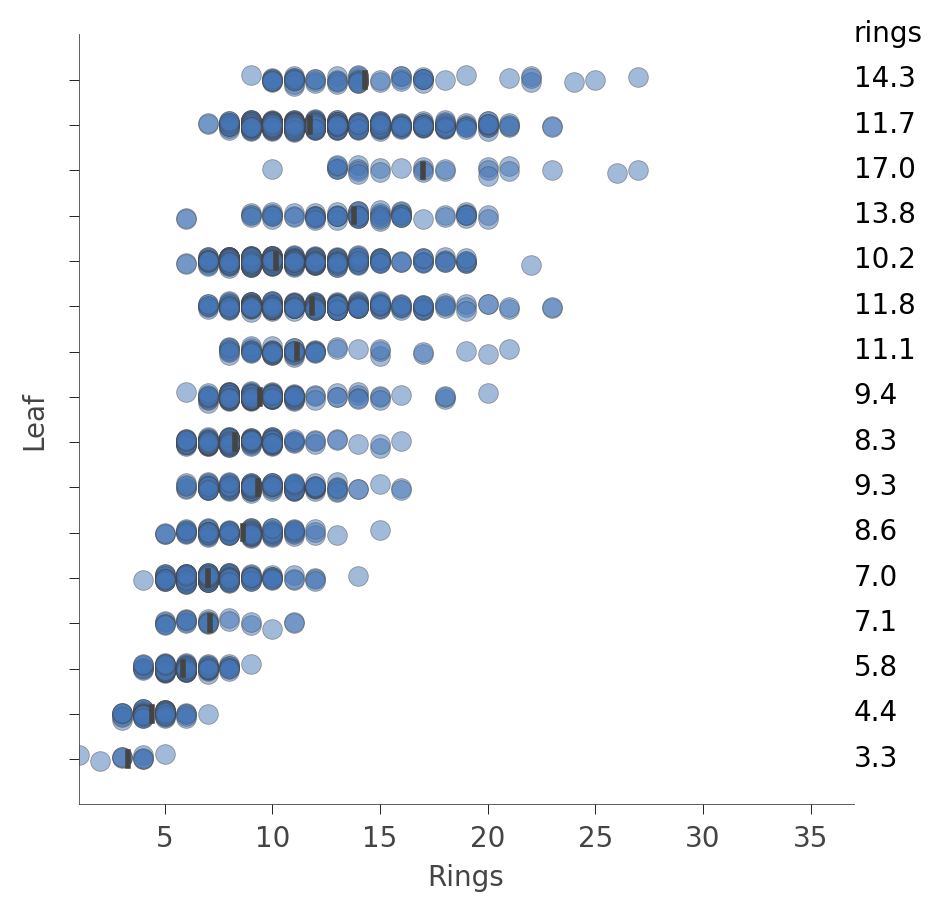
我们还可以查看叶子节点中实例的分布(Rings值)。垂直轴上每个叶子节点有一行,水平轴显示每个叶子节点中实例的Rings值的分布。右侧的列显示每个叶子节点的平均目标值。
# 调用viz_rmodel库中的rtree_leaf_distributions函数,并设置图像大小为(5,5)
viz_rmodel.rtree_leaf_distributions(figsize=(5,5))
或者,我们可以获取特定节点中实例特征的信息。例如,以下是如何获取叶节点29中特征的信息,该叶节点具有最多的实例:
# 调用viz_rmodel模块中的node_stats函数
# 传入参数node_id=29,表示要获取节点ID为29的统计信息
viz_rmodel.node_stats(node_id=29)
| Diameter | Height | LongestShell | ShellWeight | ShuckedWeight | Type | VisceraWeight | WholeWeight | |
|---|---|---|---|---|---|---|---|---|
| count | 672.0 | 672.000 | 672.00 | 672.000 | 672.0000 | 672 | 672.000 | 672.000 |
| unique | 42.0 | 18.000 | 48.00 | 262.000 | 483.0000 | 3 | 363.000 | 556.000 |
| top | 0.5 | 0.175 | 0.65 | 0.335 | 0.5985 | F | 0.318 | 1.262 |
| freq | 66.0 | 115.000 | 44.00 | 22.000 | 5.0000 | 328 | 11.000 | 4.000 |
决策树如何预测实例的值
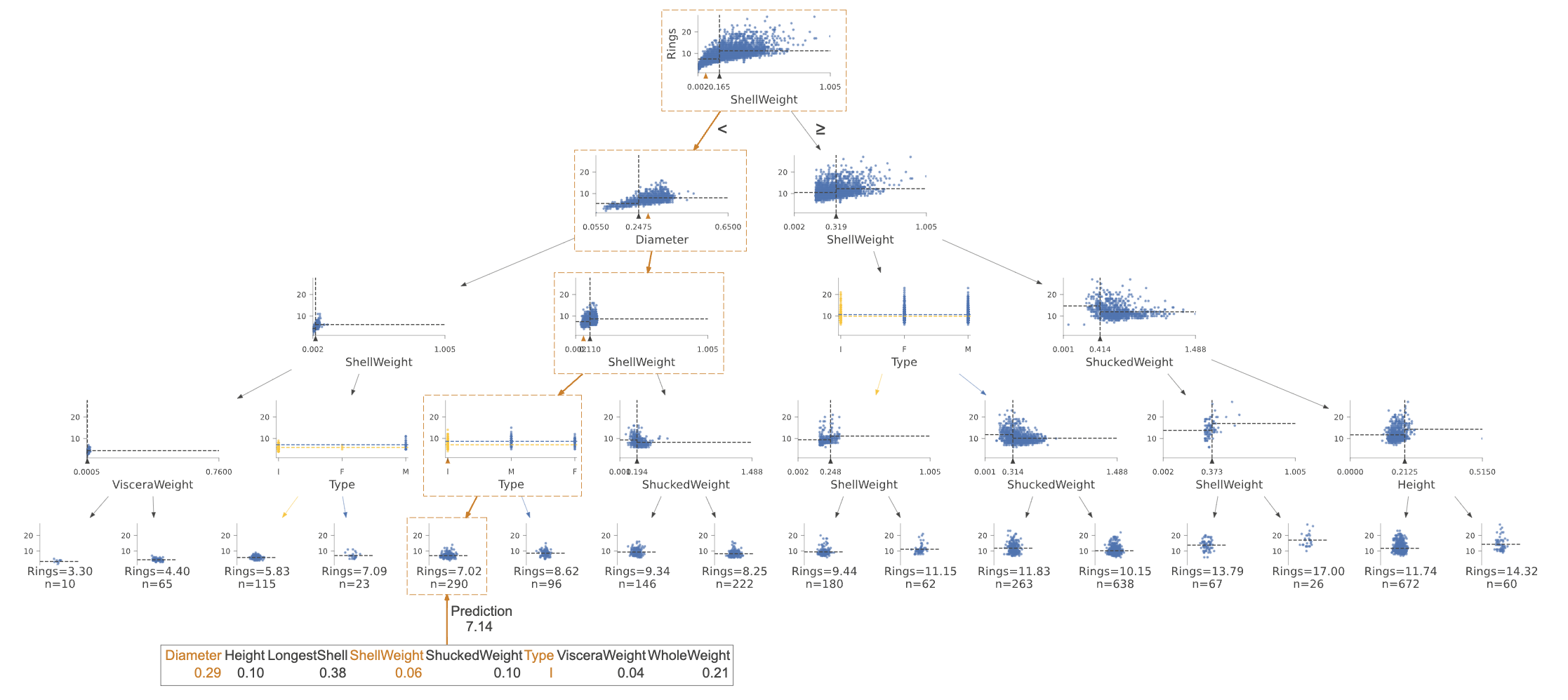
为了对特定实例进行预测,决策树根据测试实例中的特征值从根节点向下延伸到特定叶节点。单个树的预测值只是该叶节点中驻留的实例(来自训练集)的Rings值的平均值。如果我们通过参数x提供一个测试实例,dtreeviz库可以说明这个过程。
# 从df_abalone数据集中获取第1234行的数据
x = df_abalone[abalone_features].iloc[1234]
# 调用viz_rmodel库中的view函数,可视化数据
viz_rmodel.view(x=x, scale=.75)
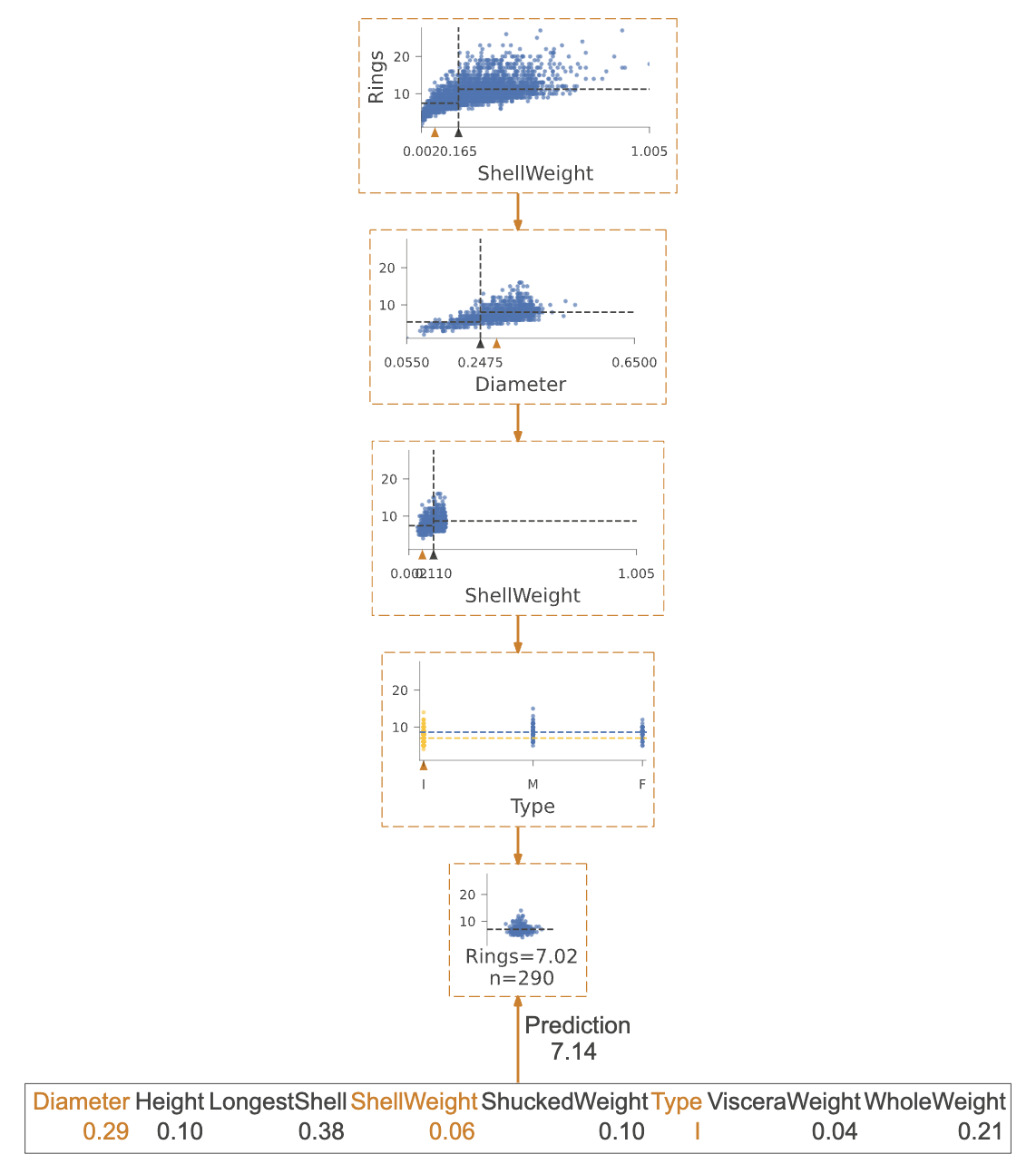
如果该可视化太大,我们可以将图表缩小到实际遍历的从根到叶子的路径。
# 调用viz_rmodel的view方法来可视化模型
# 参数x表示输入数据
# 参数show_just_path表示只显示路径
# 参数scale表示缩放比例为1.0
viz_rmodel.view(x=x, show_just_path=True, scale=1.0)
我们可以使用水平方向来使其变得更小:
# 调用viz_rmodel的view函数来可视化模型
# 参数x表示模型
# 参数show_just_path表示只显示路径
# 参数scale表示缩放比例
# 参数orientation表示图的方向为从左到右
viz_rmodel.view(x=x, show_just_path=True, scale=.75, orientation="LR")
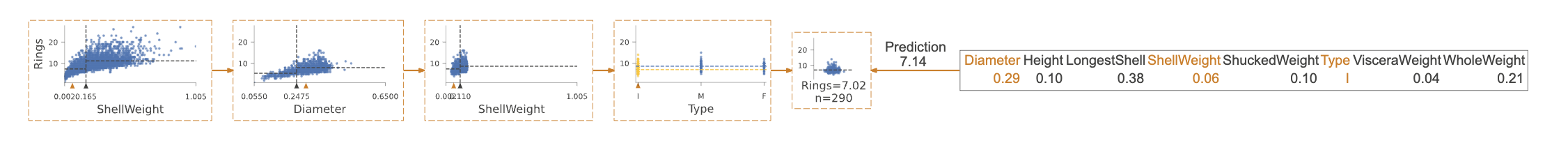
有时候,获取一个英文描述来了解模型如何测试我们的特征值以做出决策会更容易:
# 打印可视化模型的预测解释路径
# 参数 x 为输入数据
print(viz_rmodel.explain_prediction_path(x=x))
0.25 <= Diameter
ShellWeight < 0.11
Type not in {'M', 'F'}
特征空间划分
使用rtree_feature_space()函数,我们可以看到决策树通过一系列的分割来划分特征空间。例如,下面是决策树如何划分特征ShellWeight的示例:
# 使用rtree_feature_space函数来生成特征空间的可视化图表
# features参数指定要显示的特征,这里只显示'ShellWeight'
# show参数指定要显示的内容,这里只显示'splits'
viz_rmodel.rtree_feature_space(features=['ShellWeight'], show={'splits'})

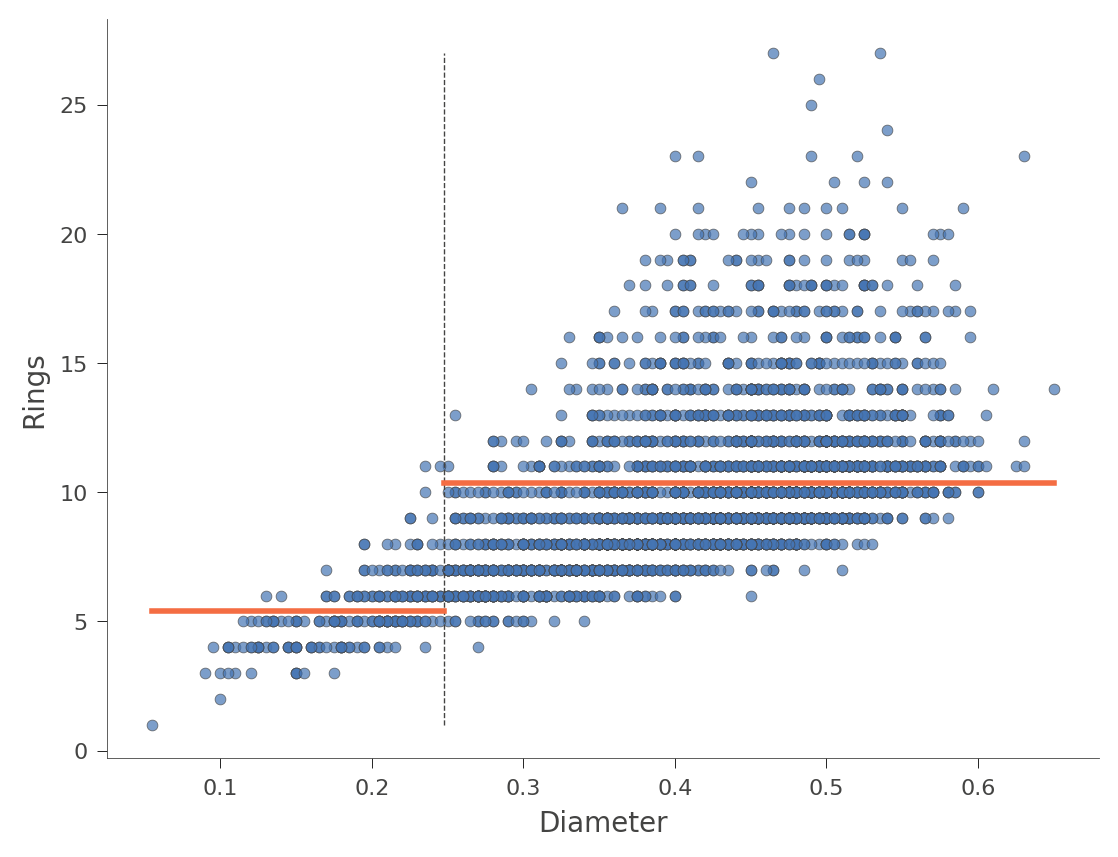
水平的橙色条表示每个区域内的平均“Rings”值。这是另一个使用特征“Diameter”的示例(树中只有一个分割点):
# 调用rtree_feature_space函数,传入参数features=['Diameter']和show={'splits'}
# features参数指定了要在可视化中展示的特征,这里只展示了直径(Diameter)这一项
# show参数指定了要展示的内容,这里指定了展示决策树的分裂情况(splits)
viz_rmodel.rtree_feature_space(features=['Diameter'], show={'splits'})
我们还可以查看二维特征空间,在这个空间中,“Rings”值的颜色从绿色(低)到蓝色(高)变化:
# 创建一个可视化模型对象
viz_model = viz_rmodel.rtree_feature_space(features=['ShellWeight','LongestShell'], show={'splits'})
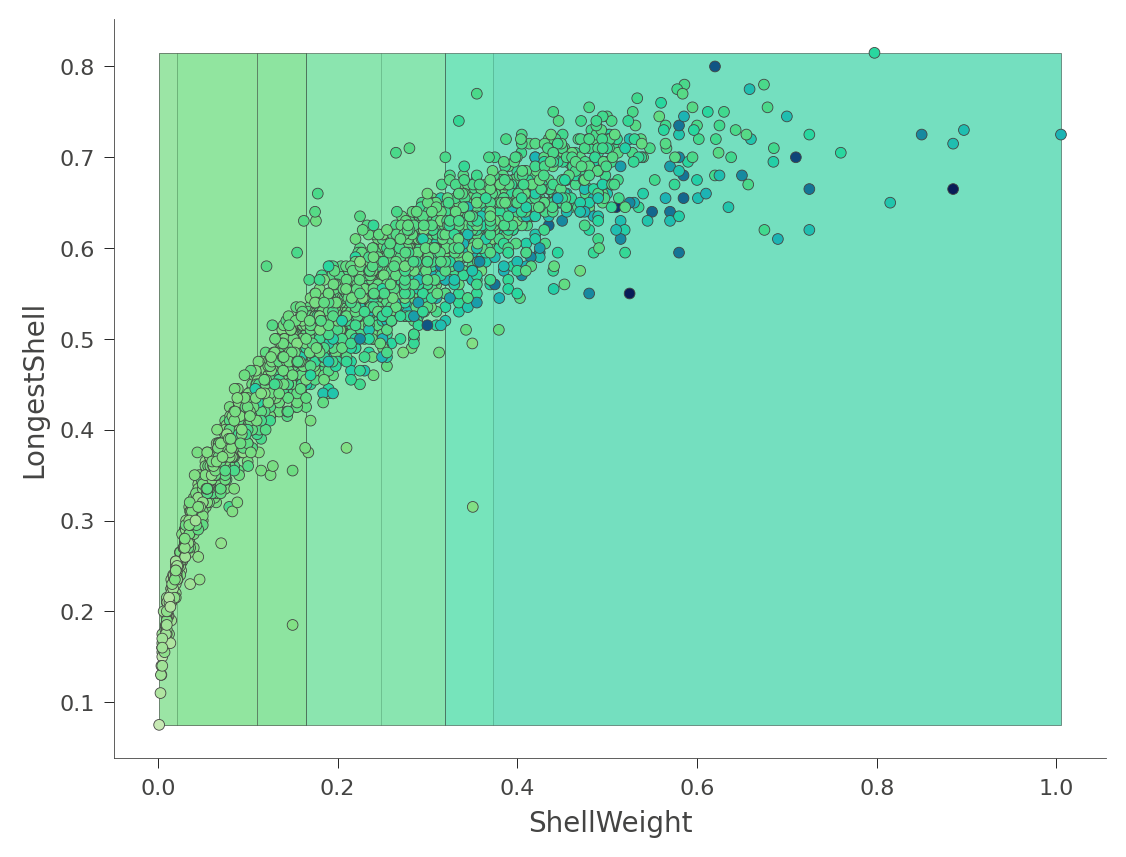
那个热力图可能会让人感到困惑,因为它实际上是一个三维空间的二维投影:两个特征 x 目标值。相反,dtreeviz可以向您展示这个三维图(从各种角度和高度)。
# 创建一个3D特征空间图
# 参数features指定要在图中显示的特征,这里选择了'ShellWeight'和'LongestShell'
# 参数show指定要在图中显示的内容,这里选择了'splits',表示显示决策树的分割线
# 参数elev、azim和dist分别指定了视角的高度、方位和距离
# 参数figsize指定了图的大小
viz_rmodel.rtree_feature_space3D(features=['ShellWeight','LongestShell'],
show={'splits'}, elev=30, azim=140, dist=11, figsize=(9,8))
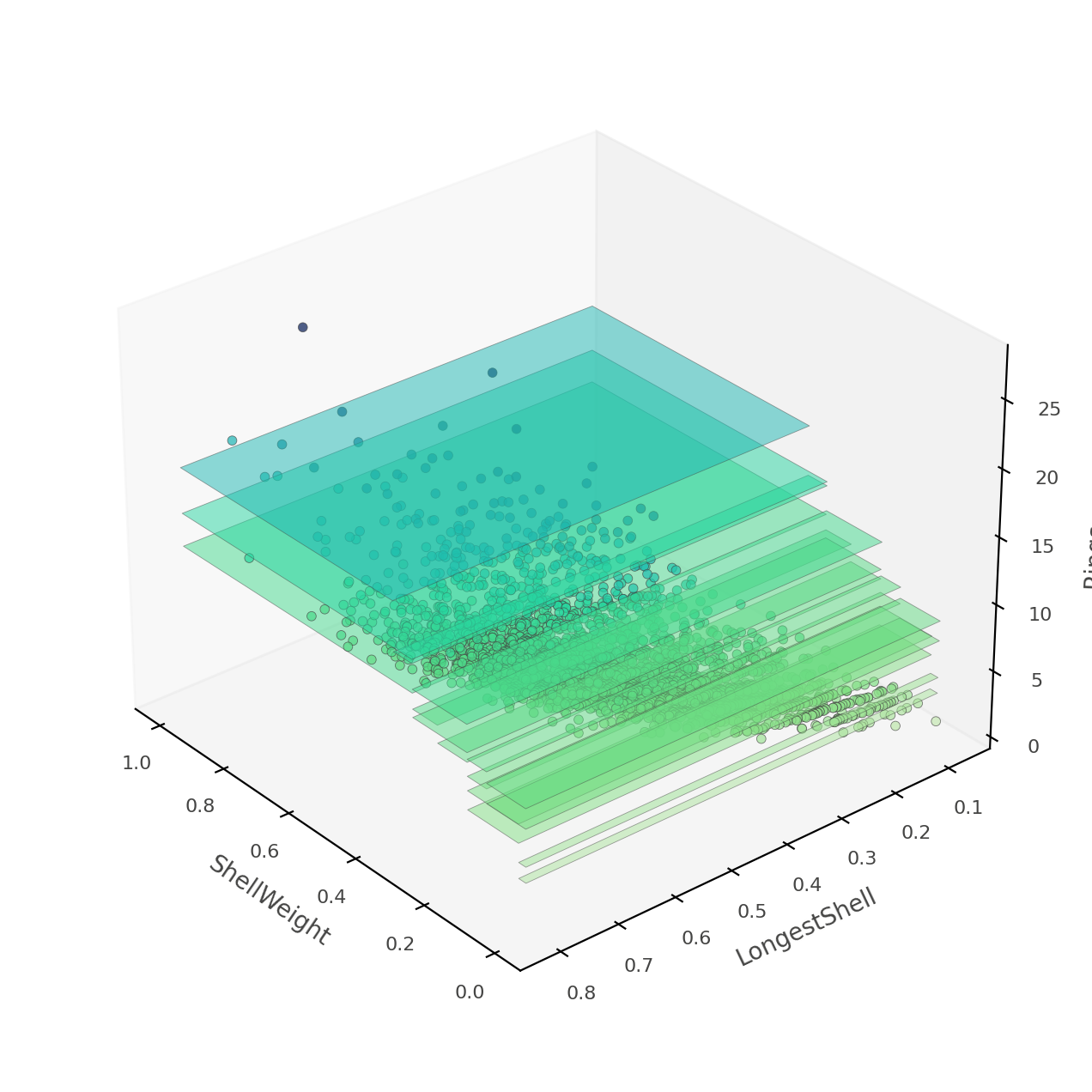
如果模型只测试了 ShellWeight 和 LongestShell 两个特征,那么就不会有重叠的垂直“板块”。每个特征空间的二维区域都会做出独特的预测。在这棵树中,还有其他特征可以区分模糊的垂直预测区域。
在这个阶段,你已经学会了如何使用dtreeviz来展示决策树的结构,绘制叶子节点信息,跟踪模型如何解释特定实例以及模型如何划分未来空间。你已经准备好使用自己的数据集可视化和解释树了!
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:chenni525@qq.com进行投诉反馈,一经查实,立即删除!
- Python教程
- 深入理解 MySQL 中的 HAVING 关键字和聚合函数
- Qt之QChar编码(1)
- MyBatis入门基础篇
- 用Python脚本实现FFmpeg批量转换
- 跳跃游戏-算法
- 网络安全35个框架合集
- faiss windows 安装使用
- HTML基础
- 线程学习(3)-volatile关键字,wait/notify的使用
- 《Python数据分析技术栈》第01章 02 Jupyter入门(Getting started with Jupyter notebooks)
- 力扣844 比较含退格的字符串--双指针
- SpringSecurity集成JWT实现后端认证授权保姆级教程-数据准备篇
- Redis实战之-分布式锁-redission
- Ubuntu 在更新内核后 Virtual Box 不能为虚拟电脑打开一个新任务