泛癌+单细胞+预后模型,纯生信思路也能轻取12+

导语
今天给同学们分享一篇生信文章“Integrated analysis of single-cell and bulk RNA sequencing data reveals a pan-cancer stemness signature predicting immunotherapy response”,这篇文章发表在Genome Med期刊上,影响因子为12.3。

结果解读:
癌症干细胞性与免疫检查点抑制剂抵抗性相关
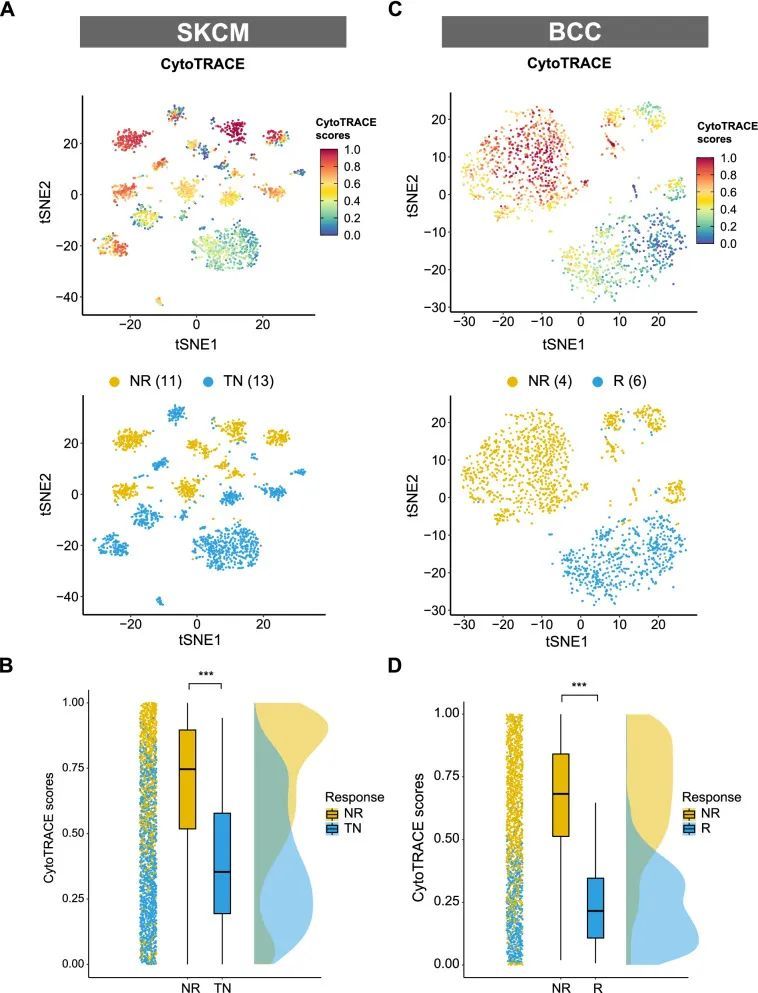
首先,作者使用之前发表的ICI SKCM队列的单细胞RNA测序数据来评估癌症干细胞性和ICI治疗结果之间的关联[14]。在删除没有恶性细胞数据的患者后,作者从该队列中选择了24名患者,其中包括11名非应答者(NR)和13名未接受治疗的患者(TN)。理想情况下,作者希望比较应答者(R)和非应答者之间的癌症干细胞性。然而,在这个队列中没有应答者的数据。鉴于未接受治疗的患者可能包括潜在的应答者和非应答者,作者按照之前的描述将干细胞性进行了NR和TN之间的比较[14]。如图1A所示,高干细胞性的癌细胞在NR亚组中富集。进一步的分析显示,NR亚组的肿瘤干细胞性水平显著较高(P < 0.001,图1B),表明癌症干细胞性与ICI治疗结果呈负相关。另一个不同癌症类型(BCC)的ICI队列被用来验证这一发现[15]。在BCC队列中,将4名非应答者的肿瘤干细胞性与6名应答者进行了比较。作者在BCC队列中发现了NR和R亚组之间更显著的干细胞水平差异(P < 0.001,图1C和D)。
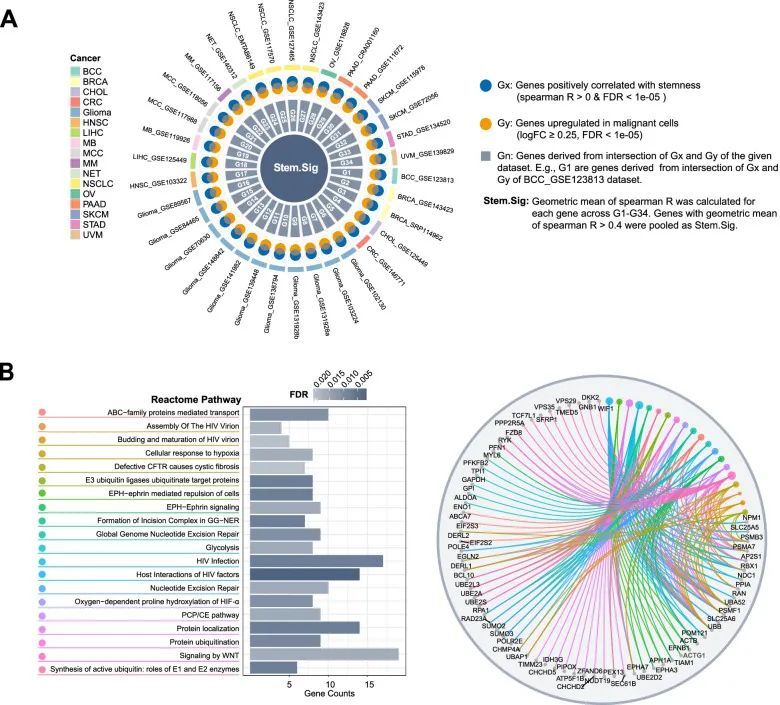
通过泛癌单细胞RNA分析的发展Stem.Sig
由于癌症干细胞性与免疫检查点抵抗力显著相关,作者假设一个反映肿瘤干细胞水平的Stem.Sig可能有助于预测免疫检查点疗效。因此,作者使用了34个单细胞RNA测序数据集来开发Stem.Sig(图2A)。作者在全癌症单细胞RNA数据集中对基因表达水平与恶性细胞的CytoTRACE分数进行了Spearman相关分析。与CytoTRACE分数呈正相关(Spearman R > 0且FDR < 1e?05)的基因被视为Gx。在恶性细胞中差异上调的基因被视为Gy。为了获得与干细胞性正相关的上调的肿瘤特异性基因,作者对每个数据集的Gx和Gy进行了交集运算[14]。例如,G0由第一个单细胞RNA测序数据集中Gx和Gy的交集基因组成。对于G1-G34中的每个基因,计算了Spearman R的几何平均值。最后,将几何平均值大于0.4(中度到强相关)的基因汇总为Stem.Sig[87]。
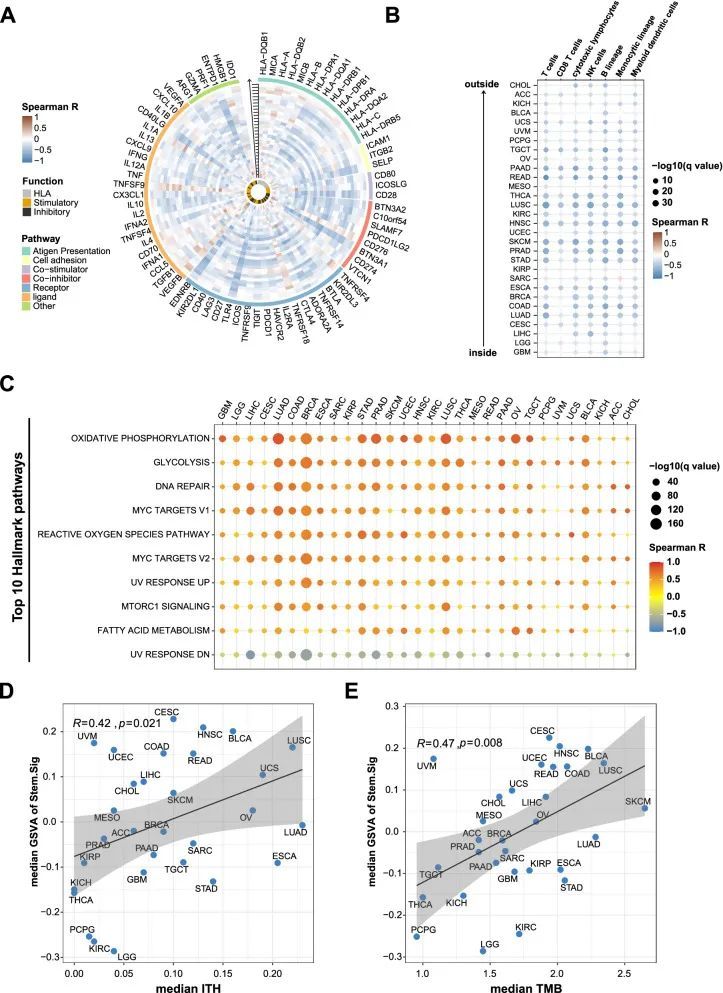
对Stem.Sig与免疫抑制之间的潜在联系进行泛癌TCGA队列的分析
首先,作者对Stem.Sig和75个免疫相关基因进行了彻底的分析[66]。在30种不同的癌症类型中观察到了Stem.Sig与免疫相关基因表达水平之间的普遍负相关(图3A)。然后,作者评估了免疫细胞的浸润状态,以更好地表征肿瘤免疫微环境(TIME)。高Stem.Sig的肿瘤中细胞毒性免疫细胞,包括CD8 T细胞、NK细胞和巨噬细胞,数量减少(图3B)。综上所述,这些结果表明Stem.Sig与抗肿瘤免疫之间存在负相关。
免疫疗法结果预测通过Stem.Sig
为了研究Stem.Sig的预测价值,作者收集了来自10个ICI队列的批量RNA-Seq数据和临床信息。对这些队列的治疗前样本进行了整理和分析。患者接受了抗PD(L)-1、抗CTLA-4或抗PD(L)-1加抗CTLA-4的治疗。这10个队列被分为3个数据集:训练集(n=620)、验证集(n=154)和测试集(n=149)。分析过程的流程图如图4A所示。首先,作者使用七种不同的机器学习算法对模型进行训练,并对每个模型进行了10次重复的5折交叉验证以进行参数优化。训练完成后,作者得到了七个模型。然后,作者在验证队列中评估和比较了这些模型的AUC。朴素贝叶斯模型的AUC达到了0.71,并被选为Stem.Sig模型(图4B)。为了进一步评估Stem.Sig模型,作者将其应用于独立的测试集,以预测ICI的反应,并观察到相同的AUC为0.71(图4C)。
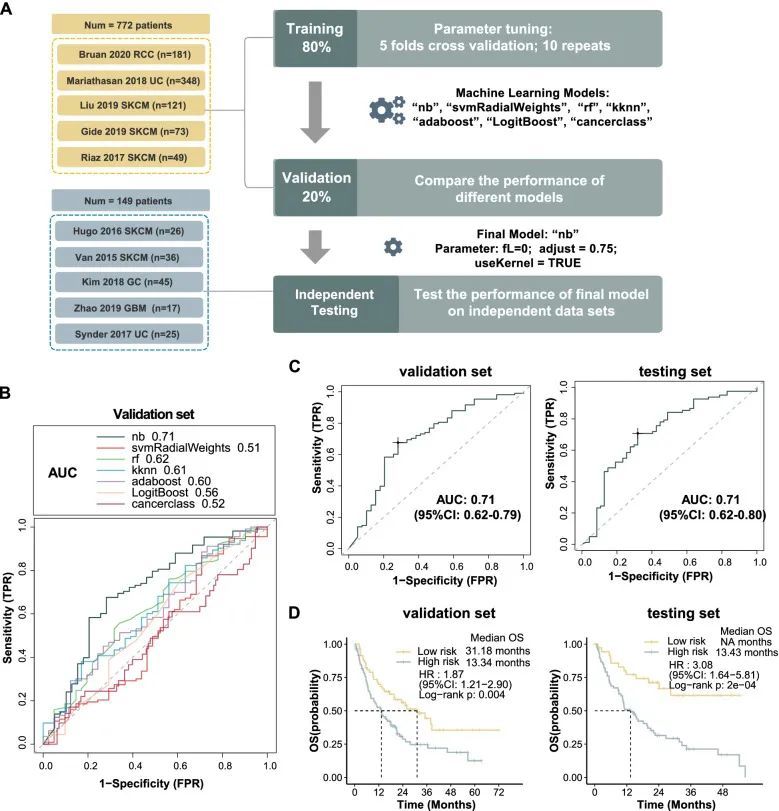
为了评估Stem.Sig模型是否能够预测总生存期,作者根据预测的“R”和“NR”将接受ICI治疗的患者分为低风险和高风险亚组。图4D显示了OS的Kaplan-Meier分析结果。在训练、验证和测试集中,低风险组的总生存期明显较长(所有log-rank p < 0.01)。在验证队列中,由Stem.Sig模型预测为高风险患者的中位OS仅为13.3个月,而低风险患者为31.2个月(HR:1.87;95%CI:1.21-2.90)。在测试集中,高风险患者观察到类似的中位OS为13.4个月,而低风险患者尚未达到中位OS(HR:3.08;95%CI:1.64-5.81)。
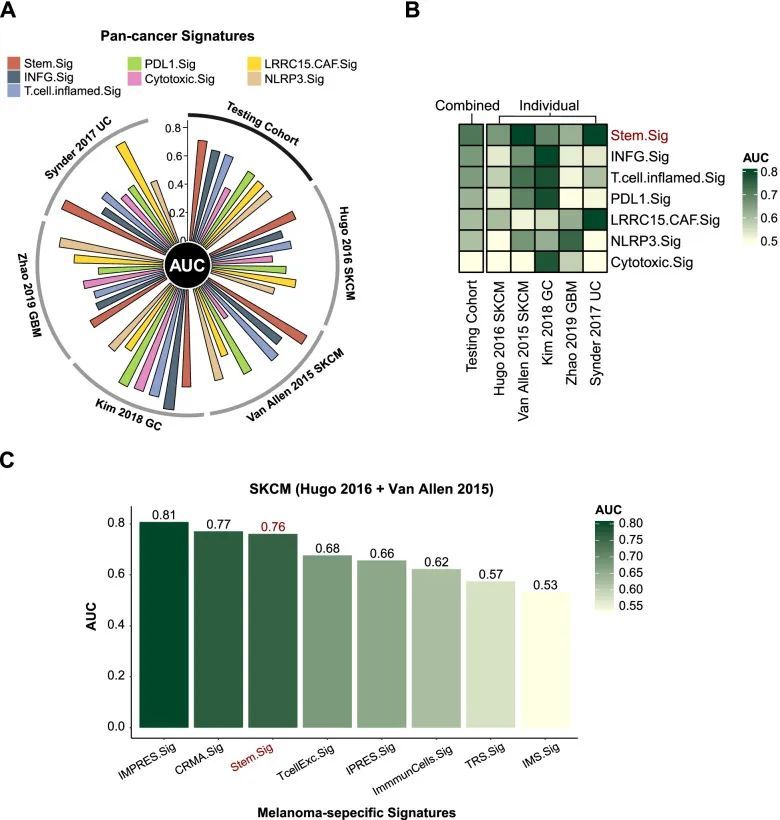
作者对贡献于测试集的五个个体队列进行了亚组分析。关于ICI反应预测,这些队列的AUC范围在0.62至0.81之间(附加文件2:图S2A)。Van Allen 2015 SKCM取得了0.81的较好AUC(95%CI:0.66?0.95),其次是Synder 2017 UC(AUC:0.80;95%CI:0.61?0.99)。与其他队列相比,Zhao 2019 GBM的AUC最低,为0.62(95%CI:0.33?0.91)。在生存分析中,由于缺乏OS数据,Kim 2018 GC被排除在外。对于其他四个队列,作者观察到Stem.Sig模型预测的高风险患者的HR在1.73至4.05之间(附加文件2:图S2B)。在调整了可用混杂因素后,仍然发现Van Allen 2015 SKCM(调整p = 0.02)和Synder 2017 UC(调整p = 0.02)具有显著的生存益处,而其他两个队列只显示数值上的生存差异。这可能是由于样本量有限。
作者进一步比较了Stem.Sig与先前已建立的预测基因标记的性能。与全癌症标记(INFG.Sig [76],T.cell.inflamed.Sig [76],PDL1.Sig [77],LRRC15.CAF.Sig [78],NLRP3.Sig [79]和Cytotoxic.Sig [80])相比,Stem.Sig在测试集中表现最佳,AUC为0.71,其次是INFG.Sig,AUC为0.66(图5A)。大多数全癌症标记只在一个或两个队列中表现理想。例如,INFG.Sig在Kim 2018 GC中的AUC达到0.85,在Van Allen 2015 SKCM中为0.67,但在其他三个队列中降至0.53-0.54(附加文件1:表S7)。然而,Stem.Sig在所有队列中都取得了足够好的表现,涵盖了四种癌症类型:SKCM,GBM,UC和GC,进一步强调了它作为全癌症ICI反应预测模型的潜力(图5B)。与黑色素瘤特异性标记(CRMA.Sig [81],IMPRES.Sig [7],IPRES.Sig [82],TcellExc.Sig [14],ImmmunCells.Sig [83],IMS.Sig [84]和TRS.Sig [85])相比,Stem.Sig在预测黑色素瘤患者的ICI反应方面仍然位居前三,AUC为0.76。IMPRES.Sig和CRMA.Sig的AUC值略高于Stem.Sig,分别为0.81和0.77。
利用CRISPR筛选数据从Stem.Sig中探索潜在的治疗靶点
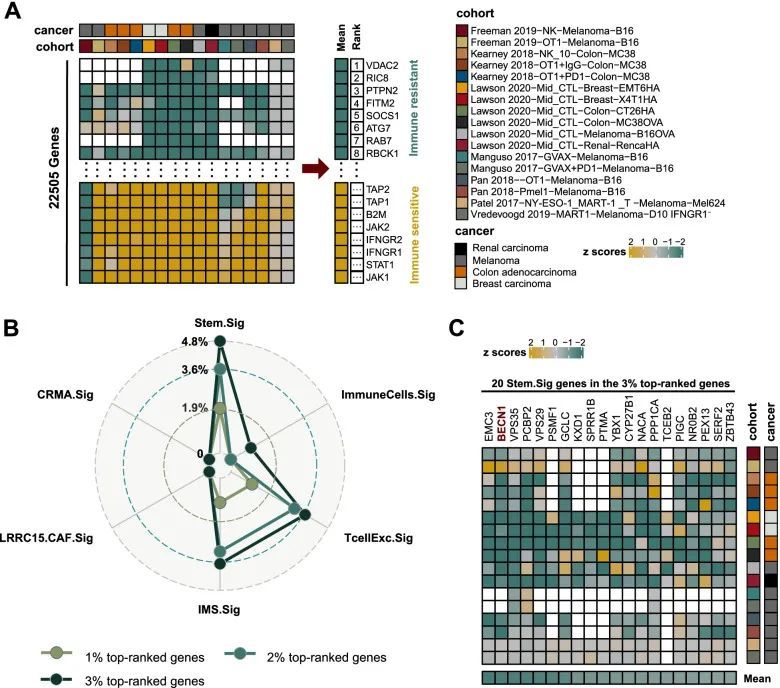
作者系统地收集了来自七个CRISPR队列的敲除基因的免疫反应数据,根据这些CRISPR队列中使用的模型细胞和处理条件,将其进一步分为17个数据集。总共有22,505个基因被这些CRISPR数据集记录。作者根据它们的平均z分数对基因进行了排名。排名靠前的基因是免疫抵抗基因,在敲除后可能促进抗肿瘤免疫。排名靠后的基因是免疫敏感基因,在敲除后可能抑制抗肿瘤免疫。基因排名的过程如图6A所示。在所有的22,505个基因中,排名前1%、2%和3%的基因数量分别为225、450和675。接下来,作者计算了在Stem.Sig和之前的免疫抵抗标志中(包括TcellExc.Sig、ImmuneCells.Sig、IMS.Sig、LRRC15.CAF.Sig和CRMA.Sig,不包括IPRES.Sig,它包含73个基因通路而不是单个基因)中出现在排名前的基因的百分比[14, 78, 81, 83, 84]。Stem.Sig、TcellExc.Sig、IMS.Sig和ImmuneCells.Sig是唯一四个在前3%中有基因排名的基因集。正如预期的那样,Stem.Sig在其他标记中具有最高比例的高排名基因(图6B)。免疫抵抗基因(3%的高排名基因)在Stem.Sig中显著过表达(P=0.03;Fisher's exact test)。Stem.Sig中有20个基因在前3%中排名,包括EMC3、BECN1、VPS35、PCBP2、VPS29、PSMF1、GCLC、KXD1、SPRR1B、PTMA、YBX1、CYP27B1、NACA、PPP1CA、TCEB2、PIGC、NR0B2、PEX13、SERF2和ZBTB43。这些干细胞相关基因的免疫抵抗特征已经通过多个独立的CRISPR数据集进行验证(图6C),可能与ICB协同作用,成为潜在的治疗靶点。
总结
作者提供了第一个坚实的临床证据,证明了癌症干细胞性与免疫疗法抗药性相关。通过对单细胞转录组数据进行全癌症分析,作者开发了一个基因表达标志,Stem.Sig,在预测多个队列中的免疫检查点抗体治疗结果方面超过其他已建立的标志。对Stem.Sig的进一步探索还揭示了一些潜在的治疗靶点。作者的研究展示了免疫疗法患者选择的有希望解决方案,并通过针对癌症干细胞性来增强抗肿瘤免疫力,为解决免疫检查点抗体治疗抗药性提供了启示。对这篇文章的思路感兴趣的老师,欢迎咨询!
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:chenni525@qq.com进行投诉反馈,一经查实,立即删除!