scVI与MultiVI
scVI:https://docs.scvi-tools.org/en/stable/user_guide/models/scvi.html
MultiVI:https://docs.scvi-tools.org/en/stable/user_guide/models/multivi.html
scVI
single cell variational inference提出了一个灵活的scRNA-seq数据生成模型。scVI功能全面,可以扩展到大规模数据集(处理超过1万个细胞),局限性在于:1.需要GPU才能快速推理,2.与线性方法不同,潜在空间不可解释。
生成
scVI将包含细胞和基因的scRNA-seq基因表达矩阵 X X X作为输入( N N N个细胞, G G G个基因),此外,一个设计的矩阵 S S S包含 p p p个观测协变量,比如day,donor等等,这是可选的输入。虽然 S S S可以包含离散协变量和连续协变量,但在下文中,我们假设它仅包含一个具有 K K K个类别的离散协变量,这代表了具有多批次数据的常见情况。
对于细胞 n n n和基因 g g g,scVI用下面过程生成观测的UMI计数 x n g x_{ng} xng?(其中, s n s_{n} sn?为细胞 n n n的batch id): z n ~ N o r m a l ( 0 , I ) l n ~ l o g N o r m a l ( l μ ? s n , l σ 2 ? s n ) ρ n = f w ( z n , s n ) π n g = f h g ( z n , s n ) x n g ~ O b s e r v a t i o n M o d e l ( l n ρ n , θ g , π n g ) z_{n}\sim Normal(0,I)\\ l_{n}\sim logNormal(l_{\mu}^{\top}s_{n},l_{\sigma^{2}}^{\top}s_{n})\\ \rho_{n}=f_{w}(z_{n},s_{n})\\ \pi_{ng}=f_{h}^{g}(z_{n},s_{n})\\ x_{ng}\sim ObservationModel(l_{n}\rho_{n},\theta_{g},\pi_{ng}) zn?~Normal(0,I)ln?~logNormal(lμ??sn?,lσ2??sn?)ρn?=fw?(zn?,sn?)πng?=fhg?(zn?,sn?)xng?~ObservationModel(ln?ρn?,θg?,πng?)简而言之,每个基因的基因表达取决于细胞特异性的潜在变量 z n z_{n} zn?。先验参数 l μ l_{\mu} lμ? 和 l σ 2 l_{\sigma^{2}} lσ2? 是根据所有细胞的对数大小在不同批次之间计算得出的均值和方差。表达数据是从一个基于计数的似然分布生成的,这里称为 O b s e r v a t i o n M o d e l ObservationModel ObservationModel,默认为ZINB分布,也可以是更简单的NB分布。
生成过程使用两个神经网络: f w ( z n , s n ) : R d × { 0 , 1 } K → Δ G ? 1 f h ( z n , s n ) : R d × { 0 , 1 } K → ( 0 , 1 ) ? f_{w}(z_{n},s_{n}):\mathbb{R}^{d}\times\left\{0,1\right\}^{K}\rightarrow\Delta^{G-1}\\f_{h}(z_{n},s_{n}):\mathbb{R}^{d}\times\left\{0,1\right\}^{K}\rightarrow(0,1)^{\top} fw?(zn?,sn?):Rd×{0,1}K→ΔG?1fh?(zn?,sn?):Rd×{0,1}K→(0,1)?分别解码去噪基因表达和ZINB概率。
下表为潜在变量以及描述:
| 潜在变量 | 描述 |
|---|---|
| z n ∈ R d z_{n}\in\mathbb{R}^{d} zn?∈Rd | 细胞状态的低维表示 |
| ρ n ∈ Δ G ? 1 \rho_{n}\in\Delta^{G-1} ρn?∈ΔG?1 | 去噪的基因表达。这是一个cell内总和为1的向量 |
| l n ∈ ( 0 , ∞ ) l_{n}\in(0,∞) ln?∈(0,∞) | RNA 文库大小。 这里它被建模为一个潜在变量,但 scVI 最近的默认设置是将文库大小视为观察到的,等于当前细胞 n n n的总 RNA UMI 计数 |
| θ g ∈ ( 0 , ∞ ) \theta_{g}\in(0,∞) θg?∈(0,∞) | NB的Inverse dispersion。例如,可以通过在模型初始化期间传递dispersion=“gene-batch”来将其设置为特定于基因或批次 |
推理
scVI 使用变分推理来学习模型参数(神经网络参数、Inverse dispersion等)和具有以下分解的近似后验分布: q η ( z n , l n ∣ x n ) = q η ( z n ∣ x n , s n ) q η ( l n ∣ x n ) q_{\eta}(z_{n},l_{n}|x_{n})=q_{\eta}(z_{n}|x_{n},s_{n})q_{\eta}(l_{n}|x_{n}) qη?(zn?,ln?∣xn?)=qη?(zn?∣xn?,sn?)qη?(ln?∣xn?)这里 η \eta η是对应于推理神经网络(编码器)的一组参数。值得注意的是,默认情况下,scVI 仅接收表达数据作为输入(没有观测到的细胞水平协变量)。根据经验,通过让编码器将这些item的串联 q η ( z n , l n ∣ x n , s n ) q_{\eta}(z_{n},l_{n}|x_{n},s_{n}) qη?(zn?,ln?∣xn?,sn?) 作为输入,我们并没有看到太大的差异。
任务
下面是scVI可以执行的任务。
降维:近似的后验 q η ( z n ∣ x n , s n ) q_{\eta}(z_{n}|x_{n},s_{n}) qη?(zn?∣xn?,sn?)被返回获得低维表示。
transfer :scVI可以根据参考数据进行训练,然后将细胞水平协变量(比如细胞类型注释)转移到查询数据。
基因表达的插补:scVI返回近似后验下 ρ n \rho_{n} ρn?的期望值。对于细胞 n n n,可以写成: E q η ( z n ∣ x n ) [ f w ( z n , s n ) ] \mathbb{E}_{q_{\eta}(z_{n}|x_{n})}[f_{w}(z_{n},s_{n})] Eqη?(zn?∣xn?)?[fw?(zn?,sn?)]
差异分析:scVI在估计后的插补数据上执行差异分析。
生成模拟数据:可以从 z n z_{n} zn?进行采样,重新模拟生成新的细胞数据。
MultiVI
multiVI是一个多模态生成模型,能够整合多组学的scRNA-seq和scATAC-seq数据,训练后,可用于许多常见的下游任务,也可用于缺失模态的插补。
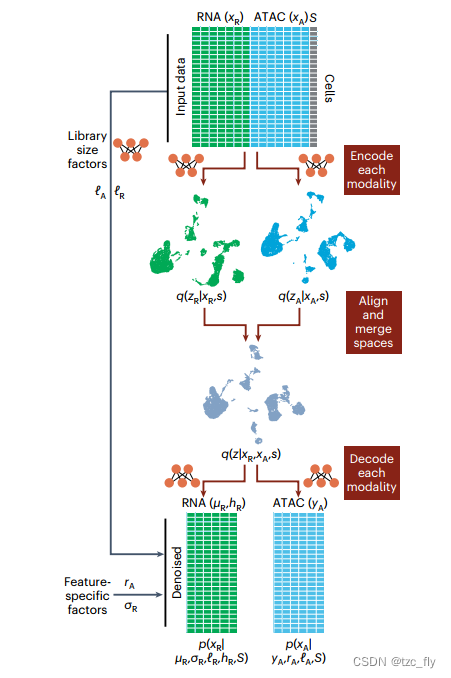
- 图1:其中输入数据由ATAC、RNA两种数据类型组成(或者加上蛋白质模态)。变量 S S S表示实验协变量,如批次或实验条件( S S S是可选的输入)。每个数据模态被编码成模态无关的潜在表示,然后,这些表示被合并到一个联合潜在空间。
生成
MultiVI的输入为多模态单细胞矩阵 X m u l t X_{mult} Xmult?,包含 N N N个细胞, G G G个基因,以及 M M M个基因区域(peaks),或者仅 X r n a X_{rna} Xrna?( N N N个细胞和 G G G个基因),或者仅 X a t a c X_{atac} Xatac?( N N N个细胞和 M M M个peaks,peaks可能是二值化的或者整数计数)。此外,一个设计的矩阵 S S S包含 p p p个观测协变量,比如day等,这是可选的输入。虽然 S S S可以包含离散协变量和连续协变量,但在下文中,我们假设它仅包含一个具有 K K K个类别的离散协变量,这代表了具有多批次数据的常见情况。
对于细胞 n n n和基因 g g g以及peaks j j j,MultiVI用下面过程生成观测的UMI计数 x n g x_{ng} xng?和 y n j y_{nj} ynj?: z n ~ N o r m a l ( 0 , I ) l n ~ l o g N o r m a l ( l μ ? s n , l σ 2 ? s n ) ρ n = f w ( z n , s n ) π n g = f h g ( z n , s n ) x n g ~ O b s e r v a t i o n M o d e l ( l n ρ n , θ g , π n g ) p n j = g z j ( z n , s n ) l n = f l ( y n ) y n j ~ B e r n o u l l i ( p n j ? l n ? r j ) z_{n}\sim Normal(0,I)\\ l_{n}\sim logNormal(l_{\mu}^{\top}s_{n},l_{\sigma^{2}}^{\top}s_{n})\\ \rho_{n}=f_{w}(z_{n},s_{n})\\ \pi_{ng}=f_{h}^{g}(z_{n},s_{n})\\ x_{ng}\sim ObservationModel(l_{n}\rho_{n},\theta_{g},\pi_{ng})\\ p_{nj}=g_{z}^{j}(z_{n},s_{n})\\ l_{n}=f_{l}(y_{n})\\ y_{nj}\sim Bernoulli(p_{nj}\cdot l_{n}\cdot r_{j}) zn?~Normal(0,I)ln?~logNormal(lμ??sn?,lσ2??sn?)ρn?=fw?(zn?,sn?)πng?=fhg?(zn?,sn?)xng?~ObservationModel(ln?ρn?,θg?,πng?)pnj?=gzj?(zn?,sn?)ln?=fl?(yn?)ynj?~Bernoulli(pnj??ln??rj?)对于scRNA-seq,先验参数 l μ l_{\mu} lμ? 和 l σ 2 l_{\sigma^{2}} lσ2? 是根据所有细胞的对数大小在不同批次之间计算得出的均值和方差。表达数据是从一个基于计数的似然分布生成的,这里称为 O b s e r v a t i o n M o d e l ObservationModel ObservationModel,默认为ZINB分布,也可以是更简单的NB分布。
对于scATAC-seq,检测可访问区域( y n j > 0 y_{nj}>0 ynj?>0)是由伯努利随机变量生成的,该变量取决于细胞特异性潜在变量 z n z_{n} zn?,该变量捕获生物异质性。另外有两个辅助的缩放因子 l n l_{n} ln?和 r j r_{j} rj?,分别考虑了特定于细胞和特定于region的批次效应。
MultiVI 的生成过程使用神经网络: f w ( z n , s n ) : R d × { 0 , 1 } K → Δ G ? 1 f h ( z n , s n ) : R d × { 0 , 1 } K → ( 0 , 1 ) ? g z ( z n , s n ) : R d × { 0 , 1 } K → [ 0 , 1 ] M f_{w}(z_{n},s_{n}):\mathbb{R}^{d}\times\left\{0,1\right\}^{K}\rightarrow\Delta^{G-1}\\f_{h}(z_{n},s_{n}):\mathbb{R}^{d}\times\left\{0,1\right\}^{K}\rightarrow(0,1)^{\top}\\ g_{z}(z_{n},s_{n}):\mathbb{R}^{d}\times\left\{0,1\right\}^{K}\rightarrow[0,1]^{M} fw?(zn?,sn?):Rd×{0,1}K→ΔG?1fh?(zn?,sn?):Rd×{0,1}K→(0,1)?gz?(zn?,sn?):Rd×{0,1}K→[0,1]M分别解码去噪基因表达、非零膨胀概率(仅当使用 ZINB 时)并估计可访问区域的概率。
推理
MultiVI 使用变分推理来学习模型参数,并得到具有以下分解的近似后验分布: q η ( z n , l n ∣ x n ) = q η ( z n ∣ x n , y n , s n ) q η ( l n ∣ x n ) q_{\eta}(z_{n},l_{n}|x_{n})=q_{\eta}(z_{n}|x_{n},y_{n},s_{n})q_{\eta}(l_{n}|x_{n}) qη?(zn?,ln?∣xn?)=qη?(zn?∣xn?,yn?,sn?)qη?(ln?∣xn?)其中, z n z_{n} zn?被确定性地计算为变分近似 z n r n a z_{n}^{rna} znrna?和 z n a t a c z_{n}^{atac} znatac?的两个潜在变量的平均值。这两个变量是正态的,因此 z n z_{n} zn?是正态的。
当获得隐变量后,使用生成模型即可完成不同模态的数据还原。
MultiVI的局限:因为 z n z_{n} zn?需要获得两个模态的 z n r n a z_{n}^{rna} znrna?和 z n a t a c z_{n}^{atac} znatac?均值,MultiVI在非联合测量数据上的整合能力有限。
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:chenni525@qq.com进行投诉反馈,一经查实,立即删除!
- Python教程
- 深入理解 MySQL 中的 HAVING 关键字和聚合函数
- Qt之QChar编码(1)
- MyBatis入门基础篇
- 用Python脚本实现FFmpeg批量转换
- [NCTF 2023] web题解
- 计算机网络 第六章(应用层)
- 基础小白快速入门Python----python的注释和变量和常量
- Kylin的工作原理及使用分享:构建大数据分析之塔
- 国内具有会员中心的wordpress主题有哪些推荐?
- Elasticsearch——快速入门
- Neo4j安装与配置以及JDK安装与配置教程(超详细)
- 旅游服务平台可视化大屏界面,UI设计PS资料
- Zuul1.x 高并发下阻塞分析以及解决方案
- limit查询报错问题