R语言【rgbif】——使用rgbif获取非行政单位区域内的物种记录信息(以泛喜马拉雅地区为例)
使用occ_search获取非行政单位区域内的物种记录信息(以泛喜马拉雅地区为例)
1. 分析使用rgbif的一些函数
根据R语言【rgbif】——用rgbif下载物种分布数据(rgbif包的初步探索),我们可以了解到要获取物种的地理分布记录的基本操作流程如下:

1.1 使用name_suggest()的一些要点
- name_suggest有一个必填参数【q】,接受的输入值是全部或部分学名**。
- name_suggest的参数【rank】可以获得指定分类阶元等级的结果。
- name_suggest的参数【limit】已经失效,最大值即默认值(100条),当实际检索结果多于100条时需要配合参数【start】使用。
- name_suggest的参数【fields】默认包含字段key,可以满足函数【occ_count()】的使用。
通过name_suggest()**获取物种对应的标识符。
1.2 使用occ_count()的一些要点
- occ_count的底层函数是occ_search,绝大部分参数与之也相同。
- occ_count特有的两个参数有参数【occurrenceStatus】和参数【curlopts】。其中参数【occurrenceStatus】默认为PRESENT,而参数【curlopts】与GBIF操作无关紧要。
- 在使用occ_search实际获取数据前,occ_count可以查看实际满足条件的结果数量,以避免将来被参数【limit】限制返回数量。
在此文中,应当以泛喜马拉雅地区和物种标识符为条件,通过occ_count查看此区域内该物种的记录数量。
1.3 使用occ_search()的一些要点
- 主要通过传入物种标识符/物种学名以及泛喜马拉雅地区区域,在考虑到实际的记录数量是否超出参数【limit】的上限(100000)的情况下,获得所有的物种记录信息。
2. 数据的准备工作和检验
以泛喜马拉雅地区为例,要获取该区域内所有被子植物的地理分布记录,起码需要解决两个问题。第一,泛喜马拉雅地区的WKT格式的字符串;第二,泛喜马拉雅地区的被子植物名录。
2.1 空间表达(用WKT表示该地区)
2.1.1 非行政区域的边界坐标点提取
泛喜马拉雅地区并不是一个标准的行政区域,它是由喜马拉雅山脉和邻近地区形成的一个天然的植物地理单元。该地区包括阿富汗东北角、巴基斯坦北部、印度北部、尼泊尔、不丹、缅甸北部和中国西南部(西藏南部、青海东南部、甘肃东南部、四川西部和云南西北部)。所以,不可能通过行政区域的组合来完成对泛喜马拉雅地区的空间描述。
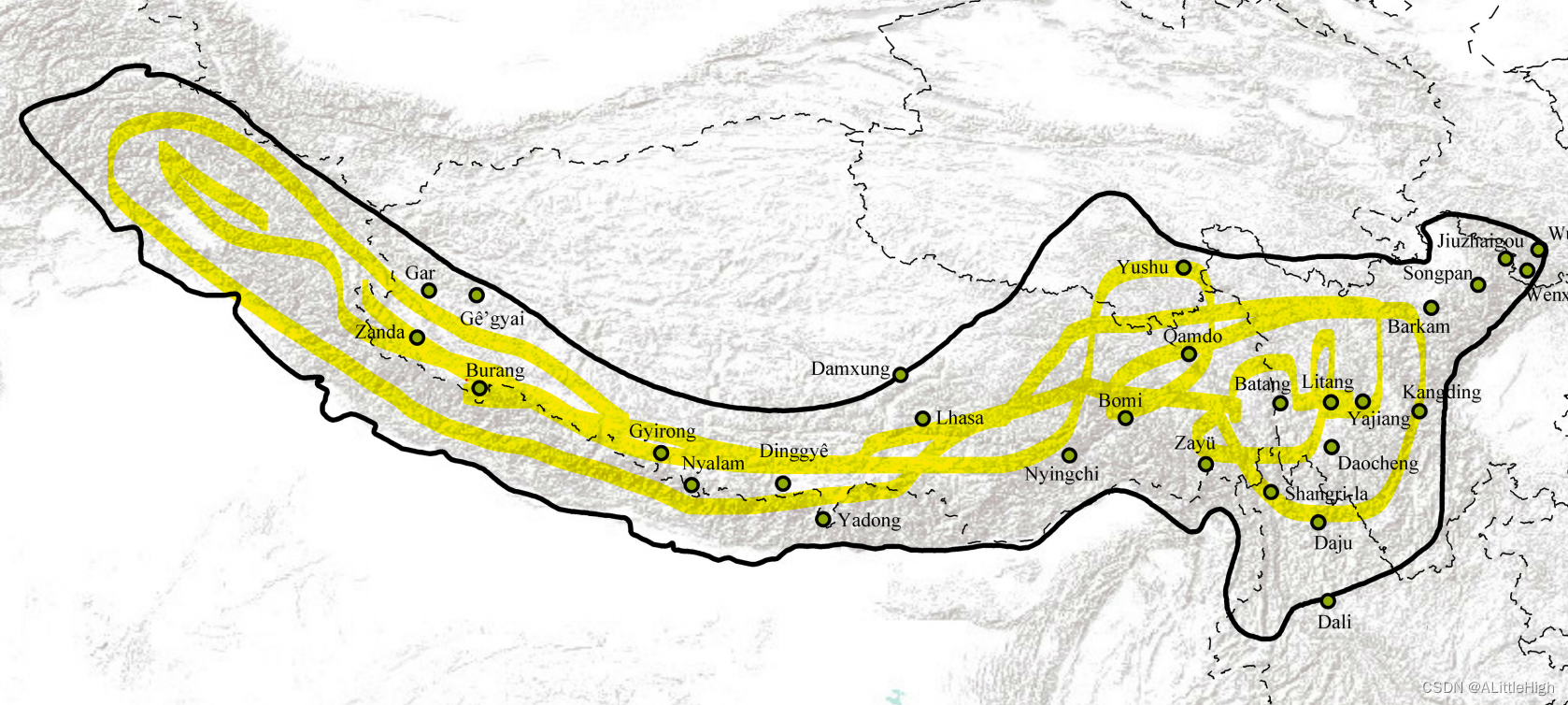
从文献中获取泛喜马拉雅地区的地理图,上图中显示了该地区的范围和边界,只需要将这些特征提取出来即可:
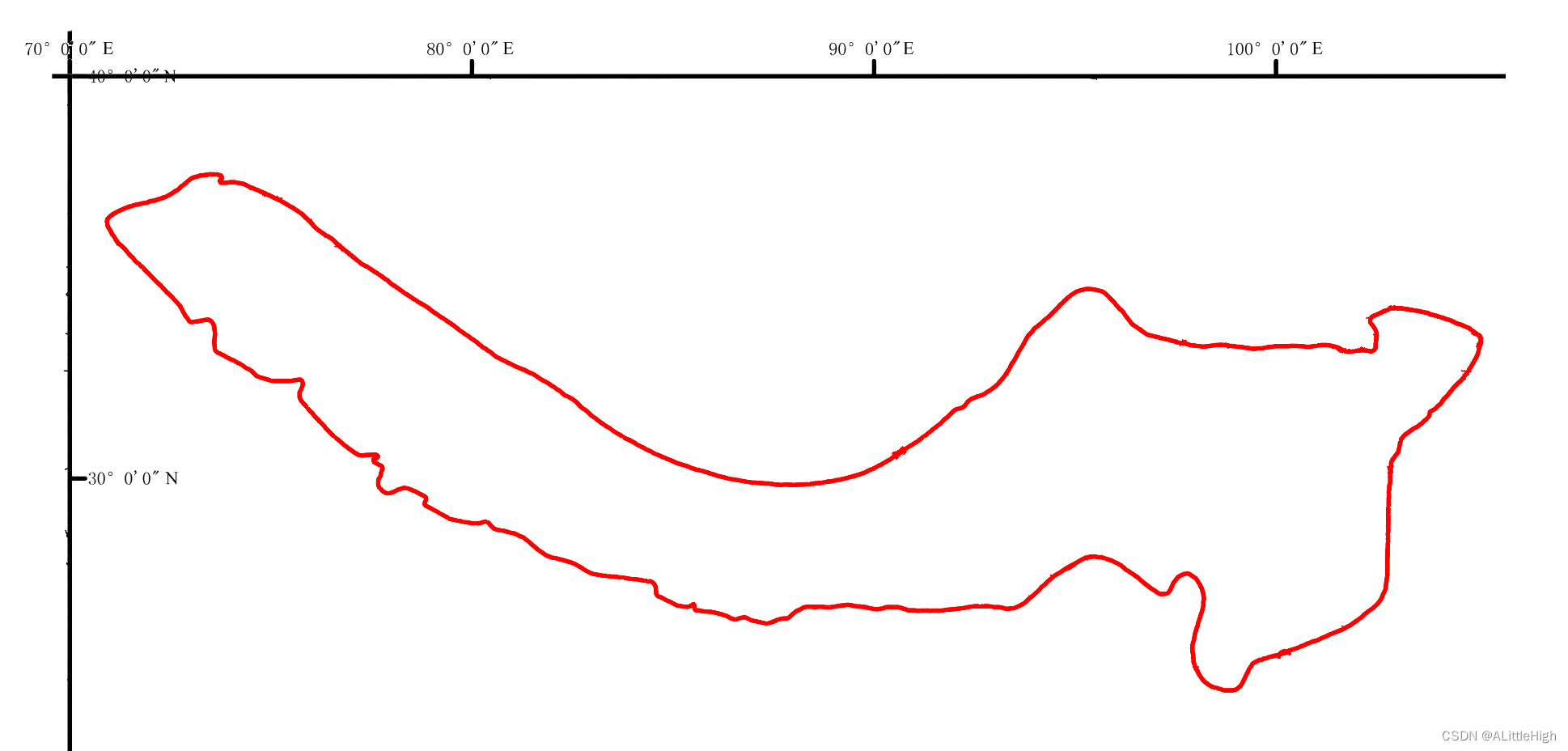
提取后的图片信息十分简洁,便于导入getdata软件中提取坐标信息。
在getdata软件中,按照 设置坐标系——沿线取点——手动修正——点位排序,即可获得表达泛喜马拉雅地区的边界坐标信息。
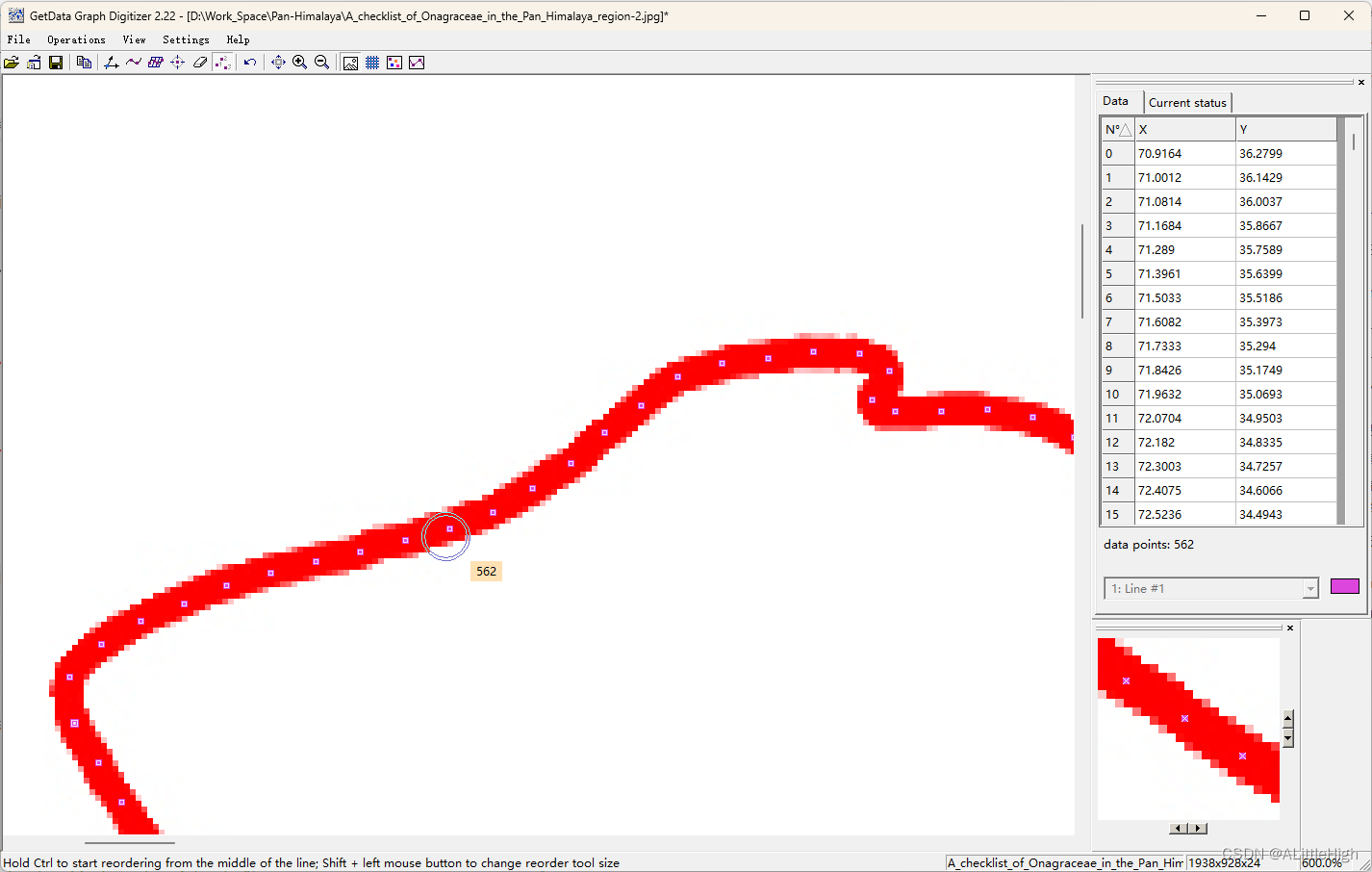
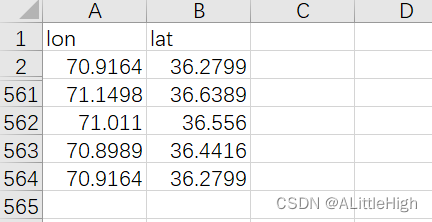
将右侧数据框中的数据选中后,手动复制保存到pan-himalay-poly-points.xlsx文件中。
注意,我将将点1复制到末尾,这样就可以构建一个封闭的polygon。
2.2.2 非行政区域的边界坐标点检验
第一步,将保存坐标的pan-himalay-poly-points.xlsx导入R中,将它们显示在世界地图上
library(readxl)
library(ggplot2)
pan_himalay_poly_points <- read_excel(
"D:/Work_Space/Pan-Himalaya/pan-himalay-poly-points.xlsx")
world <- map_data("world")
ggplot() +
geom_map(data = world, map = world,
aes(long, lat, map_id = region),
color = "black",
fill = "white",
size = 0.1) +
geom_point(data = pan_himalay_poly_points,
aes(lon, lat),
color = "red") +
geom_polygon(data = pan_himalay_poly_points,
aes(lon, lat),
color = "green",
fill = "orange")
哇哦,形状完美,看来坐标点提取的很成功。
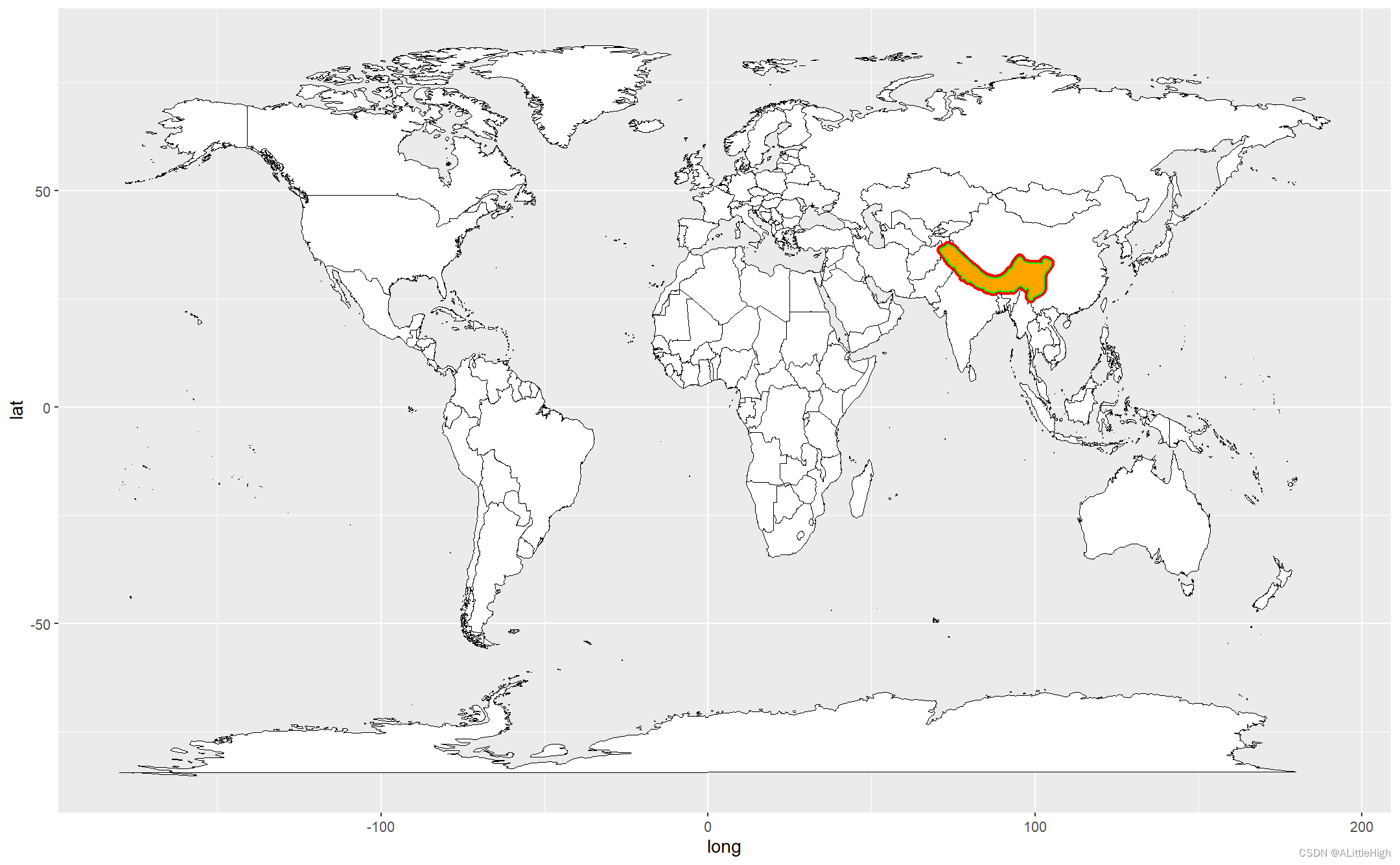
第二步,将坐标数据转换为WKT格式的polygon字符串:
wkt <- NULL
for (i in 1:nrow(pan_himalay_poly_points)){
wkt <- append(wkt, paste(pan_himalay_poly_points[i,]$lon,
pan_himalay_poly_points[i,]$lat)
)
}
wkt <- paste(unlist(wkt), collapse = ',')
wkt <- paste0("POLYGON((", wkt, "))")
wkt <- gsub("\n", " ", wkt)
第三步,检查polygon字符串的长度,如果长度 >1500,说明它是rgbif中定义的长WKT字符串,要进行分割处理
这一步的代码需要用到以下包的方法,需要导入这些包:
library(rgbif)
library(randomcoloR)
library(rgeos)
这一步要完成的处理比较多,可以分为几个小步骤,这样思路可以更加清楚:
-
判断长度
if (isTRUE(nchar(wkt) > 1500)){ wkt_for_rgbif <- NULL print("takes apart the long wkt string") } else { wkt_for_rgbif <- wkt } -
确认polygon字符串是长wkt时,可以先用rgbif自带的check_wkt()进行检查
if (isTRUE(nchar(wkt) > 1500)){ wkt_for_rgbif <- NULL print("takes apart the long wkt string") check_wkt(wkt) } else { wkt_for_rgbif <- wkt }如果字符串没问题,就会原样输出,否则会在控制台输出报错信息。
-
检查无恙后,使用rgbif的wkt_parse()对wkt进行分割,调整参数直至分割后的wkt片段均 <1500,wkt_parse的【geom_big】参数必须为 axe,【geom_size】参数的值需要调整
if (isTRUE(nchar(wkt) > 1500)){ wkt_for_rgbif <- NULL print("takes apart the long wkt string") check_wkt(wkt) wkt_for_rgbif <- wkt_parse(wkt, geom_big = "axe", geom_size = 5) if (all(nchar(wkt_for_rgbif) < 1500)){ print("long wkt string already taken apart into pecies whose length less than 1500") } else { print("some species of long wkt string still longer than 1500") } } else { print("this is not a long wkt string") wkt_for_rgbif <- wkt }
第三步运行成功时,您应该在控制台看到如下两行输出:
[1] "takes apart the long wkt string"
[1] "long wkt string already taken apart into pecies whose length less than 1500"
第四步:查看一下分割后的wkt片段是什么样子
这里我自定义了一个 map_apart_wkt 函数,向它传递分割后的wkt片段,即可在地图上展示:
map_apart_wkt <- function(apart_wkt){
check_map <- ggplot() +
geom_map(data = world,
map = world,
aes(long, lat, map_id = region),
color = "black",
fill = "white",
size = 0.1)
palette <- distinctColorPalette(length(apart_wkt))
for (i in 1:length(apart_wkt)){
read_apart <- readWKT(apart_wkt[i])
coords_apart <- as.data.frame(coordinates(read_apart@polygons[[1]]@Polygons[[1]]))
check_map <- check_map +
geom_polygon(data = coords_apart,
aes(x, y),
color = "black",
fill = palette[i])
}
check_map
}
map_apart_wkt(apart_wkt = wkt_for_rgbif)
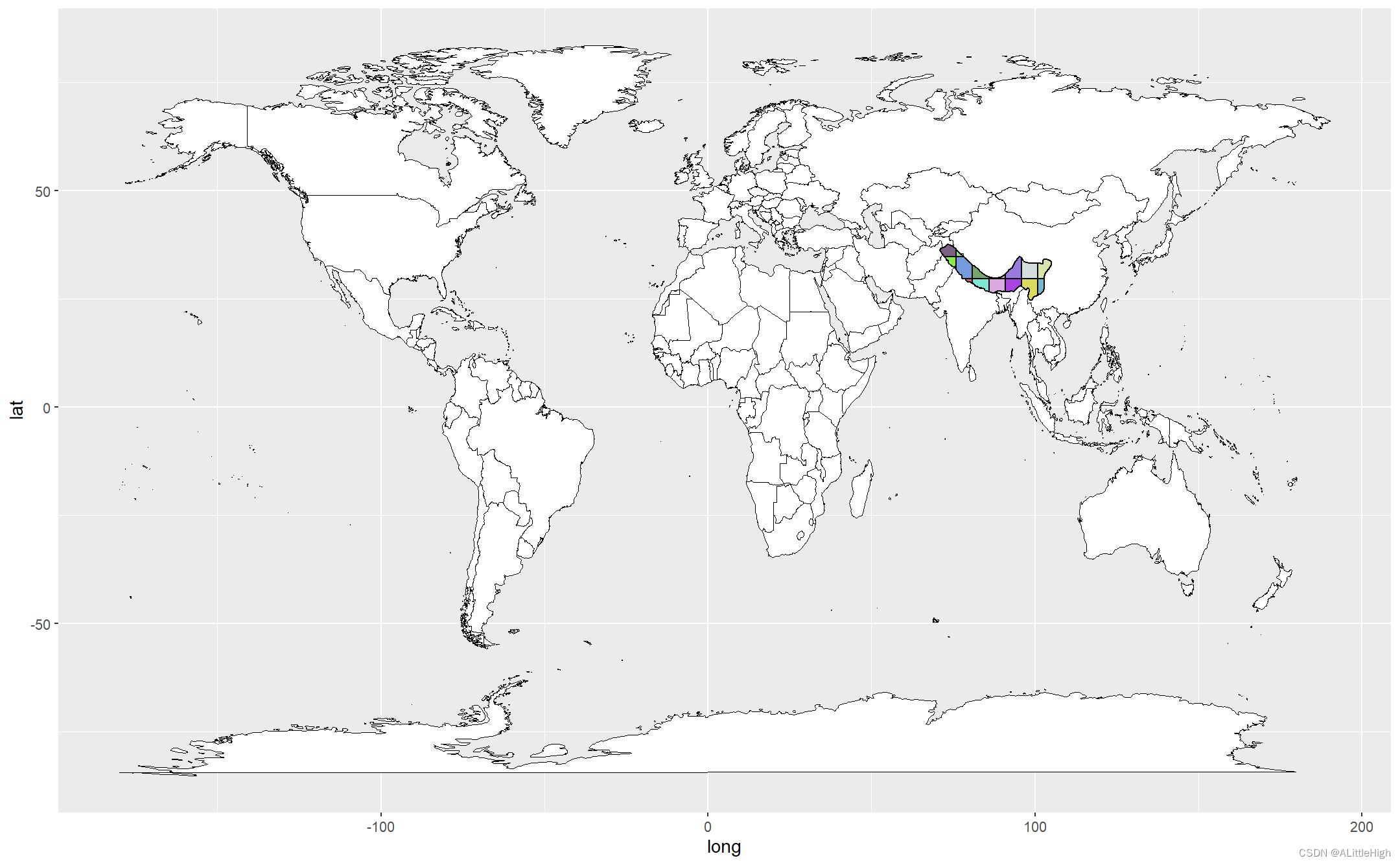
第五步:必须检测一下parse_wkt()的结果对于occ_search()是否有用
test_wkt <- occ_search(limit = 0,
hasCoordinate = TRUE,
geometry = wkt_for_rgbif)
结果会返回一个相当于分割后的wkt数量:
有关这部分的详细讨论,请阅读R语言【rgbif】——occ_search对待字符长度大于1500的WKT的特殊处理真的有必要吗?
2.2 物种列表(物种名称目录和名称校对)
那么下一个问题,获取该地区的哪些植物记录?
2.2.1 物种名录获取
我这里使用了目前泛喜马拉雅植物志在线网站中提供的名录,目前数字化的植物物种约有6000种。
pan_himalay_plant <- read_excel(
"D:/Work_Space/Pan-Himalaya/pan-himalay-plant.xlsx")
glimpse(pan_himalay_plant)
ln <- unique(pan_himalay_plant$LevelName)
sp <- c("Species", "Subspecies", "Variety", "SubSpecies")
sp_plants <- pan_himalay_plant %>% filter(LevelName %in% sp)
2.2.2 物种名称校对
并且,可以进一步适用rWCVP对这些物种名称进行验证
matches <- wcvp_match_names(sp_plants,
name_col = "LName",
fuzzy = TRUE,
progress_bar = FALSE)
具体操作过程参考R语言实践——使用rWCVP在WCVP中匹配名称。
或者使用 U.Taxonstand进行名称标准化检验
这里我从检验过后的数据中随机抽取50个物种进行后续操作。
[1] "Lepisorus mucronatus" "Rhododendron trichostomum" "Pedicularis davidii"
[4] "Rhododendron wardii" "Panax japonicus" "Saxifraga stenophylla"
[7] "Nannoglottis gynura" "Plantago depressa" "Taxillus caloreas"
[10] "Elsholtzia strobilifera" "Pteris vittata" "Sinosenecio euosmus"
[13] "Sinocrassula densirosulata" "Colquhounia vestita" "Potentilla simulatrix"
[16] "Orobanche coerulescens" "Lepisorus thunbergianus" "Globba multiflora"
[19] "Actinidia arguta" "Adenia trilobata" "Rhus punjabensis"
[22] "Erigeron breviscapus" "Silene adenocalyx" "Strobilanthes henryi"
[25] "Microula floribunda" "Silene monbeigii" "Conioselinum acuminatum"
[28] "Croton yunnanensis" "Amomum tibeticum" "Ophioglossum vulgatum"
[31] "Rubus calophyllus" "Primula tenuiloba" "Schisandra neglecta"
[34] "Cotoneaster adpressus" "Hydrangea bretschneideri" "Platycodon grandiflorus"
[37] "Colebrookea oppositifolia" "Dryopteris fuscipes" "Cotinus nanus"
[40] "Saussurea coriolepis" "Lindera neesiana" "Roscoea forrestii"
[43] "Androsace erecta" "Paraprenanthes triflora" "Pteridium revolutum"
[46] "Taraxacum eriopodum" "Alternanthera pungens" "Adenophora polyantha"
[49] "Impatiens tuberculata" "Luculia pinceana"
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:chenni525@qq.com进行投诉反馈,一经查实,立即删除!
- Python教程
- 深入理解 MySQL 中的 HAVING 关键字和聚合函数
- Qt之QChar编码(1)
- MyBatis入门基础篇
- 用Python脚本实现FFmpeg批量转换
- 仓储出入库及库存管理系统哪个好
- 如何找到最适合的数据恢复软件?10 款最佳数据恢复软件榜单
- Yi-34B Tokenizer 分析
- SOLIDWORKS编码重命名批量完成原来这么简单
- 详解DETR (End-to-End Object Detection with Transformers)
- springboot113健身房管理系统
- 初学unity学习七天,经验收获总结
- 塔夫特原则
- C++图论之强连通图
- three.js : tweenjs创建threejs动画