gephi里做多个图,保证图例相同颜色表示相同种类
首先假定你对R语言生成.graphml文件和Gephi的使用是熟悉的
然后
在做共现网络分析的时候会在R语言里跑出来.graphml文件,包含node和edge属性,按理说之后导入Gephi,按照Phylum(微生物的门水平)对node进行着色就行了,但是如果跑多个图的话,就会发现每次不同的Phylum对应的颜色都不一样
因此思路就是,给每个Phylum对应一个color属性,让它根据color属性进行着色。
好好好,开始逆推
假设我们已经有了color属性(当然,这里是我为了尝试,自己从Gephi中导出node文件,然后手动加的颜色)
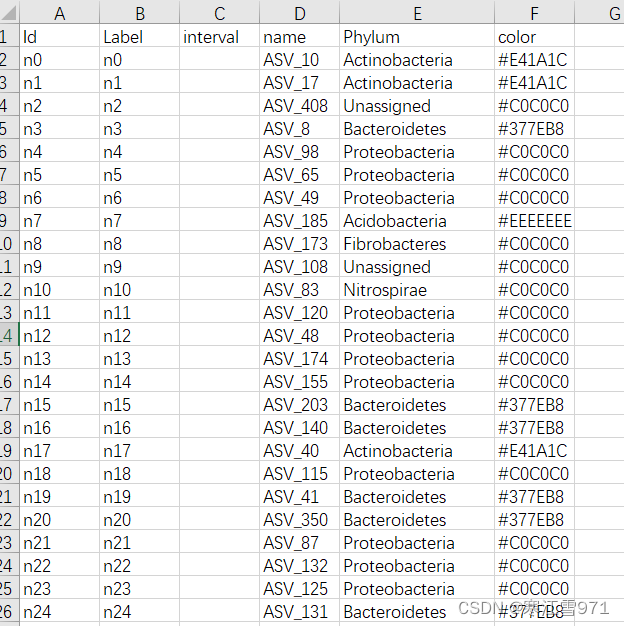
之后查了一下,要让这个属性起作用,需要在Gephi中安装一个插件,叫做give colors to nodes。具体怎么安装呢?看这里
①先去官网下载https://gephi.org/plugins/#/
②安装到自己的Gephi中:工具》》插件》》已下载》》添加插件
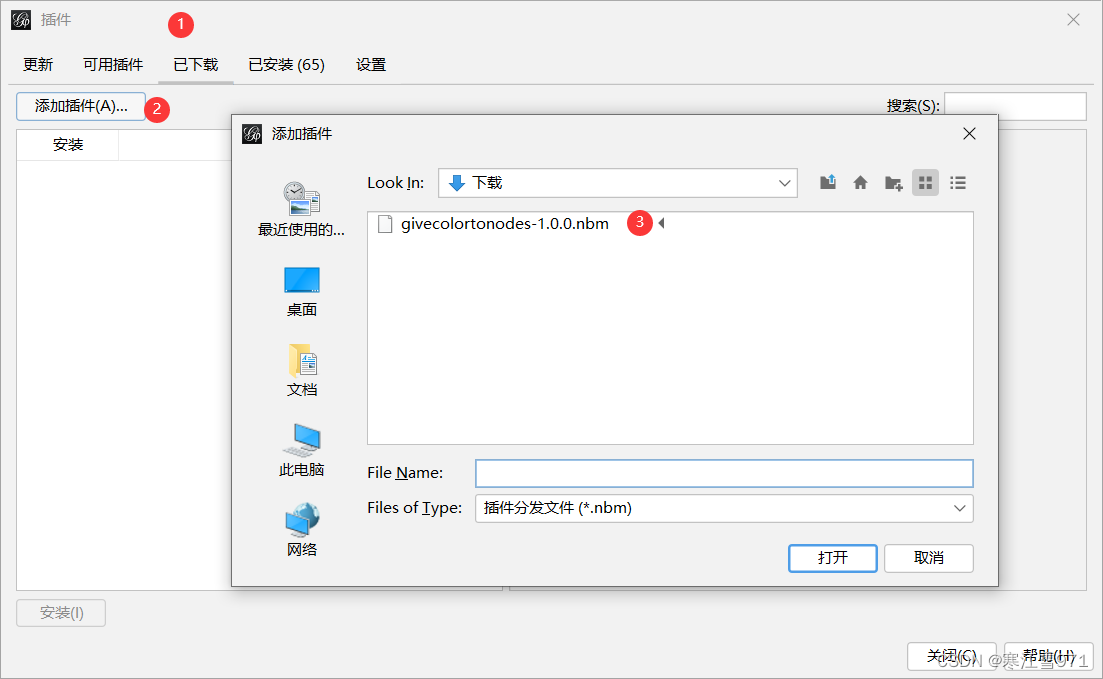
之后就是把添加了color属性的node表重新导入,点Give colors to nodes插件的图标就行啦
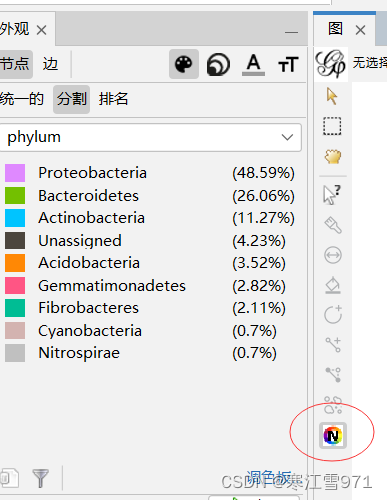
点了之后看到这个提示的一刻还是蛮激动的,证实了这个思路可行。接着就是在R语言里修改代码,让每个Phylum都能自动对应一种颜色。?

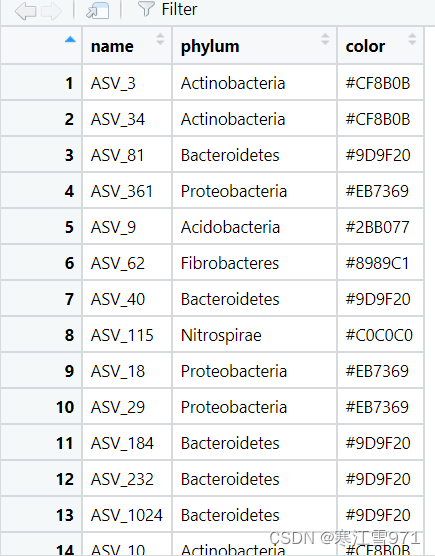
这是我的node文件,也就是说,我想给它添加一列color,并且按照Phylum的内容自动为它填充内容?

?
好好好,接着查,自然而然地就想到了if语句,就查着慢慢修改。
node$color=NA ?#添加新列
node$color <- ifelse(node$phylum=="Proteobacteria", "#EB7369",?
? ? ? ? ? ? ? ? ? ? ?ifelse(node$phylum=="Actinobacteria", "#CF8B0B",
? ? ? ? ? ? ? ? ? ? ? ? ? ? ifelse(node$phylum=="Bacteroidetes","#9D9F20",
? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ?ifelse(node$phylum=="Acidobacteria","#2BB077",
? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ifelse(node$phylum=="Gemmatimonadetes","#1BB3B7",
? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ?ifelse(node$phylum=="Verrucomicrobia","#29A4DE",
? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ifelse(node$phylum=="Fibrobacteres","#8989C1","#C0C0C0")))))))
这个代码看着复杂,但是思路很简洁,就是ifelse(条件,color列填充什么内容,不然color列填充什么内容),一直在括号里套ifelse语句就行了,这里Phylum和颜色我是随便选择的,根据自己需要再做修改就行了。
 ?
?
然后以下代码就是做.graphml文件,再导入Gephi就万事大吉啦。
igraph ?= igraph::graph_from_data_frame(edge, directed = FALSE,?
? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? vertices = node)#vertex节点? ?
write.graph(igraph, '22.graphml', format = 'graphml')
不过,不过,我发现,切换到“数据资料”这一栏的时候没有color这一列,但是这时候图就已经是自己设置的颜色了
略作为学习的小记,希望能帮到大家,也欢迎多多交流
? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ??
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:chenni525@qq.com进行投诉反馈,一经查实,立即删除!
- Python教程
- 深入理解 MySQL 中的 HAVING 关键字和聚合函数
- Qt之QChar编码(1)
- MyBatis入门基础篇
- 用Python脚本实现FFmpeg批量转换
- vue/uniapp - 返回上一页并onLoad/onShow刷新数据列表接口
- 【无标题】
- vue2 如何配置路由详解。
- 第一天使用seleninum登录系统
- Spring高手之路-Spring事务的传播机制(行为、特性)
- 网页设计(六)表格与表格页面布局
- 四款坚固耐用、小尺寸、1EDB9275F、1EDS5663H、1EDN9550B、1EDN7512G单通道栅极驱动器IC
- 湖南大学-数据库系统-2018期末考试解析
- 【LeetCode: 295. 数据流的中位数 + 堆】
- 7个理由:从Java8升级到Java17