大数据HCIE成神之路之数据预处理(2)——异常值处理
1 异常值处理
1.1 散点图
将数据用散点图的形式可视化呈现,以便观察到数据集中的异常值。
Python中 matplotlib.pyplot 库的 scatter 函数用于数据集的可视化输出。其基本格式如下:
scatter(x, y, s=None, c=None, marker=None, cmap=None, norm=None, vmin=None, vmax=None, alpha=None, linewidths=None, verts=None, edgecolors=None, hold=None, data=None, **kwargs)
关键参数详解:
-
X代表散点图的x轴数据。
-
y代表散点图的y轴数据。
-
s可选参数,代表散点图的数据
大小。 -
c可选参数,代表散点图的数据
颜色,默认为蓝色。 -
marker可选参数,代表散点图的
形状,默认为○型。 -
camp可选参数,代表指定
色图,默认为None。只有当参数C为浮点型数组时,camp参数才有效。 -
norm可选参数,代表数据
亮度,默认为None。只有当参数C为浮点型数组时,norm参数才有效。 -
vmin&vmax可选参数,与norm类似,若已使用norm参数,则此参数无效。
-
alpha可选参数,代表数据
透明度,默认为None。数值在0至1之间,0代表透明,1代表不透明。 -
linewidths可选参数,代表散点图边界线
宽度,默认为None。 -
verts可选参数,代表序列[x,y]。如果参数marker为None,则此参数发挥作用 (作用与marker类似)。
-
edgecolors可选参数,代表散点图边界
颜色,默认为‘face’。
1.1.1 实验任务
1.1.1.1 实验背景
通过输出散点图,展示图中的异常值,加深此方法的理解。
1.1.1.2 实验目标
使用pyplot函数可视化数据集,观测异常值。
1.1.1.3 实验数据解析
实验使用的是鸢尾花数据集。
1.1.2 实验思路
使用散点图可视化数据集,查看异常点:
-
导入实验数据集。
-
利用
matplotlib.pyplot函数库输出散点图。
1.1.3 实验操作步骤
步骤 1 数据导入
#导入pandas库和numpy库
import pandas as pd
import numpy as np
# 导入iris数据集
from sklearn.datasets import load_iris
iris=load_iris()
iris.target_names
输出结果:
array(['setosa', 'versicolor', 'virginica'], dtype='<U10')
解释:dtype=‘<U10’ 表示该数组的数据类型是 Unicode 字符串,每个字符串的最大长度为 10 个字符。其中 <U 表示 Unicode 字符串,而 10 表示字符串的最大长度。
在这个例子中,dtype=‘<U10’ 表示每个字符串的最大长度为 10 个字符。如果字符串的长度超过 10,它们将被截断以适应这个长度。这在鸢尾花数据集中是合理的,因为每个类别的名称都不会超过 10 个字符,所以使用这个数据类型的数组可以有效地存储和表示类别名称。
步骤 2 数据可视化
# 导入 matplotlib.pyplot 函数库
import matplotlib.pyplot as plt
# 画散点图,其中第一维数据为 x 轴,第二维数据为 y 轴
# Sepal.Length(花萼长度,特征的第一列)为 x 轴
x_index=0
# Sepal.Width(花萼宽度,特征的第二列)为 y 轴
y_index=1
# 设置数据集颜色,因为数据集分为三类,因此设置成蓝、红和绿三色。
colors = ['blue', 'red', 'green']
# 先使用 zip 函数将鸢尾花卉种类和颜色打包成元组形式,如[('setosa', 'blue'),('versicolor', 'red'), ('virginica', 'green')]
# 然后使用 for…in…循环,可视化三种数据。
for label, color in zip(range(len(iris.target_names)), colors):
plt.scatter(iris.data[iris.target == label, x_index], iris.data[iris.target == label, y_index], label=iris.target_names[label], c=color)
解释:
当我们进入for循环时,我们将遍历每个类别的鸢尾花数据,并使用不同的颜色来可视化它们。
-
label:在每次迭代中,label代表当前类别的索引。它的取值范围是0到2,对应着鸢尾花数据集中的三个类别(是数据集决定的):setosa(山鸢尾)、versicolor(杂色鸢尾)和virginica(维吉尼亚鸢尾)。
-
color:在每次迭代中,color代表当前类别的颜色。colors列表中定义了三个颜色:‘blue’(蓝色),‘red’(红色)和’green’(绿色)。zip函数将类别的索引和颜色打包成元组的形式,以便在每次迭代中同时访问它们。
-
iris.data[iris.target == label, x_index]:这是一个 NumPy 数组的索引操作。iris.data是鸢尾花数据集中的特征数据,iris.target是对应的目标变量。iris.target == label是一个布尔数组,它的值为True或False,用于指示目标变量
是否等于当前的类别索引。通过这个布尔数组,我们可以筛选出属于当前类别的特征数据。x_index表示我们希望在散点图中使用特征数据的哪个维度作为 x 轴。 -
iris.data[iris.target == label, y_index]:类似于前面的操作,这里是选择特征数据的另一个维度作为 y 轴。
-
label=iris.target_names[label]:这是为散点图中每个类别的数据添加标签。iris.target_names是鸢尾花数据集中目标变量的类别名称数组。通过label索引,我们可以获取当前类别的名称,并将其作为标签显示在散点图中。
-
c=color:这是为散点图中每个类别的数据设置颜色。c参数用于指定散点的颜色,而color变量则是当前类别对应的颜色。
综上所述,该for循环遍历了鸢尾花数据集中的每个类别,根据类别的索引选择相应的特征数据作为 x 轴和 y 轴,并使用不同的颜色和标签将它们可视化成散点图。这样可以直观地展示不同类别之间在两个特征维度上的分布情况。
输出结果如下:
补充学习一:
zip() 函数非常有用。它会返回一个由元组组成的迭代器,每个元组中包含来自输入可迭代对象的对应元素。
names = ['Alice', 'Bob', 'Charlie']
ages = [25, 30, 35]
for name, age in zip(names, ages):
print(name, age)
输出结果:
Alice 25
Bob 30
Charlie 35
补充学习二:
假设我们有以下数据:
iris.data = [[5.1, 3.5, 1.4, 0.2],
[4.9, 3.0, 1.4, 0.2],
[6.2, 2.9, 4.3, 1.3],
[5.5, 2.4, 3.8, 1.1],
[6.3, 3.3, 6.0, 2.5]]
iris.target = [0, 0, 1, 1, 2]
iris.data是一个二维数组,表示鸢尾花的特征数据(分别为: 花萼长度,花萼宽度,花瓣长度,花瓣宽度 )。每一行代表一个样本,每一列代表一个特征。 iris.target 是一个一维数组,表示对应样本的类别标签。
现在,我们来解释 iris.data[iris.target == label, x_index] 这个表达式,假设label为1,x_index为2。
-
iris.target == label:这是一个布尔数组,它的长度与iris.target相同。它的值为True或False,表示对应位置的样本是否属于label所代表的类别。在我们的例子中,iris.target == label的结果为[False, False, True, True, False]。
-
iris.data[iris.target == label]:这是一个数组索引操作,它使用布尔数组iris.target == label作为索引,
从iris.data中筛选出对应位置为True的行。在我们的例子中,这个表达式的结果是一个二维数组:
[[6.2, 2.9, 4.3, 1.3],
[5.5, 2.4, 3.8, 1.1]]
- iris.data[iris.target == label, x_index]:这是一个二维数组的索引操作,它使用布尔数组iris.target == label筛选出的行,并选择其中的第x_index列(
此处为2,即第3列)作为结果。在我们的例子中,这个表达式的结果是一个一维数组:
[4.3, 3.8]
所以, iris.data[iris.target == label, x_index] 的意思是,从iris.data中选择属于label类别的样本,并且只保留这些样本的第x_index个特征值。
1.1.4 结果验证
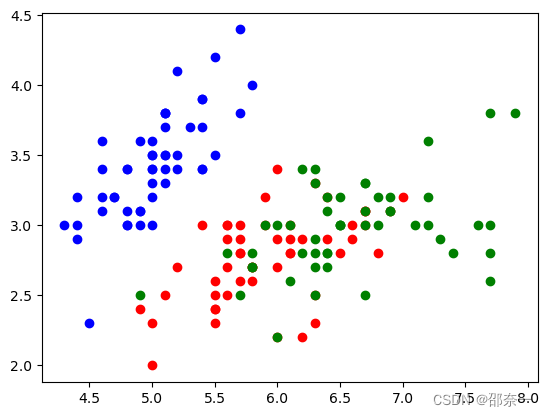
从图中可以得到,对于左上方数据点集群 (setosa,蓝圆点) 来说,左下角的蓝圆点离setosa数据集群较远,可将其认为异常值。
1.2 基于分类模型的异常检测
本实验以决策树模型为例。
Python的 sklearn.tree 库中的 DecisionTreeClassifier 函数可进决策树建模和预测,以便进行数异常检测。其基本格式如下:
DecisionTreeClassifier(criterion='gini', splitter='best', max_depth=None, min_samples_split=2, min_samples_leaf=1, min_weight_fraction_leaf=0.0, max_features=None, random_state=None, max_leaf_nodes=None, min_impurity_decrease=0.0, min_impurity_split=None, class_weight=None, presort=False)
关键参数详解:
-
criterion可选参数,代表特征选择标准,criterion=gini为
基尼系数,criterion=entropy为信息增益。默认为gini。 -
splitter可选参数,代表特征划分点选择标准,splitter=best为针对所有特征找出最优的特征划分点,splitter=random为随机的在部分特征中找局部最优的划分点。默认为best,适用于小样本数据,若数据量过大,则random较为合适。
-
max_depth可选参数,代表树的最大深度,max_depth=None为不限制树的深度,适用于小样本数据。若数据量过大,则需设置树的最大深度,10-100较为合理。默认为None。
-
min_samples_split可选参数,代表内部节点划分所需最小样本数。若某节点的样本数少于min_samples_split,则停止继续选择最优特征划分。默认为2。
-
min_samples_leaf可选参数,代表叶子节点最少样本数,默认值1。
-
min_weight_fraction_leaf可选参数,代表叶子节点最小的样本权重和。若样本有较多缺失值,则引入样本权重。默认为0,不考虑权重问题。
-
max_features可选参数,代表划分时的最大特征数。若为log2则划分时最多考虑log2(n_features)个特征;若为sqrt或auto则划分时最多考虑sqrt(n_features)个特征。默认为None。
-
max_leaf_nodes可选参数,代表构建树时的最大叶子节点数量。默认为None。
-
presort可选参数,Boolean,代表数据是否排序。数据量较小时,对数据排序可加快建模速度。默认为False。
1.2.1 实验任务
1.2.1.1 实验背景
利用分类算法将数据集分类,加深此方法的理解。
1.2.1.2 实验目标
使用分类模型判断数据A和B是否异常。
1.2.1.3 实验数据解析
实验使用的是鸢尾花数据集。
1.2.2 实验思路
使用分类模型 (tree模型) 进行异常值检测:
-
导入实验数据集。
-
生成实验测试集。
-
利用实验数据集进行回归建模。
-
利用建好的回归模型,对实验测试集中的缺失值进行预测。
1.2.3 实验操作步骤
测试数据如下。
-
“sepal length (cm)”:[5.6,4.9],
-
“sepal width (cm)”:[2.5,3],
-
“petal length (cm)”:[4.5,1.4],
-
“petal width (cm)”:[0.2,2.1]。
-
“targets”:[1,2]。
异常值检测数据集,如下:
| Index | sepal length (cm) | sepal width (cm) | petal length (cm) | petal width (cm) | Y |
|---|---|---|---|---|---|
| A | 5.6 | 2.5 | 4.5 | 0.2 | 1 |
| B | 4.9 | 3.0 | 1.4 | 2.1 | 2 |
步骤 1 导入数据集
# 导入 pandas 库和 numpy 库
import pandas as pd
import numpy as np
# 导入数据集 iris
from sklearn.datasets import load_iris
iris = load_iris()
# 导入 iris 数据集,自变量 X 命名为 dfx,分类变量 Y 命名为 dfy。
dfx = pd.DataFrame(data=iris.data, columns=iris.feature_names)
dfx
输出结果如下:

# 部分关键数据展示
dfy=pd.DataFrame(data=iris.target)
dfy
输出结果:
? 0
0 0
1 0
2 0
3 0
4 0
... ...
145 2
146 2
147 2
148 2
149 2
步骤 2 生成测试集
因iris无缺失值,因此人为生成测试集x_test,来利用决策树模型进行异常检测。
x_test = pd.DataFrame({"sepal length (cm)": [5.6, 4.9],
"sepal width (cm)": [2.5, 3],
"petal length (cm)": [4.5, 1.4],
"petal width (cm)": [0.2, 2.1]})
步骤 3 建立DecisionTreeClassifier模型
# 导入 DecisionTreeClassifier
from sklearn.tree import DecisionTreeClassifier
modelcart = DecisionTreeClassifier()
modelcart.fit(dfx,dfy)
输出结果如下:
DecisionTreeClassifier()
步骤 4 判断数据A和B是否为异常值
# 使用建立好的 tree 模型检验数据点 A 和 B 是否正确
predicted = modelcart.predict(x_test)
predicted
输出结果如下:
array([1, 0])
1.2.4 结果验证
步骤四对实验结果进行了验证,正确检测出异常值。根据决策树模型识别出A理论上应为类别1 (测试数据中A为类别1);B理论上应为类别2 (测试数据中B为类别0)。因此数据A不是异常值,B是异常值。
1.3 3σ原则
3σ原则 又称为 拉依达准则 ,先假设一组检测数据只含有随机误差,对原始数据进行计算处理得到标准差,然后按一定的概率确定一个区间,认为误差超过这个区间的就属于异常值。一般其值与平均值的偏差绝对值超过三倍标准差的值,则为异常值,需删除。
解释:
3σ原则(Three Sigma Rule),也称为 3倍标准差原则 ,是统计学中的一个概念,用于描述 正态分布 (或近似正态分布)中观测值离均值的距离。
根据3σ原则,对于一个符合正态分布的数据集,大约有:
- 68% 的观测值落在均值的一个标准差范围内;
- 95% 的观测值落在均值的两个标准差范围内;
- 99.7% 的观测值落在均值的三个标准差范围内。
这个原则可以用来判断一个观测值是否异常或离群。如果一个观测值距离均值超过三个标准差,那么可以认为它是一个异常值或离群点。
3σ原则在质量管理、过程控制和数据分析等领域得到广泛应用。它可以帮助识别潜在的问题、异常情况或变化,并指导决策和改进措施。然而,需要注意的是,3σ原则是基于 正态分布 假设的,如果数据不满足正态分布,应谨慎使用该原则。
1.3.1 实验任务
1.3.1.1 实验背景
利用3σ原则发现数据集中的异常值,加深此方法的理解。
1.3.1.2 实验目标
使用3σ原则发现数据集中的异常值。
1.3.1.3 实验数据解析
使用鸢尾花数据集。
1.3.2 实验思路
使用3σ原则发现数据集中异常值:
-
导入实验数据集。
-
根据3σ原则的公式计算出异常值。
1.3.3 实验操作步骤
步骤 1 导入数据集
iris是150*4的数据集,为实验过程更易被理解。特取其中一个属性进行3σ原则实验。本实验选取iris数据集中的第一维数据 sepal length (cm) 。
# 选取第一维数据 sepal length (cm),记为数据集 X
from sklearn.datasets import load_iris
iris=load_iris()
X=iris.data[:,1]
X
输出结果如下:
# 部分关键数据展示
array([3.5, 3. , 3.2, 3.1, 3.6, 3.9, 3.4, 3.4, 2.9, 3.1, 3.7, 3.4, 3. ,
? 3. , 4. , 4.4, 3.9, 3.5, 3.8, 3.8, 3.4, 3.7, 3.6, 3.3, 3.4, 3. ,
? 3.4, 3.5, 3.4, 3.2, 3.1, 3.4, 4.1, 4.2, 3.1, 3.2, 3.5, 3.1, 3. ,
? 3.4, 3.5, 2.3, 3.2, 3.5, 3.8, 3. , 3.8, 3.2, 3.7, 3.3, 3.2, 3.2,
? 3.1, 2.3, 2.8, 2.8, 3.3, 2.4, 2.9, 2.7, 2. , 3. , 2.2, 2.9, 2.9,
? 3.1, 3. , 2.7, 2.2, 2.5, 3.2, 2.8, 2.5, 2.8, 2.9, 3. , 2.8, 3. ,
补充解释:
iris.data[:,1] 是二位数组,举例:
import numpy as np
ar1=np.arange(12).reshape((4, 3))
ar1
array([[ 0, 1, 2],
[ 3, 4, 5],
[ 6, 7, 8],
[ 9, 10, 11]])
获取二维数组的第一列:
ar1[:,0]
输出结果为:
array([0, 3, 6, 9])
获取二维数组的第二列:
ar1[:,1]
输出结果为:
array([1, 4, 7, 10])
输出前两行的第一列:
ar1[:2,0]
输出结果为:
array([0, 3])
步骤 2 计算数据集的均值
# 导入 numpy 库,可计算均值和标准差
import numpy as np
mean=np.mean(X)
mean
输出结果如下:
3.0573333333333337
步骤 3 计算数据集的标准差和三倍标准差
# 计算数据集 X 的标准差
standard=np.std(X)
standard
输出结果如下:
0.4321465800705435
计算三倍标准差:
b=3*standard
b
输出结果如下:
1.3032329032064838
步骤 4 计算数据集X与平均值的偏差的绝对值
a=abs(X-mean)
a
输出结果如下:
# 部分关键数据展示
array([0.44266667, 0.05733333, 0.14266667, 0.04266667, 0.54266667,
0.84266667, 0.34266667, 0.34266667, 0.15733333, 0.04266667,
0.64266667, 0.34266667, 0.05733333, 0.05733333, 0.94266667,
1.34266667, 0.84266667, 0.44266667, 0.74266667, 0.74266667,
0.34266667, 0.64266667, 0.54266667, 0.24266667, 0.34266667,
0.05733333, 0.34266667, 0.44266667, 0.34266667, 0.14266667,
0.04266667, 0.34266667, 1.04266667, 1.14266667, 0.04266667,
0.14266667, 0.44266667, 0.54266667, 0.05733333, 0.34266667,
0.44266667, 0.75733333, 0.14266667, 0.44266667, 0.74266667,
0.05733333, 0.74266667, 0.14266667, 0.64266667, 0.24266667,
步骤 5 判断异常值
计算数据集X与平均值的偏差绝对值是否超过三倍标准差,并输出异常值。
# 利用 for 循环将数据集 X 的偏差绝对值依次与三倍标准差对比,超过则为异常值并输出异常值。
for i in range(1, 150):
if a[i] > b:
print('当前异常值为 :', a[i])
print('当前异常值位置为 :', i)
输出结果如下:
当前异常值为 : 1.3426666666666667
当前异常值位置为 : 15
1.3.4 结果验证
经实验发现,第一维 sepal length (cm) 的第15个数据为异常值,因为其与平均值的偏差的绝对值超多三倍标准差。
对于小样本数据集,3σ原则可能不太适用。在小样本情况下,由于数据点较少,计算均值和标准差可能会受到极端值或离群点的影响,从而导致均值和标准差的估计不准确。
此外,如果数据集不符合正态分布假设,即使数据量很大,也应该谨慎使用3σ原则。在非正态分布的情况下,数据点的分布可能不服从均值加减3倍标准差的规律。
1.4 箱型图分析
箱线图依据实际数据绘制,其判断异常值的标准以四分位数和四分位距为基础,分为上四分位、下四分位、IQR,由此计算出最大估计值和最小估计值。其基本格式如下:
DataFrame.describe()
此函数输出得到如下结果。
输出详解:
-
Count代表数据集数量。
-
Mean代表数据集均值。
-
Std代表数据集标准差。
-
Min代表数据集最小值。
-
25%代表数据集中25%分位数据。
-
50%代表数据集中50%分位数据。
-
75%代表数据集中75%分位数据。
-
max代表数据集最大值。
1.4.1 实验任务
1.4.1.1 实验背景
利用箱型图发现数据集中的异常值,加深此方法的理解。
1.4.1.2 实验目标
通过箱型图检测预测值,并输出异常值。
1.4.2 实验思路
使用箱型图检测数据集中的异常值:
- 导入实验数据集。
- 根据箱型图计算出异常值。
1.4.3 实验操作步骤
步骤 1 导入数据和函数库
iris是150*4的数据集,为实验过程更易被理解。特取其中一个属性进行3σ原则实验。本实验选取数据集中的第二维 sepal width (cm) (可以尝试用第一个维度看有面试结果【无异常值】)。
# 导入 pandas 和 matplotlib 库
import matplotlib.pyplot as plt
import pandas as pd
from sklearn import datasets
iris = datasets.load_iris()
statistics=iris.data[:,1]
statistics
输出结果如下:
# 数据展示
array([3.5, 3. , 3.2, 3.1, 3.6, 3.9, 3.4, 3.4, 2.9, 3.1, 3.7, 3.4, 3. ,
3. , 4. , 4.4, 3.9, 3.5, 3.8, 3.8, 3.4, 3.7, 3.6, 3.3, 3.4, 3. ,
3.4, 3.5, 3.4, 3.2, 3.1, 3.4, 4.1, 4.2, 3.1, 3.2, 3.5, 3.6, 3. ,
3.4, 3.5, 2.3, 3.2, 3.5, 3.8, 3. , 3.8, 3.2, 3.7, 3.3, 3.2, 3.2,
3.1, 2.3, 2.8, 2.8, 3.3, 2.4, 2.9, 2.7, 2. , 3. , 2.2, 2.9, 2.9,
3.1, 3. , 2.7, 2.2, 2.5, 3.2, 2.8, 2.5, 2.8, 2.9, 3. , 2.8, 3. ,
2.9, 2.6, 2.4, 2.4, 2.7, 2.7, 3. , 3.4, 3.1, 2.3, 3. , 2.5, 2.6,
3. , 2.6, 2.3, 2.7, 3. , 2.9, 2.9, 2.5, 2.8, 3.3, 2.7, 3. , 2.9,
3. , 3. , 2.5, 2.9, 2.5, 3.6, 3.2, 2.7, 3. , 2.5, 2.8, 3.2, 3. ,
3.8, 2.6, 2.2, 3.2, 2.8, 2.8, 2.7, 3.3, 3.2, 2.8, 3. , 2.8, 3. ,
2.8, 3.8, 2.8, 2.8, 2.6, 3. , 3.4, 3.1, 3. , 3.1, 3.1, 3.1, 2.7,
3.2, 3.3, 3. , 2.5, 3. , 3.4, 3. ])
步骤 2 利用 describe () 得到25%分位数据、75%分位数据
# describe()适用于 DataFrame 结构的数据,因此把 statistics 转化成 DataFrame结构
statistics=pd.DataFrame(statistics)
c=statistics.describe()
c
输出结果如下:
0
count 150.000000
mean 3.057333
std 0.435866
min 2.000000
25% 2.800000
50% 3.000000
75% 3.300000
max 4.400000
思考:什么叫什么是25% 分位数(25%)、中位数(50%)、75% 分位数(75%)?
回答:
-
25%分位数(第一四分位数):它将数据集划分为四个等份之一,并表示数据集中的25%的观测值位于该值以下。换句话说,
25%分位数是将数据集从小到大排序后,位于25%位置的值。有时也被称为下四分位数(Lower Quartile)。 -
中位数(第二四分位数):它将数据集划分为两个等份,即中间位置。中位数表示数据集中的50%的观测值位于该值以下。如果数据集中的观测值个数为奇数,则中位数是中间的值;如果观测值个数为偶数,则中位数是中间两个值的平均值。
-
75%分位数(第三四分位数):它将数据集划分为四个等份之一,并表示数据集中的75%的观测值位于该值以下。换句话说,75%分位数是将数据集从小到大排序后,位于75%位置的值。有时也被称为上四分位数(Upper Quartile)。
步骤 3 计算IQR
IQR=c.loc['75%']-c.loc['25%']
IQR
思考:IQR是什么意思?
IQR(Interquartile Range)是数据集中的一个统计量,表示75%分位数与25%分位数之间的距离。
IQR的计算可以提供关于数据集中间50%的离散程度的信息。它衡量了数据集中离散值的范围,即数据集中的中间50%观测值的离散程度。 较大的IQR值表示数据的离散程度较大,而较小的IQR值表示数据的离散程度较小 。
在实际应用中,IQR常用于识别数据集中的离群值(异常值)。根据常用的经验法则,可以将超过1.5倍IQR的值定义为潜在的离群值。 通过计算IQR并结合其他方法,可以帮助识别和处理数据集中的异常值,从而提高数据分析的准确性和可靠性。
输出结果:
0 0.5
dtype: float64
解释:输出的IQR值为0.5,这意味着,在这个特征上,数据的分布相对较紧密,观测值之间的差异较小。
步骤 4 计算最大预估值和最小预估值
# 最大预估值为8.35
maxCount=c.loc['75%']+1.5*IQR
maxCount
输出结果如下:
0 4.05
dtype: float64
解释:根据输出结果,最大预估值为4.05。这个结果告诉你,在这个特定的特征上,超过4.05的观测值可以被视为潜在的离群值。 超过这个值的观测值可能是相对较远离数据集中心的异常值。
# 最小预估值为 2.05
minCount=c.loc['25%']-1.5*IQR
minCount
输出结果如下:
0 2.05
dtype: float64
解释:在这个特定的特征上,小于2.05的观测值可以被视为潜在的离群值。 小于这个值的观测值可能是相对较远离数据集中心的异常值。通过计算最大预估值和最小预估值,你可以得到一个范围,在这个范围之外的观测值可能被认为是潜在的离群值。
步骤 5 验证数据集中是否有异常值,并输出异常值
# 取出 iris 第二维数据循环输出异常值。
X = iris.data[:, 0]
for i in range(1, 150):
if X[i] > maxCount.values or X[i] < minCount.values:
print('当前异常值为 :', X[i])
print('当前异常值位置为 :', i)
部分输出结果如下:
当前异常值为 : 4.4
当前异常值位置为 : 15
当前异常值为 : 4.1
当前异常值位置为 : 32
当前异常值为 : 4.2
当前异常值位置为 : 33
当前异常值为 : 2.0
当前异常值位置为 : 60
for循环结构不适用于DataFrame结构的数据, describe() 要求数据为DataFrame结构,因此statistic数据集不适用于for循环。在此实验中,使用原始数据集X进行for循环输出。
步骤 6 可视化验证分析结果是否准确
plt.figure()
plt.boxplot(statistics)
plt.show()
1.4.4 结果验证
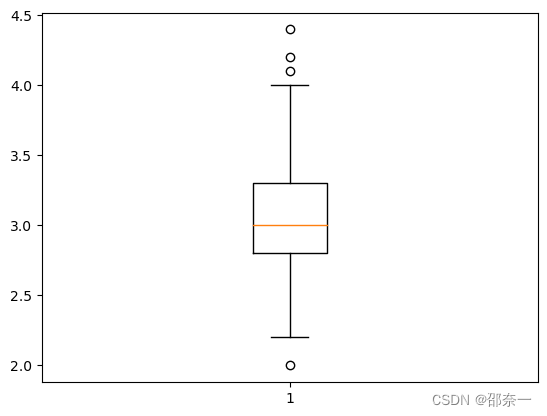
步骤六对实验结果进行了验证,正确检测出异常值。由图可看出,图中的四个圆圈为异常值,分别是 sepal width (cm) 中的第15个,第32个,第33个,第60个数据。
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:chenni525@qq.com进行投诉反馈,一经查实,立即删除!
- Python教程
- 深入理解 MySQL 中的 HAVING 关键字和聚合函数
- Qt之QChar编码(1)
- MyBatis入门基础篇
- 用Python脚本实现FFmpeg批量转换
- JAVA 版多商家入驻 直播带货 商城系统 B2B2C 之 鸿鹄云商B2B2C产品概述
- 编程江湖:Python探秘之旅-----Python 实用工具和库(七)
- FreeSwitch防止sip攻击盗打
- 1.7数算选择题专练
- 更新:TwoNav开源网址导航系统源码/书签(导航)管理工具/去授权版
- Kubernetes Operator
- 运算符重载函数
- 使用工业级以太网交换机,需要注意哪些问题?
- 在modelsim中查看断言
- ubuntu设置ssh登录,设置公钥无密登录