全面解析MONAI Transforms的用法 视频+教程+代码
MONAI提供了一系列的数据预处理操作,称之为transform。transform的目的是将原始数据转化为模型能够接受的格式,同时也可以进行一些数据增强操作,以提高模型的鲁棒性和泛化能力。MONAI中的transform分为两类:vanilla transform和dict transform。在视频中都会详细介绍
MONAI 简介
MONAI是一个基于PyTorch的开源框架,用于医疗影像的深度学习,属于PyTorch生态系统的一部分。
它的目标是:
- 建立一个学术、工业和临床研究人员合作的共同基础社区;
- 创建用于医疗影像的最先进的端到端训练流程;
- 为研究人员提供优化和标准化的方式来创建和评估深度学习模型。
MONAI旨在支持医学图像分析中的深度学习,下图显示了医学深度学习领域端到端工作流程的典型示例:
为什么需要MONAI呢?
生物医学应用有特定的需求:
- 图像模态(MR、CT、US等)需要特定的数据处理方法。
- 数据格式(DICOM、NIfTI等)是专门针对医学应用的,需要特殊的支持。
- 某些网络架构被设计用于医学应用,或者非常适合医学应用。
MONAI通过灵活的PyTorch兼容方式,提供了深度学习设施和基础架构来满足这些需求:
- 提供用于处理生物医学文件类型的数据加载和处理库。
- 大量的数据转换操作,用于在训练前、训练中和训练后处理、规范化和增强图像数据。
- 通用网络、评估和损失函数定义的库,实现了常见的架构。
- 一组可用于高效利用计算基础设施的训练和推断的现成组件。
- MONAI通过简化实验的训练和分布,有助于实现可复现性:
以上是 monai 的一个简要介绍,在这部分,主要介绍 monai transform的用法。通过官方代码,视频,Tina姐之前的博客进行全方位的学习。
【添加视频】视频在公众号相关文章中
在之前的博客中,我们拆解了常用的数据增强transform操作,并讨论了如何评估这些变换对数据的效果,并使用Python进行可视化展示。
MONAI transform
为了帮助您更好地了解MONAI Transforms,本指南将帮助您回答以下五个关键问题:
- 有哪些 Transforms 可用于创建训练数据管道?
- 什么是 array transforms?
- 如何自定义 transforms?
- 什么是 dictionary transforms?
- 如何使用transforms创建 basic dataset?
让我们先导入必要的依赖项。
import tempfile
import nibabel as nib
import numpy as np
import matplotlib.pyplot as plt
from typing import Optional, Any, Mapping, Hashable
import monai
from monai.config import print_config
from monai.utils import first
from monai.config import KeysCollection
from monai.data import Dataset, ArrayDataset, create_test_image_3d, DataLoader
from monai.transforms import (
Transform,
MapTransform,
Randomizable,
AddChannel,
AddChanneld,
Compose,
LoadImage,
LoadImaged,
Lambda,
Lambdad,
RandSpatialCrop,
RandSpatialCropd,
ToTensor,
ToTensord,
Orientation,
Rotate
)
print_config()
1 有哪些 Transforms 可用于创建训练数据管道?
Medical image data I/O, processing and augmentation: 医学图像需要高度专业化的方法进行输入/输出、预处理和增强。医学图像通常以专门的格式存储,包含丰富的元信息,并且数据容量通常是高维的。这些要求需要经过精心设计的操作流程。
Transforms 支持 Dictionary and Array 格式的数据,一共有六个类别的Transform:裁剪和填充、强度、输入/输出、后处理、空间和实用工具。 有关更多详细信息,请访问MONAI transform
特定于医学的transfroms:MONAI旨在提供全面的医学图像特定转换。目前包括以下内容:
- LoadImage:从指定路径加载医学专用格式文件
- Spacing:将输入图像重新采样为指定的像素尺寸
- Orientation:将图像的方向更改为指定的axcodes
- RandGaussianNoise:通过添加统计噪声扰动图像强度
- NormalizeIntensity:基于均值和标准差进行强度归一化
- Affine:根据仿射参数对图像进行变换
- Rand2DElastic:在二维空间中进行随机弹性变形和仿射变换
- Rand3DElastic:在三维空间中进行随机弹性变形和仿射变换
我们将创建一个临时目录,并用几个示例的Nifti文件格式的图像填充它,这些图像包含了一些随机排列的球体。我们还将创建一个匹配的segmentation ground truth,稍后在笔记本中使用。
fn_keys = ("img", "seg") # filename keys for image and seg files
root_dir = tempfile.mkdtemp()
filenames = []
for i in range(5):
im, seg = create_test_image_3d(256, 256, 256, num_objs=25, rad_max=50)
im_filename = f"{root_dir}/im{i}.nii.gz"
seg_filename = f"{root_dir}/seg{i}.nii.gz"
filenames.append({"img": im_filename, "seg": seg_filename})
n = nib.Nifti1Image(im, np.eye(4))
nib.save(n, im_filename)
n = nib.Nifti1Image(seg, np.eye(4))
nib.save(n, seg_filename)
什么是 array transforms?
在MONAI中,transform是可调用对象,接受来自数据集中初始数据或先前transform 的输入。我们可以直接创建和调用这些transform,而无需进行任何基础设施或系统设置,因为MONAI中的组件设计尽可能解耦。例如,我们可以通过创建transform并调用它来直接加载其中一个Nifti文件。

transform可以使用Compose组合成一个操作序列。由于Compose本身是一个transform,我们也可以直接调用它。这里的img类型是numpy.ndarray,所以为了将其转换为Pytorch张量作为训练数据管道的一部分,我们将在序列中的最后一个变换使用ToTensor
如何自定义 transforms?
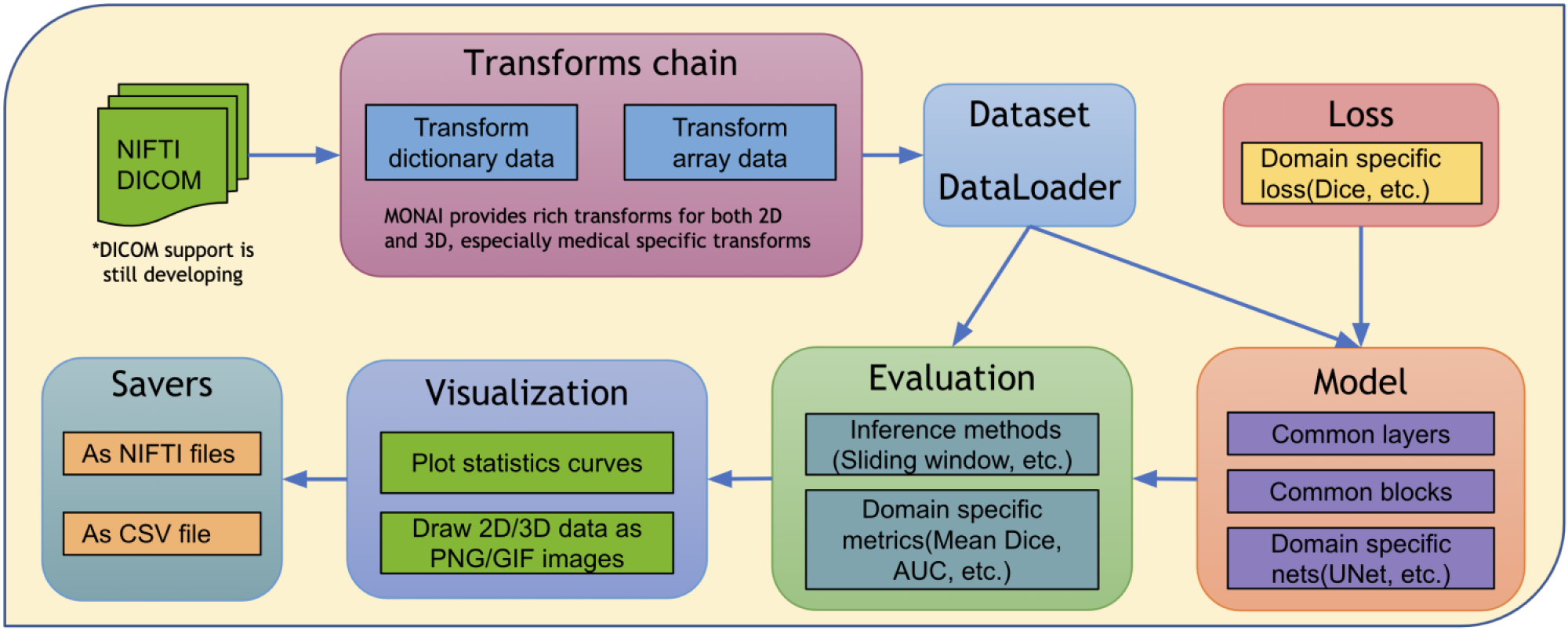
我们可以通过多种方式定义自己的自定义transform。如果使用简单的可调用对象作为操作符,可以使用Lambda将其包装为transform。在本例中,我们定义了一个变换,将第1个(宽度)维度中的图像求和以产生2D图像:
def sum_width(img):
return img.sum(1)
trans = Compose([LoadImage(image_only=True), AddChannel(), Lambda(sum_width)])
img = trans(filenames[0]["img"])
plt.imshow(img[0])
创建Transform子类第二种方法,它的优点是可以在实例化对象时定义属性。让我们定义一个类来在选择的维度上求和,并使用它在第2个(高度)维度上求和
class SumDimension(Transform):
def __init__(self, dim=1):
self.dim = dim
def __call__(self, inputs):
return inputs.sum(self.dim)
trans = Compose([LoadImage(image_only=True), AddChannel(), SumDimension(2)])
img = trans(filenames[0]["img"])
plt.imshow(img[0])
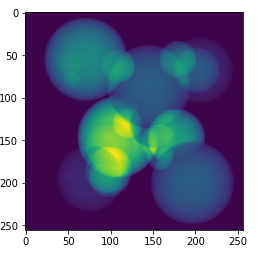
到目前为止,所有这些示例变换都是确定性的。要定义在输入数据上执行某些随机操作的变换,我们还希望继承Randomizable。这个类用于随机化变量,并与确定性变换区分开来。稍后我们将看到为什么这一点很重要,涉及到缓存数据加载器。
在这个类中,我们有一个numpy.random.RandomState对象来提供随机值。可以使用Randomizable.set_random_state()替换它,以控制随机化过程。randomize()方法负责根据prob概率成员确定是否执行随机操作,如果是,则创建随机噪声数组。这个功能在这个方法中是为了可以由Compose或其他外部控制器调用。
现在让我们定义一个简单的变换来添加噪声。
什么是 dictionary transforms?
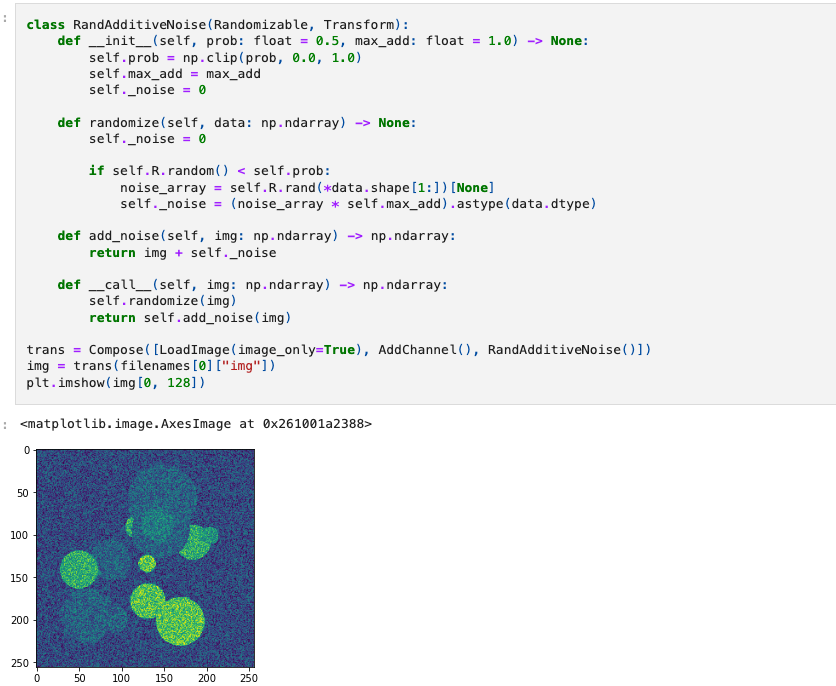
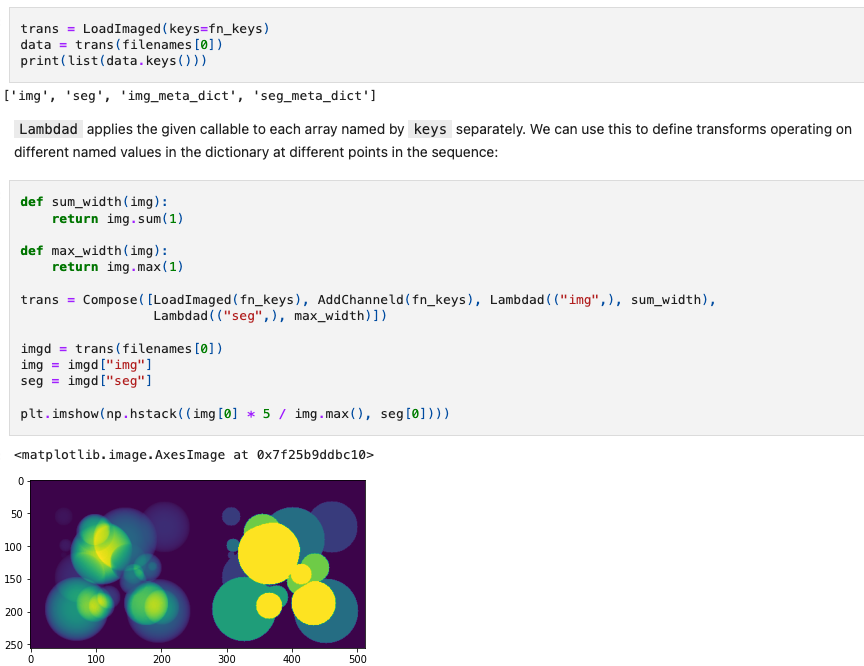
到目前为止,我们已经看到了应用于单个Numpy数组的变换,然而对于大多数训练方案,需要一个包含多个值的流水线。为了解决这个问题,MONAI包括了用于操作数组字典的变换,每个等效的数组变换都有一个对应的字典变换。这些变换可以应用于输入字典中的命名值,同时保持未命名值不变,例如在图像上添加噪声而保持相关标签图像不变。
在笔记本的早些时候,我们导入了字典等效的变换,它们的名称后面加上了d,我们将在本节中使用这些变换。LoadNiftid中的keys参数用于指定哪些键包含Nifti文件的路径,输入字典中的所有其他值将被保留。设置好之后,我们可以查看调用变换时返回的键值:
如何使用transforms创建 basic dataset?
现在我们已经了解了transform,让我们看看数据集。有了定义的数据源和变换,我们现在可以创建一个数据集对象。MONAI的基类是Dataset,这里只用于加载Nifti图像文件。
Dataset继承自Pytorch中同名的类,仅添加了将给定的变换应用于所选项的功能。如果您熟悉Pytorch中的该类,则它的使用方式相同。
images = [fn["img"] for fn in filenames]
transform = Compose([LoadImage(image_only=True), AddChannel(), ToTensor()])
ds = Dataset(images, transform)
img_tensor = ds[0]
print(img_tensor.shape, img_tensor.get_device())
MONAI专门为监督式训练应用提供了ArrayDataset。它可以接受针对图像的数据数组以及针对分割或标签的数据数组,并具有各自独立的变换操作。在这里,我们将再次分开图像和分割文件名,以演示这种用法:
images = [fn["img"] for fn in filenames]
segs = [fn["seg"] for fn in filenames]
img_transform = Compose([LoadImage(image_only=True), AddChannel(),
RandSpatialCrop((128, 128, 128), random_size=False), RandAdditiveNoise(), ToTensor()])
seg_transform = Compose([LoadImage(image_only=True), AddChannel(),
RandSpatialCrop((128, 128, 128), random_size=False), ToTensor()])
ds = ArrayDataset(images, img_transform, segs, seg_transform)
im, seg = ds[0]
plt.imshow(np.hstack([im.numpy()[0, 48], seg.numpy()[0, 48]]))
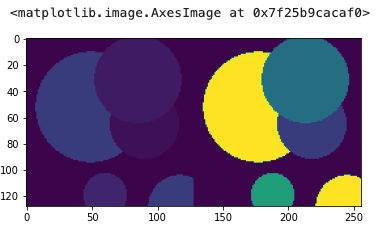
访问数据集的成员时,不再返回单个图像,而是返回一个包含经过各自变换处理后的图像和分割结果的元组。这个类的一个重要方面是,在应用变换之前,将每个变换(在本例中是Compose)的随机状态设置为数据集的随机状态。这确保对每个输出应用相同的随机操作,这就是为什么RandSpatialCrop操作会选择相同的裁剪窗口用于图像和分割的原因。通过使用独立的变换,可以对图像应用操作而不对分割进行操作(或反之),但要注意这些非共享操作要在共享操作之后执行。
另外,Dataset还可以与基于字典的变换一起使用,以构建结果映射。对于超出简单的输入/标签配对的训练应用,这种方式更合适:
trans = Compose([LoadImaged(fn_keys), AddChanneld(fn_keys), RandAdditiveNoised(("img",)),
RandSpatialCropd(fn_keys, (128, 128, 128), random_size=False), ToTensord(fn_keys)])
ds = Dataset(filenames, trans)
item = ds[0]
im, seg = item["img"], item["seg"]
plt.imshow(np.hstack([im.numpy()[0, 48], seg.numpy()[0, 48]]))
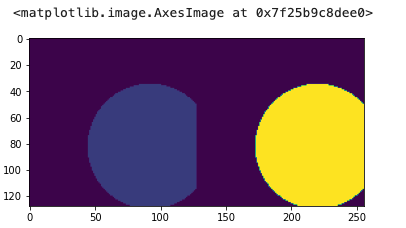
有了定义的数据集,我们现在可以创建dataloader以创建数据批次。这直接继承自Pytorch的DataLoader类,但有一些更改默认构造函数参数。MONAI功能应与PyTorch DataLoader兼容,但它是子类,包括我们认为关键并且不能使用标准DataLoader类实现的附加功能。
DataLoader将使用五个工作进程来加载实际数据。MONAI提供了许多数据集子类来提高此过程的效率。这些和其他功能将在接下来的实验中介绍。
loader = DataLoader(ds, batch_size=5, num_workers=5)
batch = first(loader)
print(list(batch.keys()), batch["img"].shape)
f, ax = plt.subplots(2, 1, figsize=(8, 4))
ax[0].imshow(np.hstack(batch["img"][:, 0, 64]))
ax[1].imshow(np.hstack(batch["seg"][:, 0, 64]))

array transform和dictionary transfrom的区别弄明白了吗?
Tina姐说:二者就是喂数据的方式不一样。image和label是以数组形式给到Dataset就使用array transform;image和label是以字典对形式就使用dictionary Transform。功能是一样的,只是dictionary Transforms在每个变换后面都加了一个"d", 也可以写成”D“。如LoadImage变为LoadImaged, Resize变为Resized。使用dictionary Transforms时,必须指明该变换是对image做,还是label做。如,LoadImaged(keys=‘image’),表明只加载image
# array transfrom
images = [
"IXI314-IOP-0889-T1.nii.gz",
...
"IXI574-IOP-1156-T1.nii.gz",
"IXI585-Guys-1130-T1.nii.gz",
]
# binary labels for gender classification: man and woman
labels = np.array([0, 0, 0, 1, 0, 0, 0, 1, 1, 0, 0, 0, 1, 0, 1, 0, 1, 0, 1, 0], dtype=np.int64)
train_transforms = Compose([ScaleIntensity(), EnsureChannelFirst(), Resize((96, 96, 96)), RandRotate90()])
train_ds = ImageDataset(image_files=images, labels=labels, transform=train_transforms)
# dictionary transfrom
images = [
"IXI314-IOP-0889-T1.nii.gz",
...
"IXI574-IOP-1156-T1.nii.gz",
"IXI585-Guys-1130-T1.nii.gz",
]
# binary labels for gender classification: man and woman
labels = np.array([0, 0, 0, 1, 0, 0, 0, 1, 1, 0, 0, 0, 1, 0, 1, 0, 1, 0, 1, 0], dtype=np.int64)
train_files = [{"img": img, "label": label} for img, label in zip(images, labels)]
train_transforms = Compose(
[
LoadImaged(keys=["img"], ensure_channel_first=True),
ScaleIntensityd(keys=["img"]),
Resized(keys=["img"], spatial_size=(96, 96, 96)),
RandRotate90d(keys=["img"], prob=0.8, spatial_axes=[0, 2]),
]
)
train_ds = monai.data.Dataset(data=train_files, transform=train_transforms)
总结:
我们已经介绍了MONAI的Transforms。以下是一些关键亮点:
- MONAI提供了大量特定于医学领域的变换操作。
- 有数组和字典版本的Transforms。
- 您可以创建一个简单的可调用的lambda函数,或者基于Transform创建一个类来创建自定义的变换。
- 您可以创建一个MONAI数据集,并直接将一个Compose变换链传递给它。
更多阅读:
使用MONAI轻松加载医学公开数据集,包括医学分割十项全能挑战数据集和MedMNIST分类数据集
使用MONAI时,如何选择合适的Dataset加载数据,提升训练速度
文章持续更新,可以关注微公【医学图像人工智能实战营】获取最新动态,一个关注于医学图像处理领域前沿科技的公众号。坚持以实践为主,手把手带你做项目,打比赛,写论文。凡原创文章皆提供理论讲解,实验代码,实验数据。只有实践才能成长的更快,关注我们,一起学习进步~
我是Tina, 我们下篇博客见~
白天工作晚上写文,呕心沥血
觉得写的不错的话最后,求点赞,评论,收藏。或者一键三连

本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:chenni525@qq.com进行投诉反馈,一经查实,立即删除!
- Python教程
- 深入理解 MySQL 中的 HAVING 关键字和聚合函数
- Qt之QChar编码(1)
- MyBatis入门基础篇
- 用Python脚本实现FFmpeg批量转换
- HubSpot CRM:卓越客户服务的关键引擎
- Flutter中实现中国省份地图
- 自监督学习综述
- Nginx 常用变量 与 防盗链
- 数仓项目6.0配置大全(hadoop/Flume/zk/kafka/mysql配置)
- Spring+Vue实战项目环境准备跑通程序
- Web即时通信技术——WebRTC
- msvcp140.dll丢失怎样修复?全面分析msvcp140.dll的修复方法
- 2023年03月22日_谷歌Bard开放公测的解读
- 操作系统详解(5.2)——信号(Signal)的题目进阶