医学图像的数据增强技术 --- 切割-拼接数据增强(CS-DA)
医学图像的新型数据增强技术
?
CS-DA 核心思想
论文链接:https://arxiv.org/ftp/arxiv/papers/2210/2210.09099.pdf
大多数用于医学分割的数据增强技术最初是在自然图像上开发的,没有考虑到医学图像整体布局标准固定的特点。
基于医学图像的特点,作者开发了切割-拼接数据增强(CS-DA)方法,这是一种用于医学图像分割的新型数据增强技术。
CS-DA通过将从不同原始医学图像中切割的不同位置分量拼接成新图像来增强数据集。
CS-DA的思想很简单:
- 将原始医学图像以相同方式切割成多个组件;
- 然后从不同的原始图像中切割出不同位置的组件,
- 将它们拼接在一起形成新的图像。
假设有五张原始医学图像,每张图像都代表不同的医学情境,如X光片或MRI图像。
每张原始图像都被切割成四个相同大小的组件,就像将图像分成四个拼图块一样。
现在,我们可以创建新的图像,通过从这些原始图像中选择不同位置的组件并将它们拼接在一起。
假设我们从第一张原始图像中选择了第一个组件,从第二张原始图像中选择了第二个组件,依此类推,直到从第五张原始图像中选择了第五个组件。
这样,我们就创建了一张新的图像,它的组成部分来自不同原始图像的不同位置。
这个新图像会保持与原始图像相同的整体布局和外观,因为每个组件都是从医学图像中切割出的,并且它们有相似的对象或背景。
与传统的数据增强技术(如Cla-DA)不同,CS-DA不需要对原始图像进行复杂的数学函数处理,只需将组件按矩阵格式拼接起来。
而且,CS-DA不会引入任何噪音或虚假信息到新图像中,因为所有信息都来自于原始图像的合法组件。
更重要的是,Cla-DA技术通过随机改变原始图像的信息来生成新图像,这会引入虚假信息到新图像中。
相比之下,CS-DA不会向原始图像引入任何噪音或虚假信息。
CS-DA创建的新图像中的所有信息都来自原始图像。
- CS:cutting-splicing 切割-拼接
整体布局的一致性导致了不同医学图像中相同位置区域的互换性。
可以通过使用一个医学图像中的区域来替换另一个医学图像中相同位置的区域来创建新的医学图像。
新的医学图像具有与原始医学图像相同的整体布局,并且新医学图像中的对象完整。
在分割任务中,新的医学图像可以与原始医学图像混合在一起,用于训练分割模型。
?
自然图像和医学图像之间的关键差异

自然图像和医学图像之间的关键差异,这些差异在以下六个方面得到反映:摄像机方向、对象姿势、对象位置、对象完整性、对象比例和对象类别。
-
摄像机方向
- 在自然场景中,对摄像机的方向没有具体的要求,可以从任何方向拍摄对象。例如,如图1所示,摄像机可以位于熊的对面或熊的下方。
- 然而,在临床医学中,扫描仪或其他图像获取设备的方向是固定的。例如,肺部X光图像都是在后前位进行扫描的。
-
对象姿势
- 自然场景中的对象在拍摄过程中可以处于任何姿势。例如,在图1中,熊可以玩球或游泳。
- 而在扫描过程中,患者必须保持特定的姿势。对于无法自控的年幼儿童,放射科医生甚至会使用辅助设备或镇静剂来固定他们的身体。因此,扫描的器官在医学图像上也保持特定的形状。
-
对象位置
- 在自然场景中,摄影师会根据自己的布局思想来安排对象在自然图像中的位置。自然图像的布局设计没有固定的模式,因此对象的位置总是随机的。
- 在临床医学中,放射科医生会调整扫描仪以确保器官位于图像的中心或特定位置。
-
对象完整性
- 自然场景复杂多变,有时在对象和摄像机之间会有遮挡物,阻止对象完全显示在自然图像中。例如,图1中的熊的部分身体被池塘中的水阻挡。
- 医学图像在更清洁的场景中获取。不允许在患者和扫描仪之间存在异常的遮挡物,因此可以保证对象的完整性。
-
对象比例
- 自然图像中不记录像素大小信息,因此无法通过自然图像中的对象区域来计算对象的实际大小。
- 相反,在医学图像中,比例是基本信息,通常在文件头中提供像素间距和切片厚度的信息,这可以帮助我们将不同图像中的对象标准化到一个标准空间中。
-
对象类别
- 在自然图像的分割任务中,对象的种类太多,无法精细定义。因此,对象被分配为粗略的类别。例如,图1中的"熊"是一个粗略的类别,可以被细化为"棕熊"或"北极熊"等。
- 相反,人体器官已经被明确定义,医学图像中的每个分割区域都有明确的类别。
在上述六个方面,自然图像具有很多可能性。
这些方面的不确定性使得自然图像变化多样。
因此,包含相同类型对象的自然图像之间存在巨大差异。
另一方面,医学图像在这六个方面受到标准扫描设备、经过良好训练的放射科医生以及严格遵守扫描规范的患者的限制。
因此,在特定的分割任务中,医学图像的整体布局是标准和固定的。
这一特点使得医学图像之间的整体布局保持一致。
?
CS-DA 步骤

当涉及到2D图像时,通常有两个维度可以用于切割,例如横向和纵向。
- 在图2a中,可以将2D视网膜血管图像切割成两个、三个和四个组件,具体取决于切割线的位置和数量。
- 这些切割线将图像分割成不同的部分。
而在3D图像中,通常有三个维度可以用于切割,例如长度、宽度和高度。
- 可以将3D脑图像切割成两个、三个、四个和八个组件,具体取决于切割线的位置和数量。
- 这些切割线将3D图像分割成不同的体积或部分,以便进行进一步的分析或处理。
?
基于医学图像的区域互换性,本研究提出了CS-DA技术,包括两个步骤:切割图像成组件,以及将组件拼接成新图像。

1) 切割图像成组件
- 在这一步骤中,将原始图像切割成多个组件。
- 每个切割线是垂直于图像的某个维度,并穿过整个图像。
- 同一维度中的n条切割线将图像分成(n+1)个相等的组件。
- 在2D图像中有两个可以切割的维度,而3D图像有三个可以切割的维度。
- 同一数据集中的所有图像都以相同的方式进行切割,因此从每个图像中切割出的组件数量是相同的。
- 同样的方式也适用于其分割对象的掩模。
2) 将组件拼接成新图像
- 这一步可以通过两种方法进行:正常拼接(NorS)和对称拼接(SymS)。
-
原始图像的特定位置组件用于填充新图像的相同位置区域(NorS):
- 假设我们有两张原始医学图像,每张图像都代表同一个器官的不同部分。
- 在NorS方法中,我们选择了第一张图像的特定位置的组件,并将其用于填充第二张图像的相同位置。
- 例如,我们选择了第一张图像中的左侧肺部的组件,并将其用于填充第二张图像中的左侧肺部区域。
- 这样,我们创建了一个新的医学图像,它具有与原始图像相同的整体布局,但使用了不同位置的组件。
-
原始图像的翻转组件用于填充新图像的对称位置区域(SymS):
- 假设我们有一张原始医学图像,代表了一个具有对称结构的器官,如肺部。
- 在SymS方法中,我们选择了原始图像中的特定位置的组件,并将其翻转后用于填充新图像的对称位置。
- 例如,我们选择了原始图像中的左侧肺部的组件,并将其翻转后用于填充新图像的右侧肺部区域。
- 由于人体的器官通常具有左右对称性,因此这种翻转操作是可行的,从而保持了图像的整体布局和对称性。
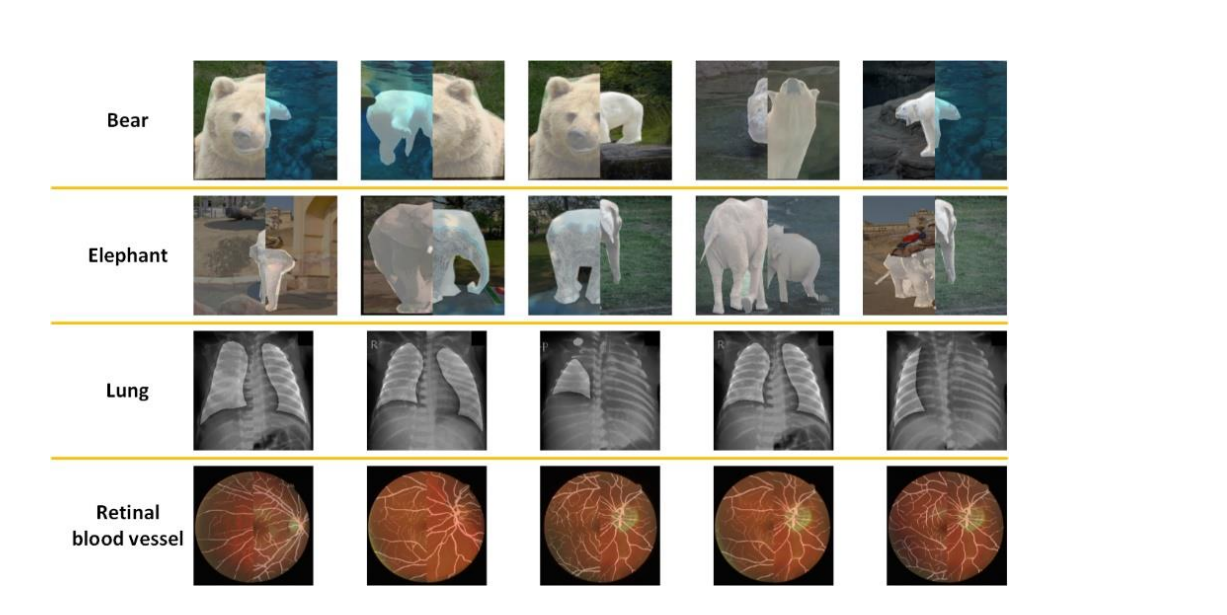
上图展示了由CS-DA创建的一些自然图像和医学图像。
自然图像整体布局的多样性使新图像异常。
新自然图像中对象的完整性被破坏。
相比之下,新医学图像看起来正常。
每个新医学图像都具有完整的对象。
这个过程允许利用医学图像的互换性来增加数据集的多样性,从而提高分割模型的性能。
?
确定增强后的数据数量

假设我们有一个原始数据集,其中包含两个不同的医学图像:
- 图像 A 和图像 B。
每个原始图像都可以被切割成两个组件:
- 左组件(-L)和右组件(-R)。
-
非对称情况(使用NorS方法):
- 原始数据集中有两个图像,每个图像被切割成两个组件。
- 因此,每个原始数据集中有 2 * 2 = 4 个组件。
- 使用NorS方法,我们可以创建新图像,其中组件可以来自不同的原始图像。
- 例如,我们可以将左组件(-L)从图像 A 和右组件(-R)从图像 B 组合在一起形成新图像 A-L+B-R。
- 这个过程可以生成 4 个不同的新图像:A-L+B-R、A-L+A-R、B-L+B-R 和 B-L+A-R。
- 因此,增强后的数据集样本大小是 4。
-
对称情况(使用SymS方法):
- 同样,原始数据集中有两个图像,每个图像被切割成两个组件,总共有 2 * 2 = 4 个组件。
- 使用SymS方法,我们可以创建新图像,其中左组件可以是原始图像的左组件或原始图像的翻转右组件。
- 这个过程可以生成更多的新图像,因为对称性允许更多的组合。
- 例如,我们可以创建新图像 B-L+A-R,其中左组件来自图像 B 的左组件,右组件来自图像 A 的右组件。
- SymS方法可以生成更多的新图像选择,增强后的数据集样本大小是 NorS 方法的四倍,即 4 * 4 = 16。
总结:
确定增强后的数据集样本大小的方法:
-
原始数据集大小: 根据原始数据集中包含的图像数量来确定。
-
组件数量: 根据每个原始图像可以切割成多少个组件来计算。
-
拼接方法: 根据使用的拼接方法来调整样本大小。
-
非对称情况: 如果使用NorS方法,增强后的数据集样本大小等于
原始数据集大小乘以组件数量。 -
对称情况: 如果使用SymS方法,增强后的数据集样本大小是NorS方法的四倍,即
原始数据集大小乘以组件数量乘以4。
?
代码复现
import cv2
import numpy as np
# 定义NorS方法
def cs_da_nors(original_image, num_components):
height, width, _ = original_image.shape
component_width = width // num_components
new_image = np.zeros_like(original_image)
for i in range(num_components):
start_x = i * component_width
end_x = (i + 1) * component_width
component = original_image[:, start_x:end_x, :]
new_image[:, start_x:end_x, :] = component
return new_image
# 定义SymS方法
def cs_da_syms(original_image, num_components):
height, width, _ = original_image.shape
component_width = width // num_components
new_image = np.zeros_like(original_image)
for i in range(num_components):
start_x = i * component_width
end_x = (i + 1) * component_width
component = original_image[:, start_x:end_x, :]
if i % 2 == 1:
component = cv2.flip(component, 1) # 翻转组件
new_image[:, start_x:end_x, :] = component
return new_image
# 使用NorS方法
original_image = cv2.imread("original_image.png") # 假设加载原始图像
num_components = 4
# 保存所有生成图像到文件
for i in range(num_components):
augmented_image_nors = cs_da_nors(original_image, num_components)
cv2.imwrite(f"augmented_image_nors_{i}.png", augmented_image_nors)
# 使用SymS方法
num_components = 4
# 保存所有生成图像到文件
for i in range(num_components):
augmented_image_syms = cs_da_syms(original_image, num_components)
cv2.imwrite(f"augmented_image_syms_{i}.png", augmented_image_syms)
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:chenni525@qq.com进行投诉反馈,一经查实,立即删除!
- Python教程
- 深入理解 MySQL 中的 HAVING 关键字和聚合函数
- Qt之QChar编码(1)
- MyBatis入门基础篇
- 用Python脚本实现FFmpeg批量转换
- SCT52A40——120V,4A,高频高压侧和低压侧栅极驱动器,替代UCC27200/UCC27201/MIC4604YM等
- 力扣题目学习笔记(OC + Swift)15. 三数之和
- 【PostgreSQL】从零开始:(二十八)数据类型-XML类型
- HTTPS的加密流程
- 面试算法114:外星文字典
- JS-元素尺寸与位置
- easyexcle处理导出合并单元格图片填充
- 基于SSM+Jsp+layui的酒店民宿管理系统(Java毕业设计)
- 台灯怎么挑选不伤眼睛?精选适合考研使用的护眼台灯
- 【论文阅读笔记】A Recent Survey of Vision Transformers for Medical Image Segmentation