实用技巧——缺失数据的处理
缺失值
处理缺失数据的一般步骤:
- 识别缺失数据;
- 检查导致数据缺失的原因;
- 删除包含缺失值的实例或用合理的数值代替(插补)缺失值
缺失数据的分类:
(1)完全随机缺失???? 若谋变量的缺失数据与其他任何观测或未观测变量都不相关,则数据为完全随机缺失(MCAR)。
(2)随机缺失??? 若某变量上的缺失数据与其他观测变量相关,与他自己的未观测值不相关,则数据为随机缺失(MAR)。
(3)非随机缺失??? 若缺失数据不属于MCAR和MAR,则数据为非随机缺失(NMAR)。
大部分处理缺失数据的方法都是假定数据是MCAR或MAR,此时可以忽略缺失数据的生成机制,并且(在替换或删除缺失数据后)可以直接对感兴趣的关系进行建模。
当数据是NMAR时,模型处理相较困难,目前有的方法有模型选择法和模式混合法。以下主要讨论的是MCAR和MAR。
案例:VIM包中的哺乳动物睡眠数据(sleep)。该研究了62种哺乳动物的睡眠、生态学变量、和体质变量间的关系。
睡眠变量包括做梦时长(Dream),不做梦时长(NonD)以及它们的和(sleep)。体质变量包括体重(Bodyweight,单位是千克),脑重(BrainWgt),寿命(Span)和妊娠期(Gest)。生态学变量包括物种被捕食的程度(pred),睡眠时暴露程度(Exp)和面临的总危险度(Danger)。
识别缺失值
在R中,NA代表缺失值,NAN(不是一个数)代表不可能值。
符合-Inf和Inf代表负无穷和正无穷。is.na(), is.nan(), 和 is.infinite()可以分别识别缺失值、不可能值和无穷值。
| x | is.na() | is.nan() | is.infinite() |
|---|---|---|---|
| x<- NA | TRUE | FALSE | FALSE |
| x<- 0 / 0 | TRUE | TRUE | FALSE |
| x<- 1 / 0 | FALSE | FALSE | TRUE |
函数complete.cases( )可以用来识别矩阵或数据框中没有缺失值的行。若每一行都有一个或多个缺失值,则返回FALSE
rm(list=ls())
#install.packages("VIM")
data(sleep,package = "VIM")
#列出没有缺失值的行
sleep[complete.cases(sleep),]
#列出有一个或多个缺失值的行
sleep[!complete.cases(sleep),]根据TRUE和FALSE的数量进行统计,可以结合sum()和mean()函数获取相关信息
#Dream变量中缺失值的数目
sum(is.na(sleep$Dream))
#变量Dream中缺失值占的比例
mean(is.na(sleep$Dream))
#在数据框中缺失行所占的比例
mean(!complete.cases(sleep))列表显示缺失值
?mice包中的md.pattern()函数可生成一个以矩阵或数据框形式展示缺失值模型的表格。
library(mice)
md.pattern(sleep)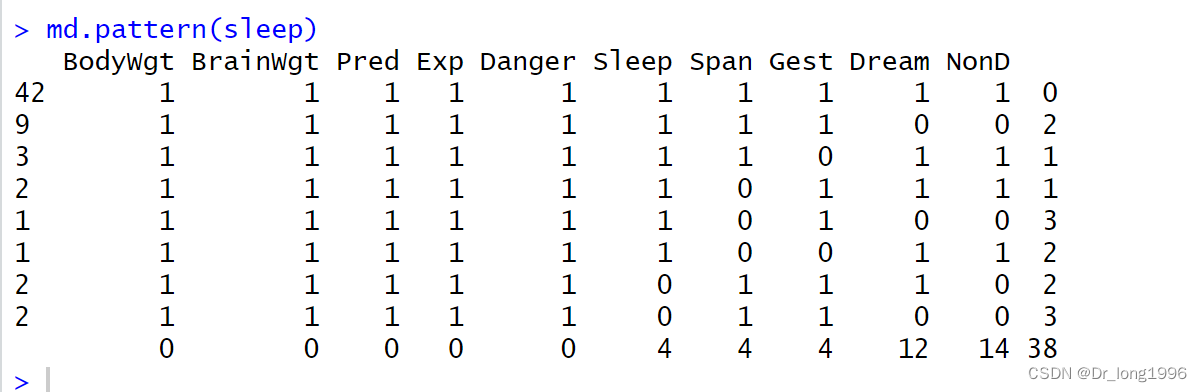
表中的1和0显示了缺失值模式:0表示变量的列中有缺失值,1则表示没有缺失值。第一行表述了无缺失值的模式(所有元素都为1)。第二行表述了“除了span之外无缺失值”的模式。第一列表示各缺失值模式的实例数,最后一列表示各模式中有缺失值的变量的个数。
理解:42个实例没有缺失值,仅2个实例缺失了Span,9个实例同时缺失了NonD和Dream的值。数据集包含了总共(42*0)+(2*1)+……+(1*3)=38个缺失值。最后一行给出了每个变量中缺失值的数目。
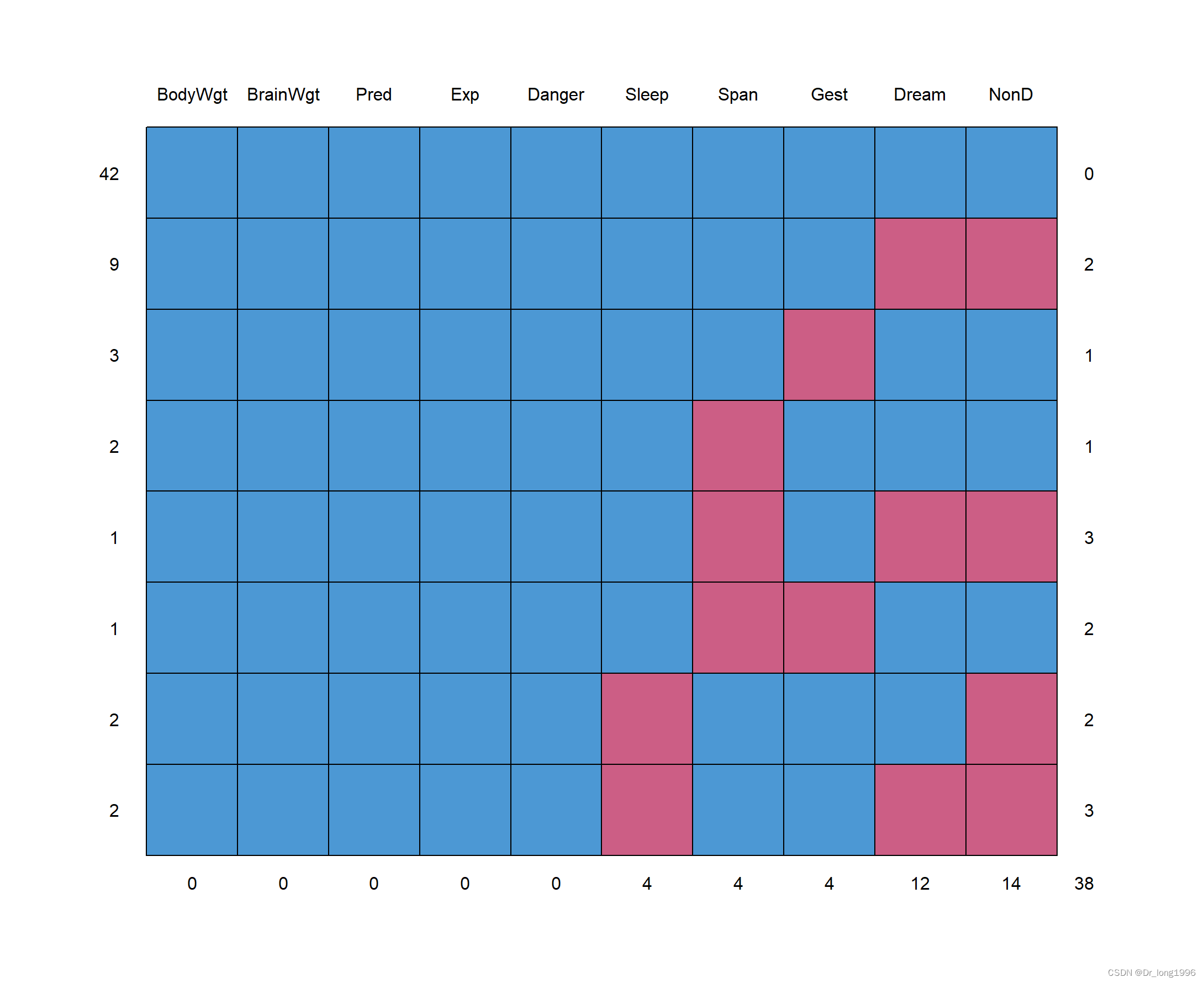
图形显示缺失值
aggr()函数不仅绘制每个变量的缺失值数据,还绘制了每个变量组合的缺失值数。
library("VIM")
aggr(sleep,prop=F,number=T)?prop设置是否显示比例,number显示是否数量。
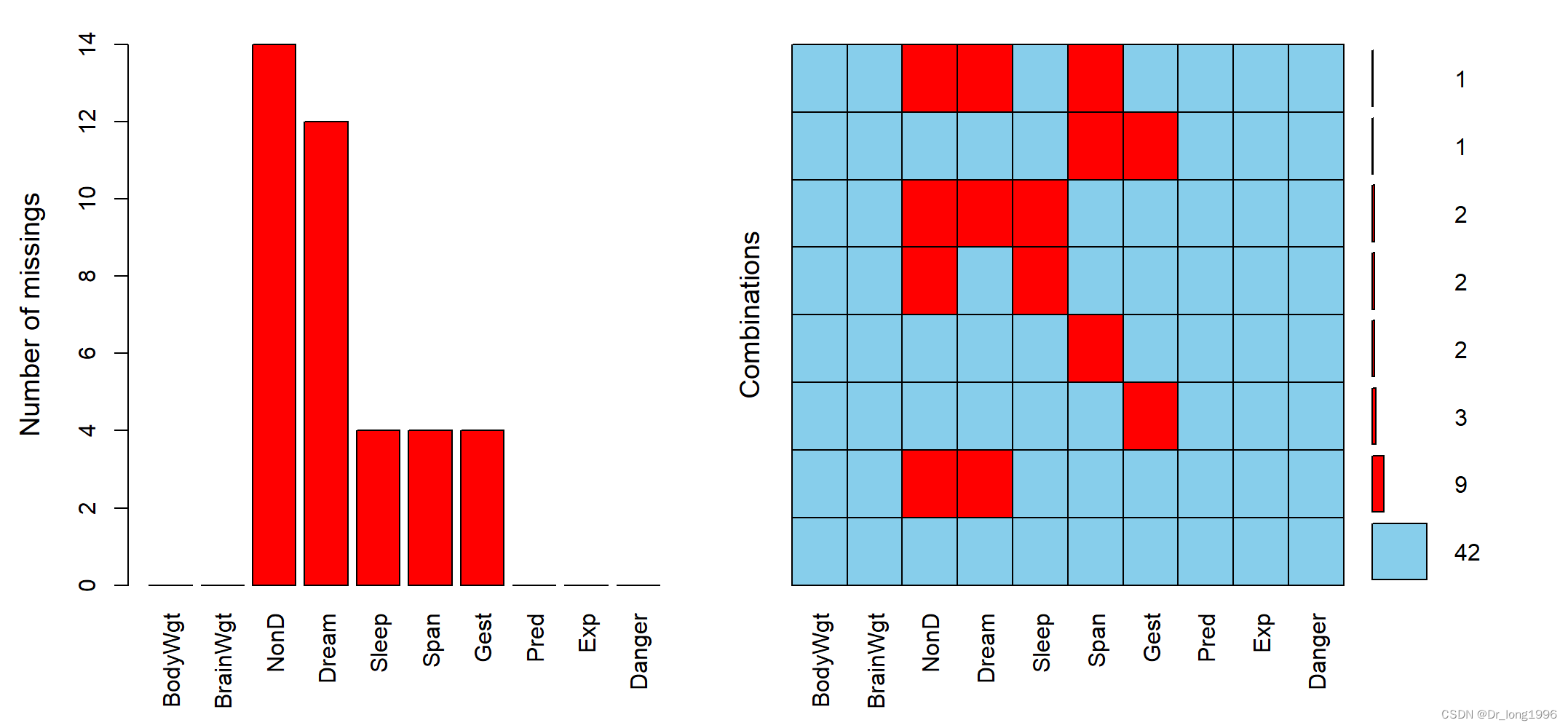
?可以看到NonD有最大的缺失值数,其中有2种动物同时缺失了NonD、Dream和Sleep的评分。
?
matrixplot()函数可生成展示每个实例数据的图形。
matrixplot(sleep)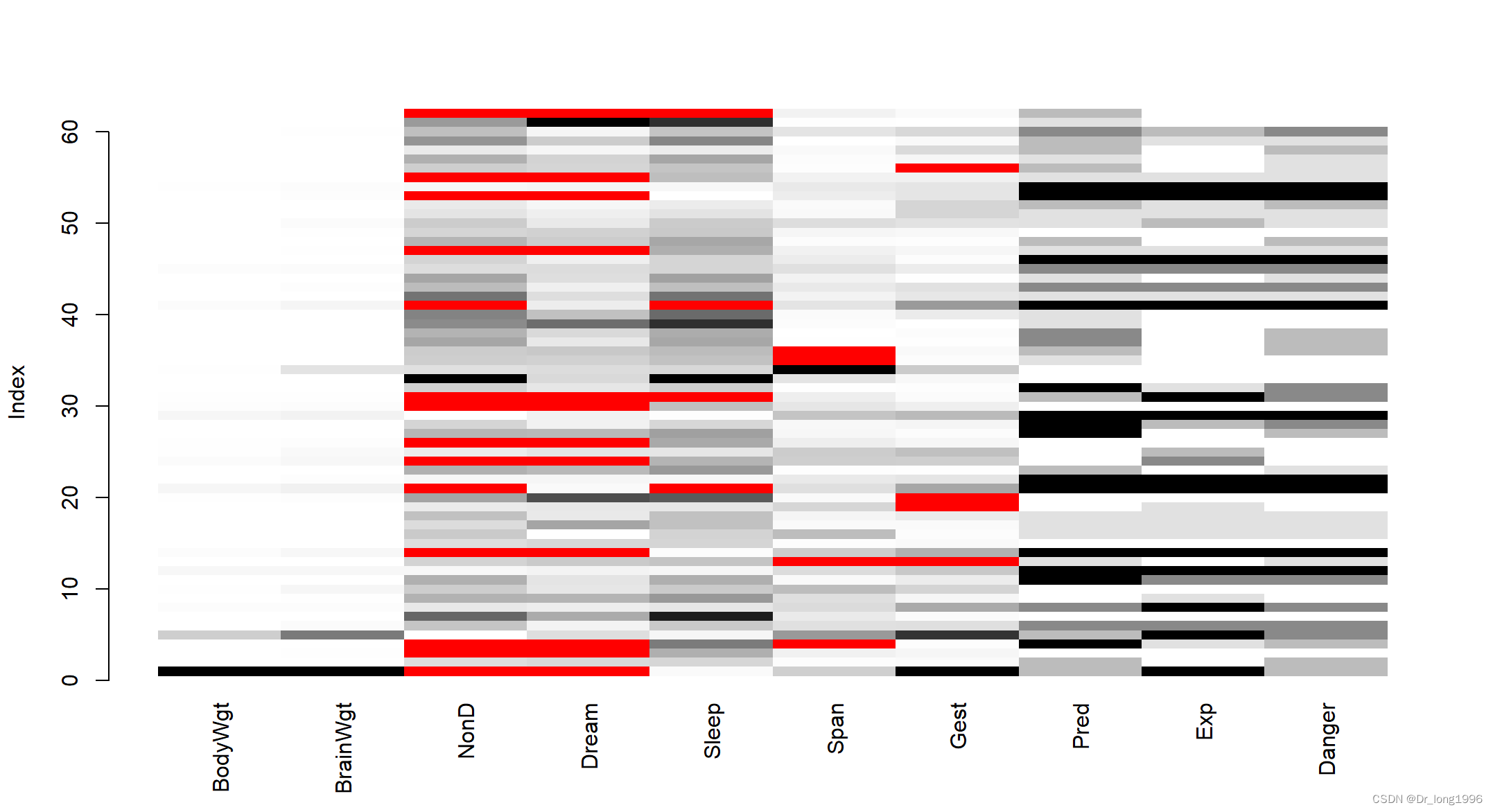
灰度表示大小:浅色表示值小,深色表示值大。默认缺失值为红色。
缺失数据的处理
行删除
将数据种存在缺失值的行全部删除,可以通过下面两个函数实现:
newdata <- mydata[complete.cases(mydata),]
newdata <- na.omit(mydata)行删除法假定数据为MCAR,也即是完整样本被认为是总体总随机抽取的子样本。删除行,减少了可用样本,这回导致统计效力降低。
多重插补
多重插补(MI)是一种基于重复模拟的处理缺失值的方法。对于复杂的缺失值问题,MI是最常用的方法,它将从一个包含缺失值的数据集中生成一组完整的数据集。每个模拟数据集中,缺失数据将用蒙特卡洛方法来填补。
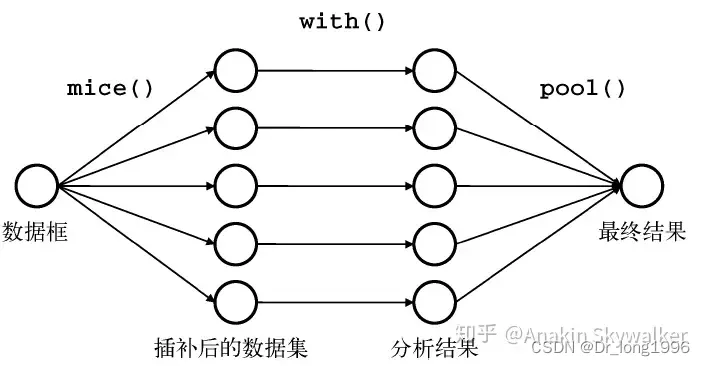
基本步骤:
mice()首先从一个包含缺失数据的数据集开始,返回包含多个(默认=5)完整数据集的对象。每个完整数据集都是通过对原始数据集中的缺失数据进行插补而生成的。由于插补有随机成分,因此插补后的每个完整数据集有所不同。
with()可依次对每个完整数据集应用统计模型(如广义线性模型)。
pool()将这些单独的分析结果整合为一组结果。最终模型的标准误和p值都将准确地反映出由于缺失值和多重插补而产生的不确定性。
基本格式:
library(mice) imp <- mice(data,m) fit <- with(imp,analysis) pooled <- pool(fit) summary(pooled)data是一个包含缺失值的矩阵或数据框
imp是一个包含m个插补数据集的列表对象,同时还含有完成插补过程的信息。默认m为5;
analysis是一个表达式对象,用来设定应用于m个插补数据集的统计分析方法。
fit是包含m个单独统计分析结果的列表对象;
pooled是一个包含这m个统计平均结果的列表对象。
imp <- mice(sleep,m=5,seed = 1234)
fit <- with(imp,lm(Dream~Span+Gest))
pooled <- pool(fit)
summary(pooled)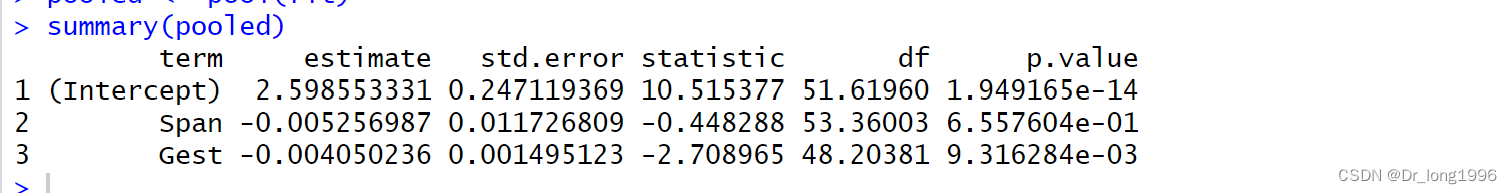
可以通过imp获取更多的插补信息。
imp
从数据可以看到Bodyweight,Brainwgt,Pred,Exp,Danger没有进行插补,其他的变量通过预测均值(pmm)进行了缺失值处理。
visitSequence从左到右展示了插补的变量,predictorMatrix展示进行插补过程的含有缺失值的变量,他们利用了数据集中其他变量的信息。其中行代表插补变量,列代表为插补提供信息的变量,1和0分别表示使用和未使用。
imp$imp$Dream
展示了在Dream变量中有缺失值的12个动物有5次插补值。
complete()函数可以查看m个插补数据集中任意一个
complete(imp,action = 3)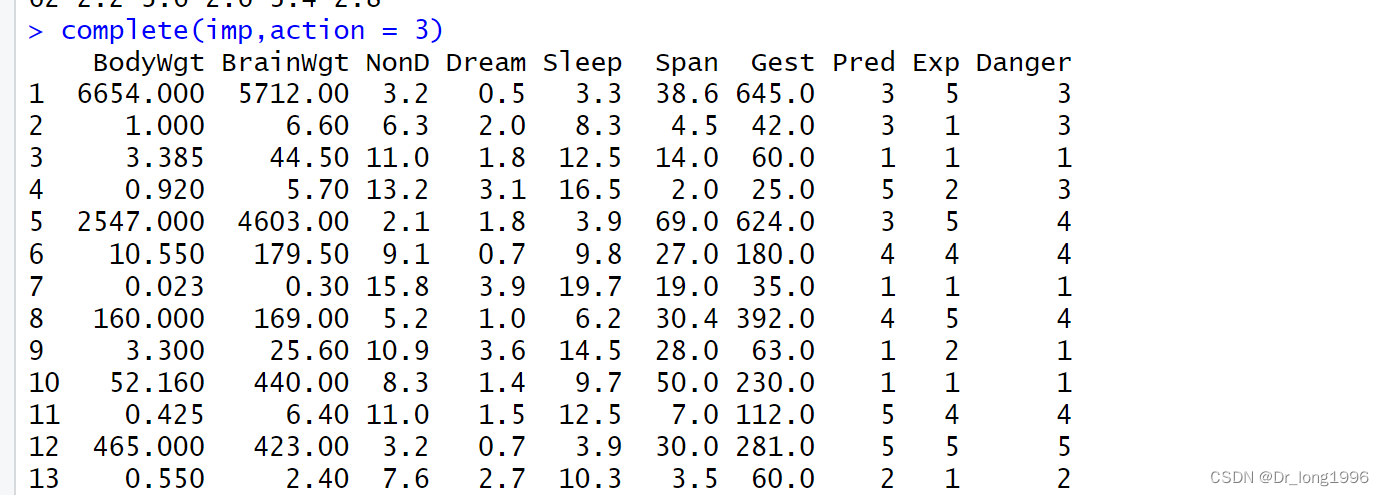
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:chenni525@qq.com进行投诉反馈,一经查实,立即删除!
- Python教程
- 深入理解 MySQL 中的 HAVING 关键字和聚合函数
- Qt之QChar编码(1)
- MyBatis入门基础篇
- 用Python脚本实现FFmpeg批量转换
- HPC平台搭建
- Windows 平台下安装与配置 MySQL 5.7.36
- VitulBox中Ubuntu虚拟机安装JAVA环境——备赛笔记——2024全国职业院校技能大赛“大数据应用开发”赛项
- 性能优化(CPU优化技术)-ARM Neon详细介绍
- Python字符串的编码和解码
- 操作系统期末复习 (南昌大学)
- WWAN(4GLTE无线网络模块)开启IPV6协议操作
- 双位置继电器DLS-5/2TH 额定电压:110VDC 触点形式:7开3闭 柜内安装
- Sectigo DV单域名证书买一年送一个月
- 大数据学习之Flink、Flink容错机制的注意事项