数据分析为何要学统计学(2)——如何估计总体概率分布
明确总体的概率分布类型及参数是进行数据分析的基础,这项工作称为分布推断与参数估计。在总体分布及其参数不明确的情况下,我们可以利用手头掌握的样本来完成这项工作。具体过程由以下步骤组成。
第一步,样本统计特性直观估计
我们采用Seaborn软件的histplot函数绘制核密度分布图(一种通过核密度函数KDE估计得出的近似概率密度曲线与频数分布直方图的合体)。参考代码如下:
import numpy as np
#输入样本数据
x=np.array([2.12906357, 0.72736725, 1.05152821, 0.48600398, 1.91963227,
1.62165678, 8.86319952, 0.24399412, 4.19883103, 2.80846683,
1.34644303, 0.35146917, 1.7575424 , 3.90572887, 1.07404978,
4.05247124, 0.65839571, 0.40166037, 2.03241598, 0.53592929])
import seaborn as sns
#绘制核密度分布图。kde=True会绘制概率密度曲线,否则只有直方图
sns.histplot(x,kde=True)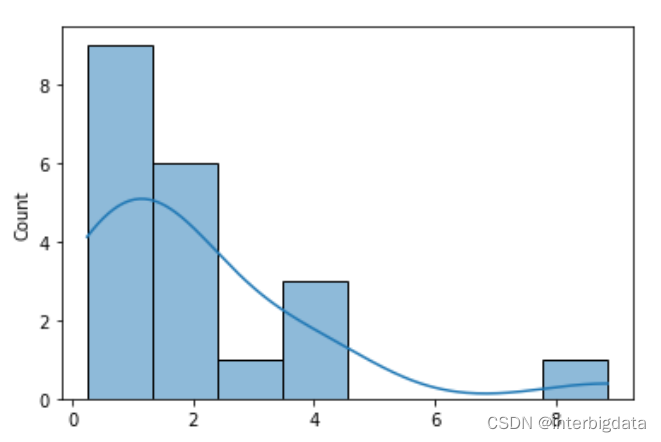
凸曲线(最高点两侧都有曲线)。即使实际概率密度曲线为??单调
曲线(如指数分布),其近似函数的曲线也是凸的,只不过??????一侧
曲线很长,而另一侧很短。
第二步,确定几个与之相近的候选概率分布(一般3个左右)。
从上图来看,可以根据概率密度曲线选择最相似的卡方分布、指数分布、伽玛分布。

第三步,分别拟合候选分布的参数,并使用拟合得出的分布参数检验每一个候选分布 。
代码如下:
import scipy.stats as stats
#构造候选分布集合
dists={'expon':stats.expon,'chi2':stats.chi2,'gamma':stats.gamma}
for dist in dists:
#拟合每一个分布,将参数存放于param。不同分布参数会不同,但最后两个分别为loc和scale
#loc和scale代表的含义因分布不同而异,如正态分布norm的情况,loc是均值scale是标准差
#具体可查看分布类的__doc__属性值中的相关介绍
param=dists[dist].fit(x)
#检验每一个分布。KS检测是分布推断的常用方法,第一个参数是样本,第二个参数是分布的累计分布函数,第三个是分布参数
test=stats.kstest(x,dists[dist].cdf,params)
print(dist,test.pvalue,params)第四步,选择p值(输出结果中的第一列值)最大的作为最终的分布(括号内是分布参数)
expon 0.9001 (0.016, 1.91) chi2 0.3800 (1.78, 0.016, 1.37) gamma 0.8080 (0.94, 0.016, 1.95)
?从以上数据可以看出,样本最可能是参数的指数分布。而事实上,原始样本确实是以
生成的随机数样本。因为用样本拟合总体总存在一定水平的误差,所以不能追求结果的完美性。
最后我们专门介绍正态分布检验及其参数估计。
正太分布有多种检验方法,分别针对不同的情况
Shapiro–Wilk test:适用于小样本(3-5000),检验函数为scipy的stats.shapiro,根据p值是否大于显著水平a进行判断。公认小样本效果最可靠。
Anderson–Darling test:适用于极小样本(8-26),检验函数为scipy的stats.anderson。可以检'norm, expon, logistic, gumbel,'gumbel_l, gumbel_r, extreme1, weibull_min分布,默认正态分布(norm)。该方法不输出p值,因此需要根据统计量是否小于显著水平对于的阈值来判断。
AndersonResult(statistic=0.8978770382274689, critical_values=array([0.527, 0.6 , 0.719, 0.839, 0.998 ]), significance_level=array([15. , 10. , 5. , 2.5, 1. ]), fit_result= params: FitParams(loc=1.9203141072704806, scale=0.2784872286328154, success: True, message: '`anderson` successfully fit the distribution to the data.')
在该结果中,统计量小于1%显著水平的阈值,所以在该水平下才可以接受服从正态分布,其他显著水平下都拒绝接受服从正态分布的假设。
D'Agostino's K-squared test (Skewness-Kurtosis test):适用于8个以上的样本,样本容量越大越有效。公认大样本最有效。检验函数为scipy的stats.normaltest,根据p值是否大于显著水平a进行判断。
当样本通过正太分布检验后,可以使用stats.norm.fit函数获得分布参数。
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:chenni525@qq.com进行投诉反馈,一经查实,立即删除!