【论文阅读笔记】两篇完整模态脑瘤分割
两篇完整模态脑瘤分割论文,都是使用Transformer,没有什么特别的特色,也没有开源代码,因此只是简单记录一下。
3D CATBraTS: Channel attention transformer for brain tumour semantic segmentation
El Badaoui R, Coll E B, Psarrou A, et al. 3D CATBraTS: Channel attention transformer for brain tumour semantic segmentation[C]//2023 IEEE 36th International Symposium on Computer-Based Medical Systems (CBMS). IEEE, 2023: 489-494.
关键贡献:
- 提出了一种新型网络,使用ViT进行多模态MRI上的脑肿瘤3D分割,命名为3D CATBraTS。
- 提出了一种修改的CNN编码器架构,使用残差块和通道注意模块。
- 通过评估表明,该模型在BraTS 2021数据集的验证阶段,性能优于顶级模型,如Swin UNETR和SegResNet。
研究方法:

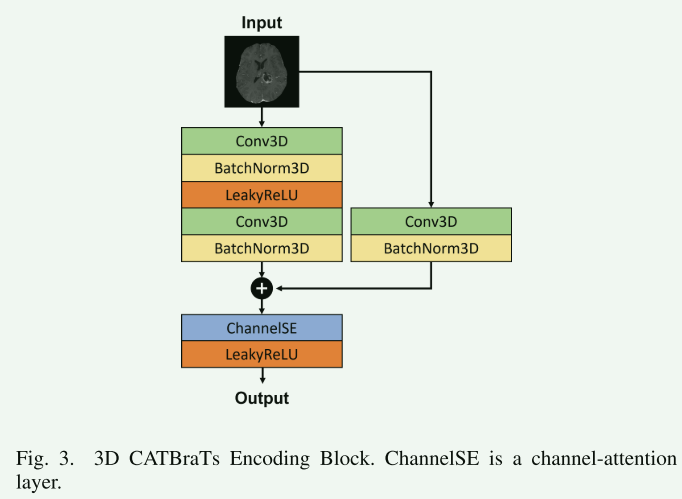
- 使用Swin变换器和通道注意残差块修改Swin UNETR模型的编码器。
- 输入网络的是使用四种不同MRI模态获取的脑MRI扫描。
- 在下采样阶段,编码器包含五个修改的Res块,增强了挤压和激励通道注意层。
- 使用跳跃连接和上采样阶段还原图像的原始分辨率。
数据集与实验:
- 使用BraTS 2021数据集进行训练和评估,该数据集包括1251个脑肿瘤案例的多参数MRI扫描。
- 进行了定量分析,使用Dice相似系数(DSC)作为评价指标。
结果与讨论:
- 3D CATBraTS在DSC方面表现优于Swin UNETR和SegResNet。
- 该方法在ET(增强肿瘤)、TC(肿瘤核心)和WT(整个肿瘤)的DSC得分分别为0.826、0.799和0.876,而Swin UNETR的分数分别为0.780、0.731和0.817。
总结: 论文提出的3D CATBraTS基于CNN编码器-解码器架构的Swin-T模型,在多模态MRI上进行脑肿瘤的3D分割。通过在编码器上的通道注意Res块的修改,该模型在肿瘤分割准确度上显示出更高的性能。在BraTS 2021数据集上的验证表明,该方法超越了当前最先进的方法。
Automated multi-modal Transformer network (AMTNet) for 3D medical images segmentation
Zheng S, Tan J, Jiang C, et al. Automated multi-modal Transformer network (AMTNet) for 3D medical images segmentation[J]. Physics in Medicine & Biology, 2023, 68(2): 025014.
这篇论文介绍了一种新型的自动化多模态Transformer网络,称为AMTNet,用于三维医学图像的分割。该研究的主要目的是将Transformer应用于多模态医学图像分割。过去几年,基于卷积神经网络的方法主导了医学图像分割领域,但这些方法难以表示长距离依赖关系。最近,Transformer因其自注意机制和在图像上编码长距离依赖性而在计算机视觉中表现出色,并已成功应用于医学图像分割。
AMTNet是一个U形网络结构,对特征编码、融合和解码部分进行了有效的改进。编码部分包括三维嵌入、三维多模态Transformer和三维共学习下采样模块。对称地,解码部分包括三维Transformer块、上采样块和三维扩展块。此外,设计了基于Transformer的自适应通道交错Transformer特征融合模块,以充分融合不同模态的特征。这种方法利用了Transformer和CNN的优势,有效地结合了全局和局部特征。
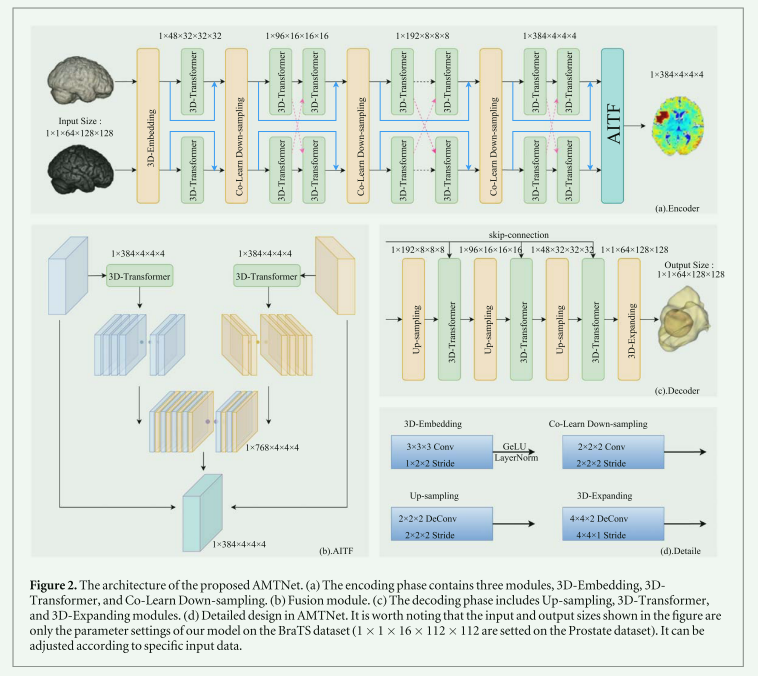
研究中使用了前列腺数据集和BraTS2021数据集进行全面的实验分析。结果表明,该方法在这两个数据集上分别实现了平均DSC(Dice Similarity Coefficient)0.907和0.851,显著优于现有的分割网络。此外,该研究还与其他先进的多模态分割方法进行了比较,如MFNet、TCSM、MAML、WNet和MSAM,并使用5折交叉验证进行评估。结果显示,AMTNet在DSC、Jaccard相似性系数、相对体积差异和95% Hausdorff距离等评估指标上表现优越。
实验过程中使用了Python 3.6、PyTorch 1.8.1和Ubuntu 16.04,所有训练过程均在单个12 GB NVIDIA 2080 Ti GPU上进行。数据预处理包括重采样、图像旋转、缩放、添加高斯噪声、高斯模糊、调整亮度和对比度等操作。
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:chenni525@qq.com进行投诉反馈,一经查实,立即删除!