【project】estimate Aβ-PET pattern
1.17
1.16
1.14
- 写一个函数,输入是每个文件的地址,然后能做这一系列的操作
- 用AFM0095进行bbr的配准
- 方法一,间接配准,frmi先到str,再到mni(str2fmri后再fmri2str)
-
fmri2str
只需要dof 6,6个自由度进行刚性变换
在AFM0367上str2fmri线性要比bbr配的好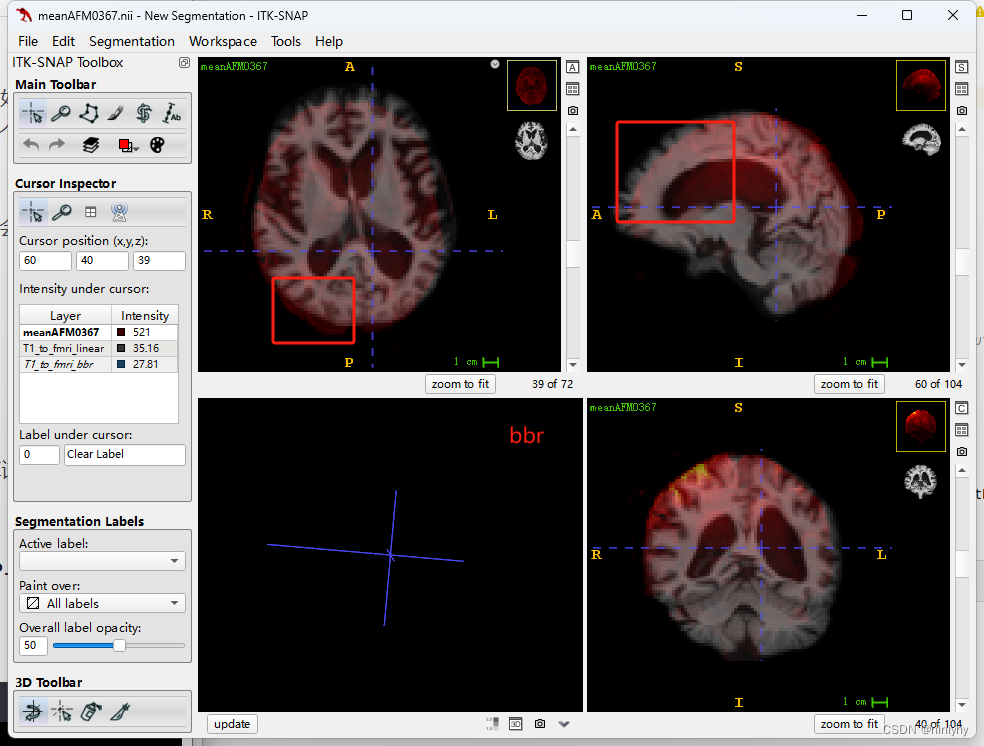
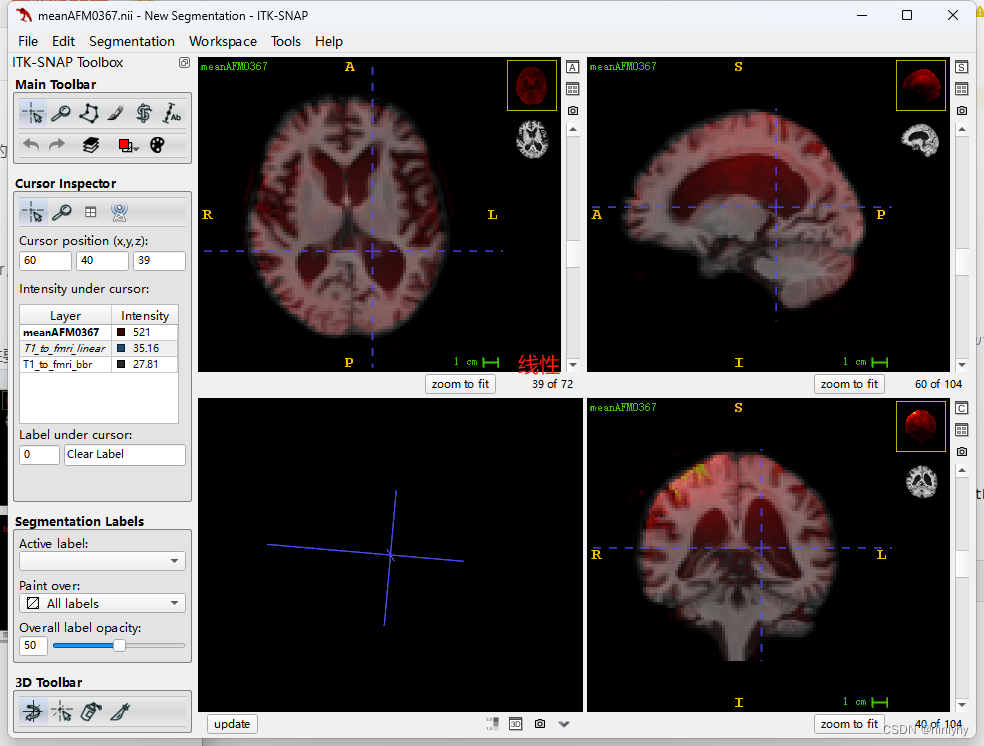
反用逆矩阵将fmri2str,也是线性的更好(AFM0367)
同AFM070也是线性更好(dof=6)
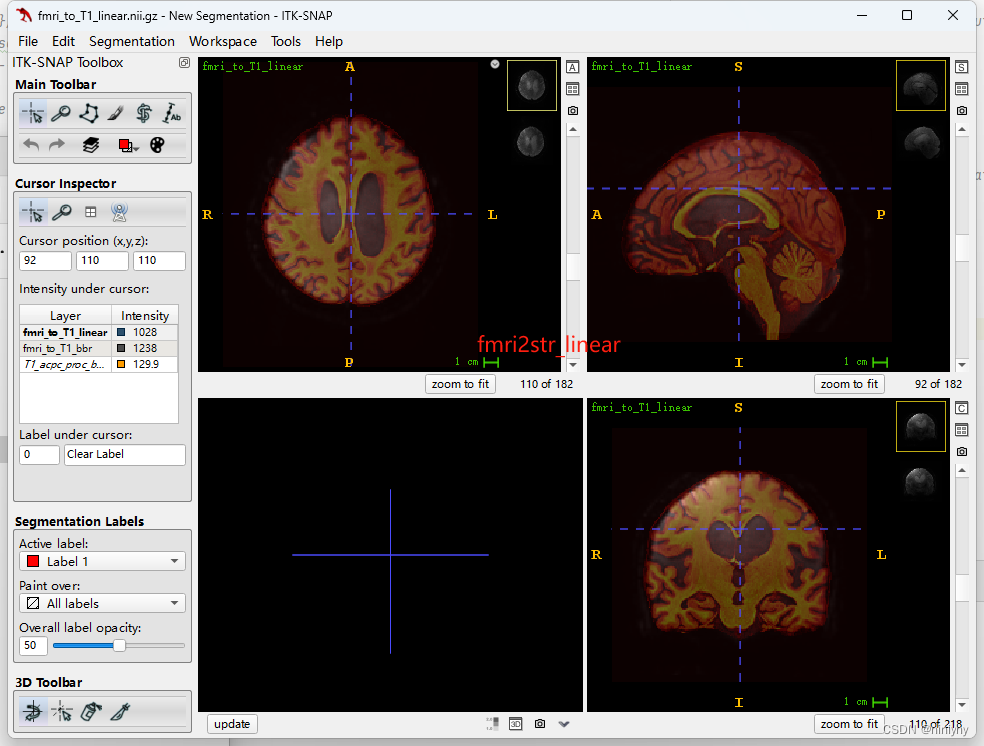
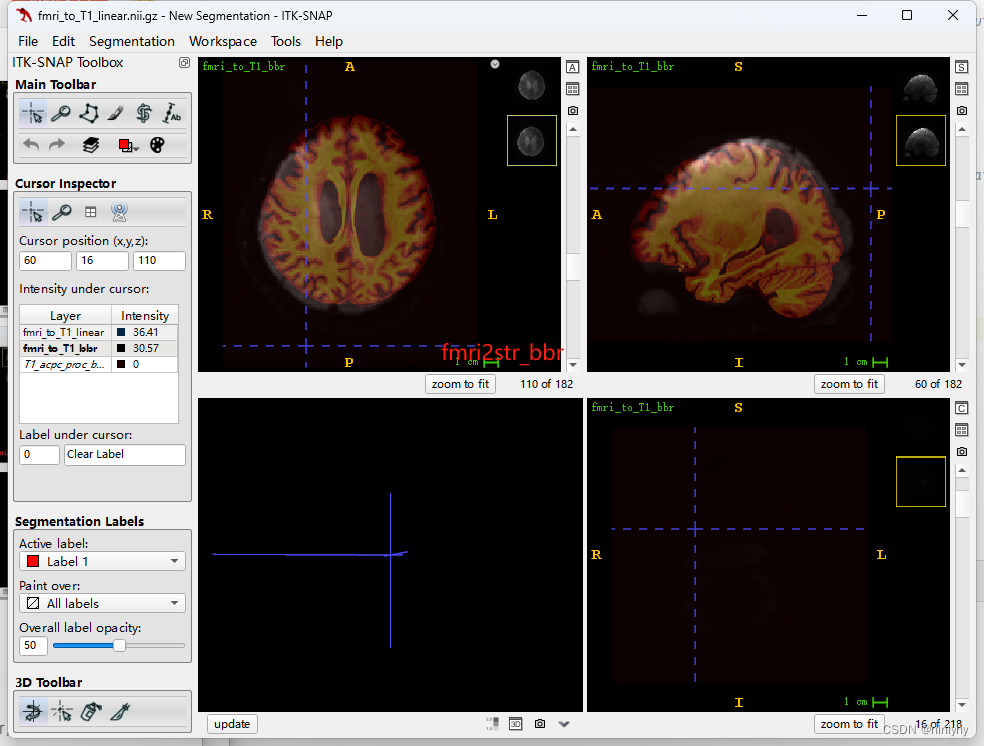
-
t12mni
-
fmri2mni
-
- 方法二,间接配准,frmi先到str,再到mni(直接fmri2str)
- fmri2str
在subjectAFM0224(非常坏的情况下)6个自由度要比12个好
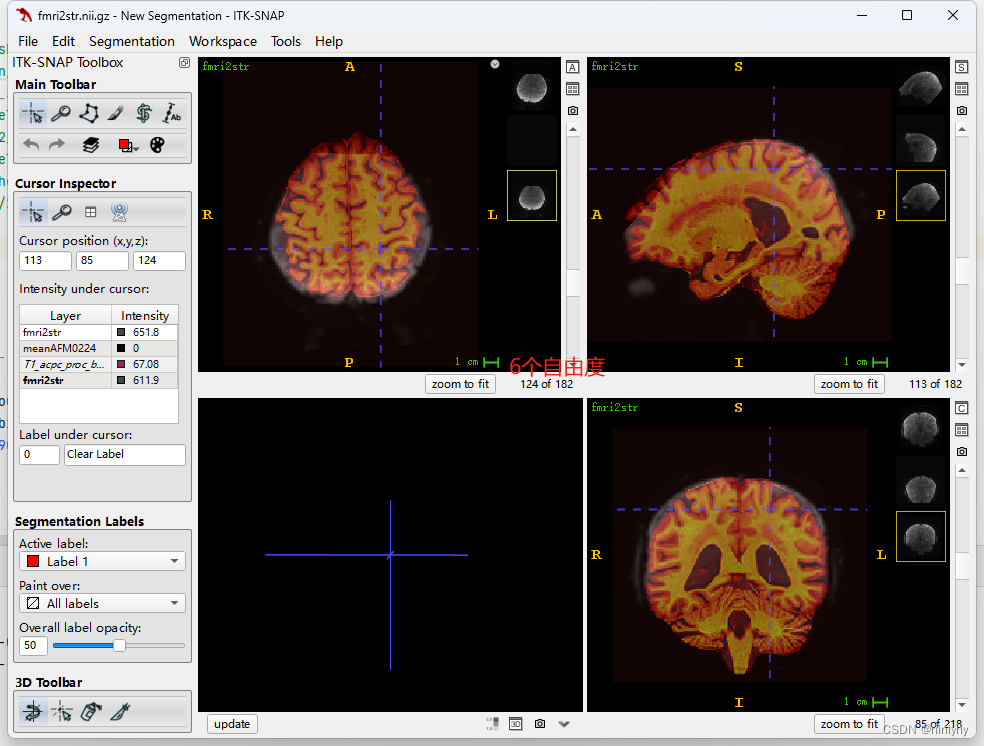
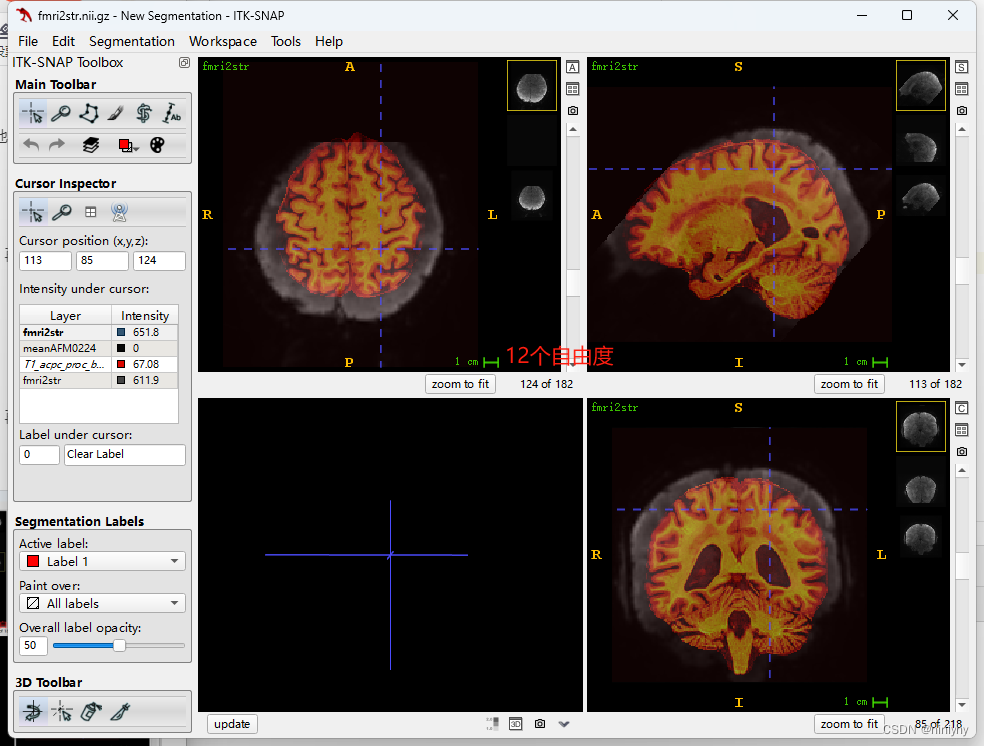
但是在AFM0367(Good)上6个自由度要比12个差
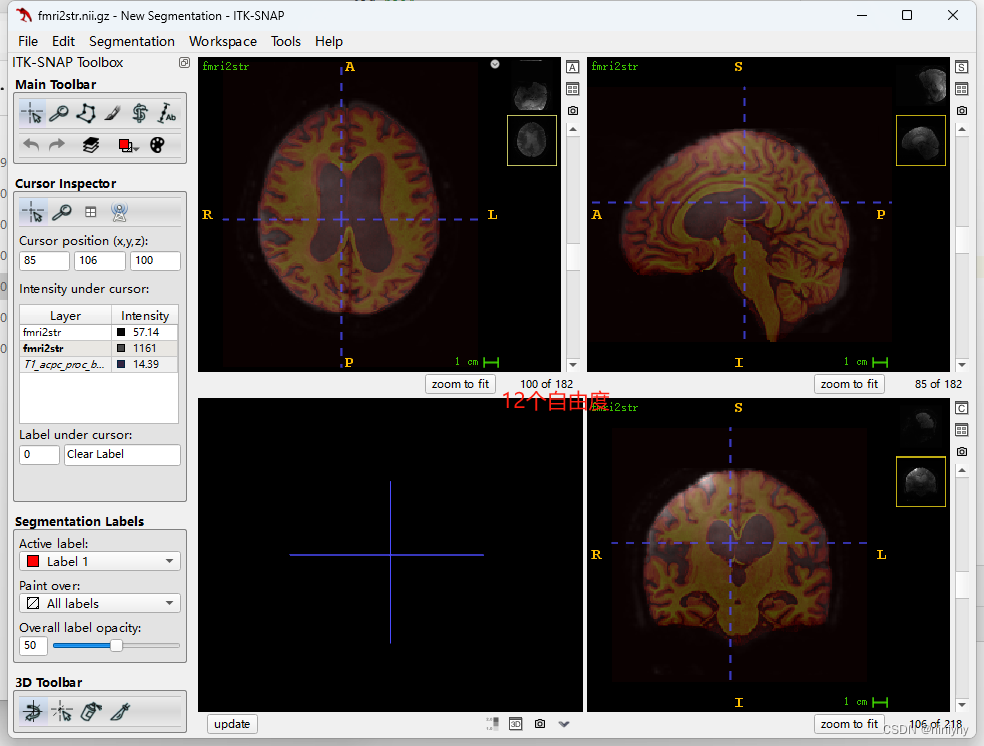
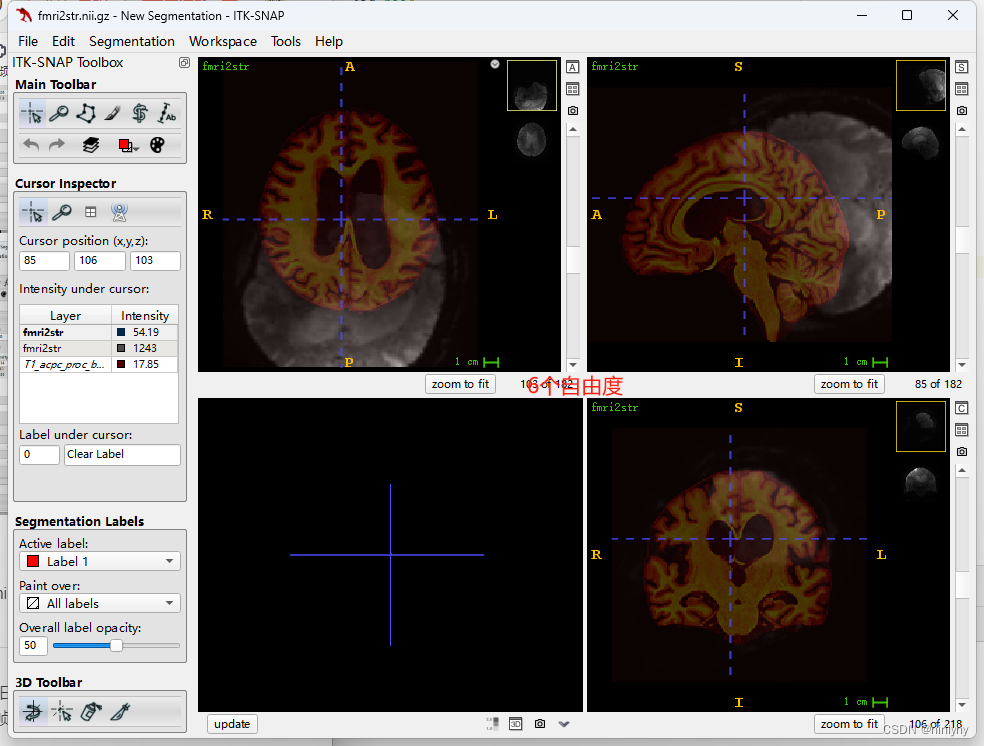
- t12mni 用的是ants的多模态配准,就差将矩阵应用在fmri上进行转换了
- fmri2mni
- fmri2str
- 查看merg的日志,看哪些没有被merge然后手动查看是不是没有单帧配准
- 重新配AFM0006
- fmri2str
- t12mni
- fri2mni
线性插值要比最近邻看起来平滑
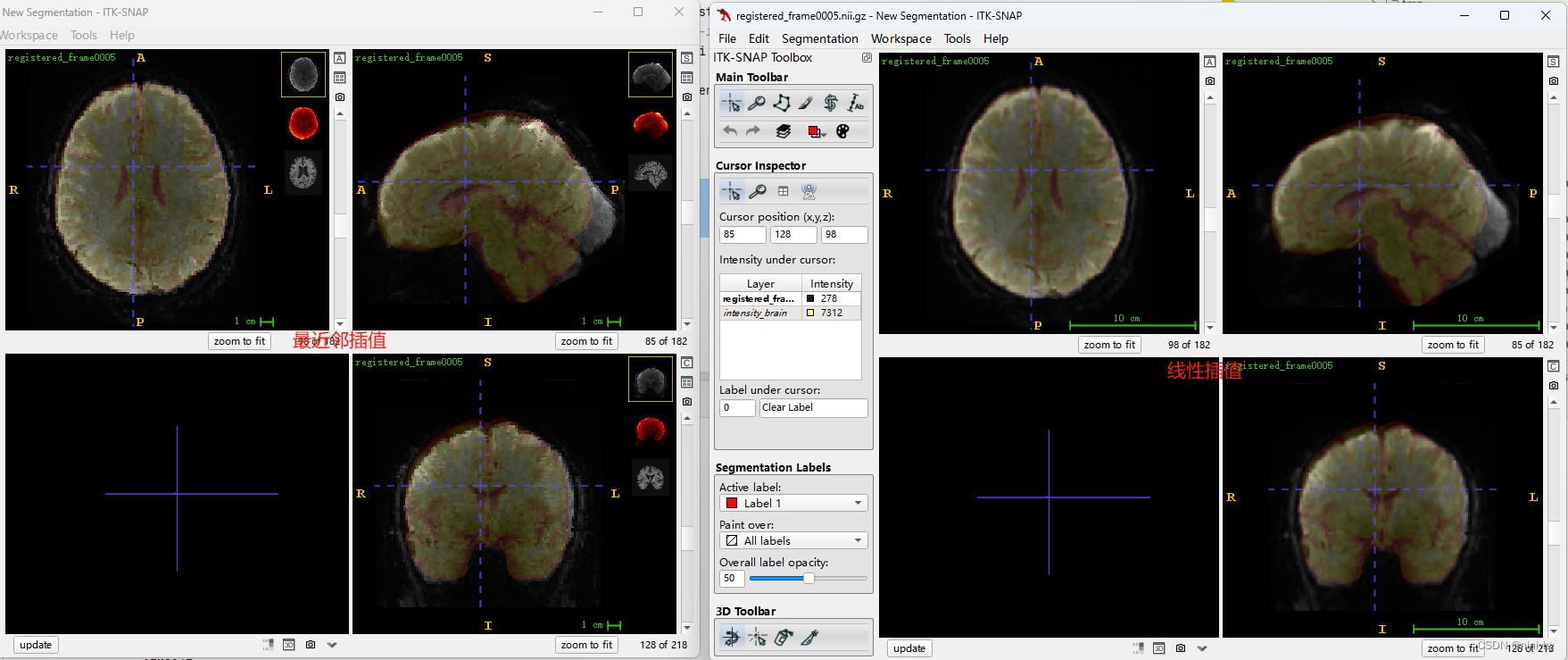
333的右上角变形了
- merge 是将原来线性插值得到的merge在一起
- fmri2MNI是使用最近邻插值
但是用最近邻插值配的也不准【只是针对AFM0006】
用线性插值得到的结果不对,先将mergekill掉
AFM0208
AFM0120
AFM0124
AFM0122
AFM0203
AFM0121
AFM0278
AFM0204
AFM0092
AFM0492
AFM0202
AFM0123
657 669
这些是没有得到配准的文件,没有fmri
rAFM0062MNI 到后面的时间点看起来不好的可能是因为没有merge好,看每一帧的qc图没有问题
-
/public_bme/data/yuanhy/Pet_center/Huashan/meanfMRI2MNIQC_ants个体的meanfmri到个体的T1 tissue去看看哪个好配,哪个不好配 -
/public_bme/data/yuanhy/Pet_center/Huashan/fMRI2MNIQC_ants每一帧配准后的到标准空间的T1 tissue上去看看配的好不好;
- 1.15-10:49目前看来,fmri每一帧配准到MNI上的维度没有问题
- 【1.15-10:49】等merge完就可以画每一帧
-
/public_bme/data/yuanhy/Pet_center/Huashan/meanfmri2strQC_antsfmri2T1,
需要查看fmri与T1的差异,直接用fmri与T1 的Tissue放在一块看,找一个比较简单的,一个比较难的去用bbr配一下
/public_bme/data/yuanhy/Pet_center/Huashan/meanfmri2tissueQC_ants 这个地址就是
check data
AFM0084d,AFM0385质量不好
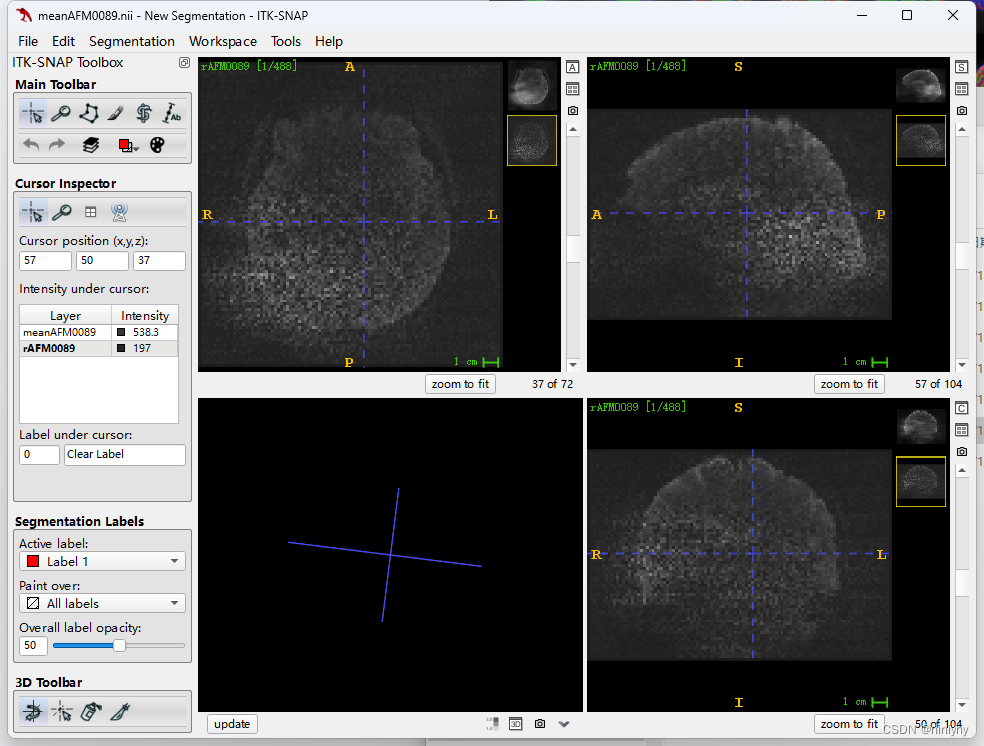
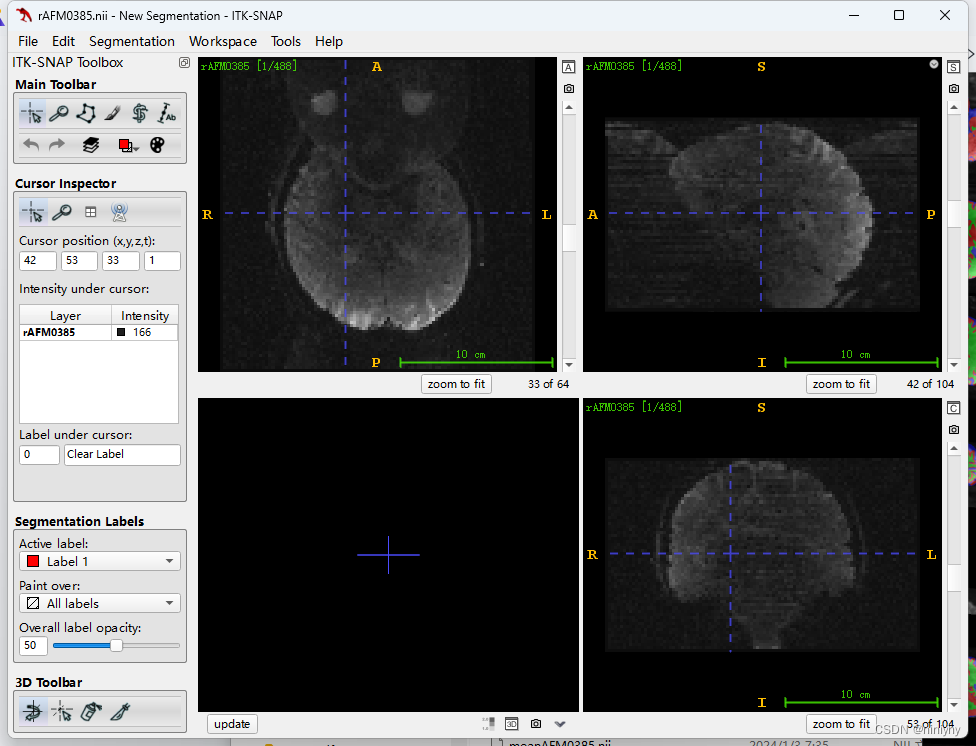
1.13
还要做的
- merge 正在测试
- 画出每一帧配准后的QC图,之前的不对 也就是把这个路径填满/public_bme/data/yuanhy/Pet_center/Huashan/fMRI2MNIQC_ants
- 正在重新跑每一帧的配准文件,因为之前的第0镇有4多个数据点,不对
/public_bme/data/yuanhy/Pet_center/Huashan/meanf2MNIQC_ants meanfMRI配准到MNI空间的QCmap,为了方便查看,将标准的T1tissue作为mask【基于ants】这个需要重新生成,因为插值的方法变了×
/public_bme/data/yuanhy/Pet_center/Huashan/meanfmri2tissueQC_ants meanfMRI配准到T1上的QCmap, 为了方便查看,将T1 tissue 作为mask【基于ants】 得到了,这个也不对,改变了插值方法×
/public_bme/data/yuanhy/Pet_center/Huashan/fMRI2MNIQC_ants fmri每一帧配准到标准空间的QCmap,为了方便查看,将标准的T1tissue作为mask【基于ants】
- merge的代码正在重跑,之前的有9百多帧,将没有配准的的也合并到了一起去,第0帧的问题
之前配的fmri到T1有一个问题,就是插值应该是线性插值而不是最近邻
1.12
-
拿到每一个mean与T1的map找一个差别比较大的和差别比较小的分别配一下
fmri和T1不一样大怎么合到一起 fmri更小,把他放到中间,其他的作为0 -
得到每一帧的QC,一个一个去看,看看配的怎么样
-
merge,与上面一步同时进行 已经在跑了
-
通过QC查看AFM0019为什么配的不好,应该是T12MNI的时候不太好
正在跑fmri2mni ,没有加merge步骤
分割的差值用最近邻,图像插值用线性的,之前我的配准都是用最近邻(fmri配到t1)
全部需要重新来过 没有做错
- [ ] T1和Tissue一起配准到MNI空间
- [ ] fmri利用T1和Tissue一起配准
海峰给的代码
echo "Running BBR"
flirt -ref ${T1_bet} -in ${OutputDC}/SBRefAP_dc_jac.nii.gz
-dof ${dof} -cost bbr -wmseg ${T1_WM} -init ${OutputF2S}/${voutAP}_init.mat
-applyxfm -omat ${OutputF2S}/${voutAP}.mat -out ${OutputF2S}/${voutAP} -schedule ${FSLDIR}/etc/flirtsch/bbr.sch
1.11
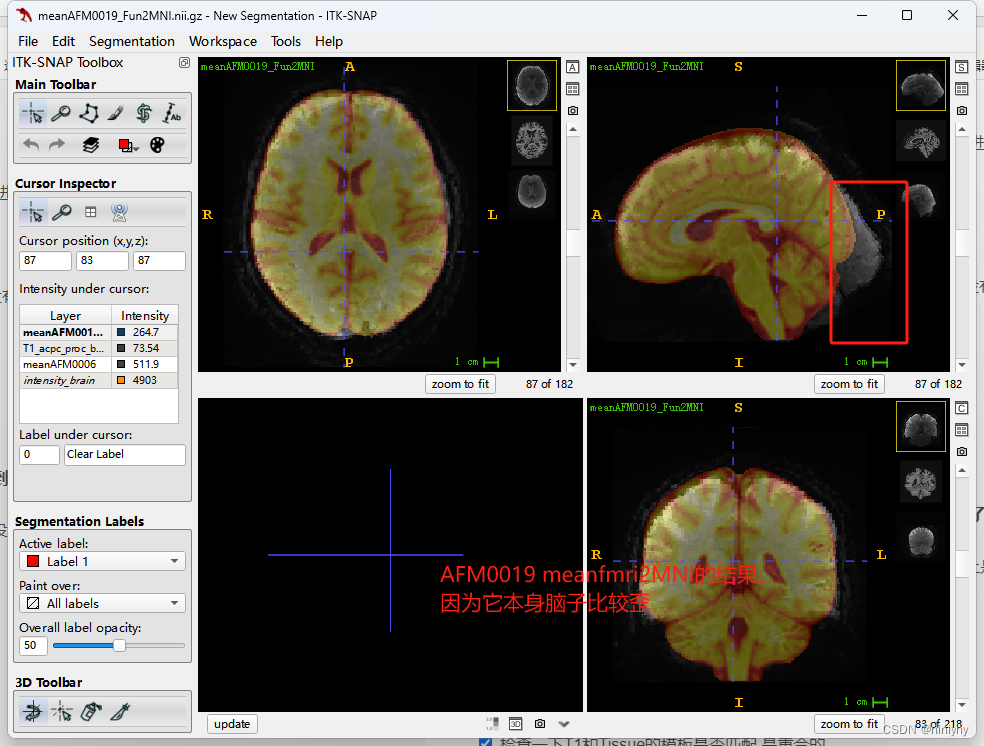
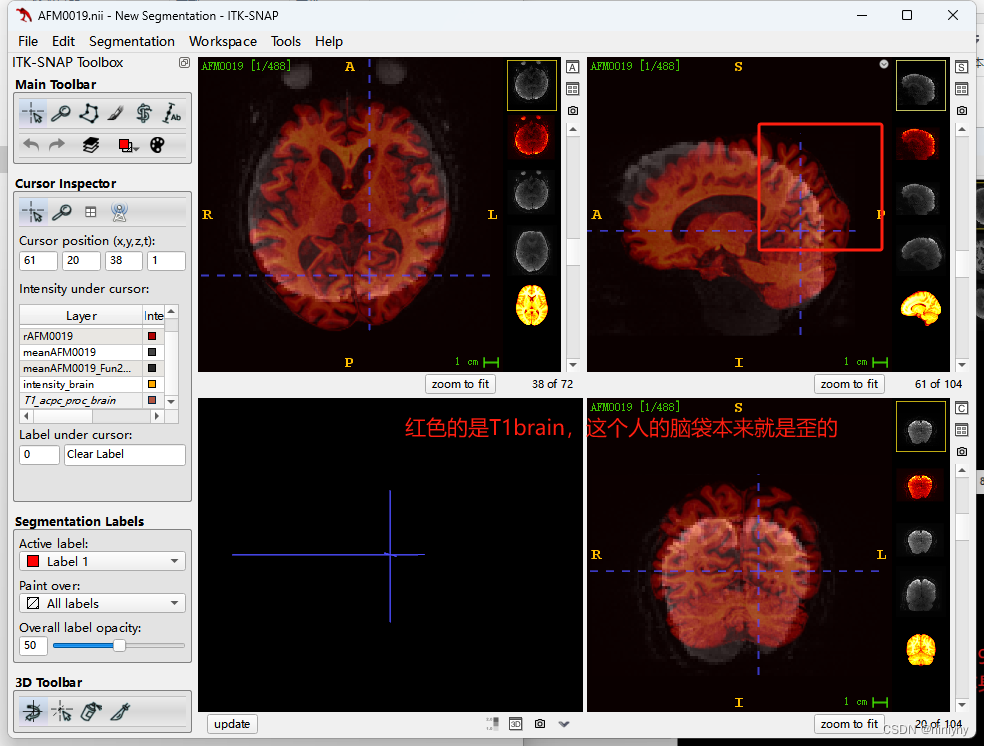
上面这个图是将AFM0019 的T1 mask上AFM0019 的原始fmri,可以发现这个人的脑袋本来就是歪的。
原来的可视化的脚本就需要更改,因为现在的图像是4D的,需要抽一些进行查看
-
调整一下结构,把fMRI2MNI放在2_shellssubject目录下
-[ ] 跑一下fmri2T1brain的 -
生成AFM006 和AFM009 的QCdata
-
做一下AFM0019 看一下是不是原来的T1 Tissue配准有问题,如果没有问题可以直接批处理fmri2MNI,同时跑bbr处理fmri到T1的配准
- 配mean没有问题 除了脑子是歪的配不上去没有大问题
- 配所有的time point fmri
-
删除不需要的文件
-
配一个fmri所有time_point
-
warp的时候 fmri脑子会变得很大,不确定是不是 -r没选好,成功配到了T1MNI上面,而且比之前配的要好
排除一下:
√使用T1配到MNI+Tissue配到Tissue的MNI,都warp到T1和Tissue上是没有问题的 -
用T1 做中间
T1配准到标准空间没有问题 -
用Tissue做中间
单独使用Tissue配准到Tissue的MNI是不对的,越配越差 -
检查一下T1和Tissue的模板是否匹配 是重合的
1.9
集群上:AFM0600 6
AFM0006 16
AFM0326 6
AFM0181 6
AFM0462 6
本地:
AFM0594
AFM0600
AFM0682
AFM0190 5
AFM0600 6
AFM0682 11
1.8
画图要记得该透明度看配的好不好
bbr配准,fsl
Boundary-Based Registration (BBR) 是一种用于图像配准的方法,特别是在脑部 MRI 图像处理中。BBR 通常用于对比不同模态的图像(例如,将结构 MRI 和功能 MRI 图像对齐),它依赖于找到脑组织边界(通常是灰质和白质之间的边界)上的对应点。
在 FSL (FMRIB Software Library) 中,BBR 是通过 flirt 命令行工具实现的,具体是通过 -B 选项来启用 BBR 功能。BBR 配准通常需要一个权重图像,这通常是从功能 MRI 中提取的,以及一个初始的对齐估计,这通常是通过线性配准得到的。
一个基本的 BBR 配准命令可能看起来像这样:
flirt -in func.nii.gz -ref struct.nii.gz -out func_to_struct.nii.gz -omat func_to_struct.mat -cost bbr -wmseg wmseg.nii.gz
在这个例子中:
-in func.nii.gz是要配准的图像(例如,功能 MRI)。-ref struct.nii.gz是参考图像(例如,结构 MRI)。-out func_to_struct.nii.gz是配准后的输出图像。-omat func_to_struct.mat是输出的仿射变换矩阵。-cost bbr指定了使用 BBR 作为成本函数。-wmseg wmseg.nii.gz是白质分割图像,用于帮助找到灰质和白质之间的边界。
在实践中,您可能还需要提供额外的选项,例如设置 BBR 用于配准的点的数目、设置搜索范围以及提供初始配准矩阵等。同时,实施 BBR 之前,通常需要进行一些预处理步骤,如脑提取(brain extraction)、线性配准(linear registration)和白质分割(white matter segmentation)。
- 正在跑原来的fmri2MNI 跑完了但是错了,4d的要拆成一帧一帧跑

- fmri使用多模态配准:T1 以及T1 template,Tissue 以及Tissue Template,使用mean获得转换,但是还是要warp到全部的bold signal上
- 怎么抠出来每个roi的bold signal,将fmri配到各个脑区的template上,然后每个脑区是一个值,得到每一个脑区的bold signal,MNI配到fMRI上,要记得resample
- 将区分左右脑的tissue改成一个值
“D:\yhy\data\template\intensity-tissue_hemisphere.nii.gz” 使用这个区分左右脑的tissue template去配准,需要先转换一下
先copy数据
- tissue转换
fslmaths mrbrain/intensity-tissue_hemisphere.nii.gz -thr 1 -uthr 2 tmp/gm_msk.nii.gz fslmaths mrbrain/intensity-tissue_hemisphere.nii.gz -thr 3 -uthr 4 tmp/wm_msk.nii.gz fslmaths mrbrain/intensity-tissue_hemisphere.nii.gz -thr 5 tmp/csf_msk.nii.gz fslmaths tmp/gm_mask.nii.gz -add tmp/wm_mask.nii.gz -add tmp/csf_mask.nii.gz tmp/tissue.nii.gz - ants多个模态配准
antsRegistrationSyNQuick.sh -d 3 -m $t1_sub -f $t1_tmp -m $t2_sub -f $t2_tmp -m $tiss_sub -f $tiss_tmp -o $proc_folder/tmp_odf_reg/sub2tem_b -n 50 -t s # -m $sub -f $temp
1-7
使用T1带脑壳和不带脑壳的一起去配fMRI
把分左右脑的tissue 变成灰质白质脑积液
bbr配准
用不同ROI atlas 扣脑网络
模版大小为 193, 229, 193
他的大小为227, 272
使用刚性变换进行配准后只有656个 subject
/public_bme/data/yuanhy/Pet_center/Huashan/fMRI2MNI
-t: transform type (default = ‘s’)
t: translation (1 stage)
r: rigid (1 stage)
a: rigid + affine (2 stages)
s: rigid + affine + deformable syn (3 stages)
sr: rigid + deformable syn (2 stages)
so: deformable syn only (1 stage)
b: rigid + affine + deformable b-spline syn (3 stages)
br: rigid + deformable b-spline syn (2 stages)
bo: deformable b-spline syn only (1 stage)
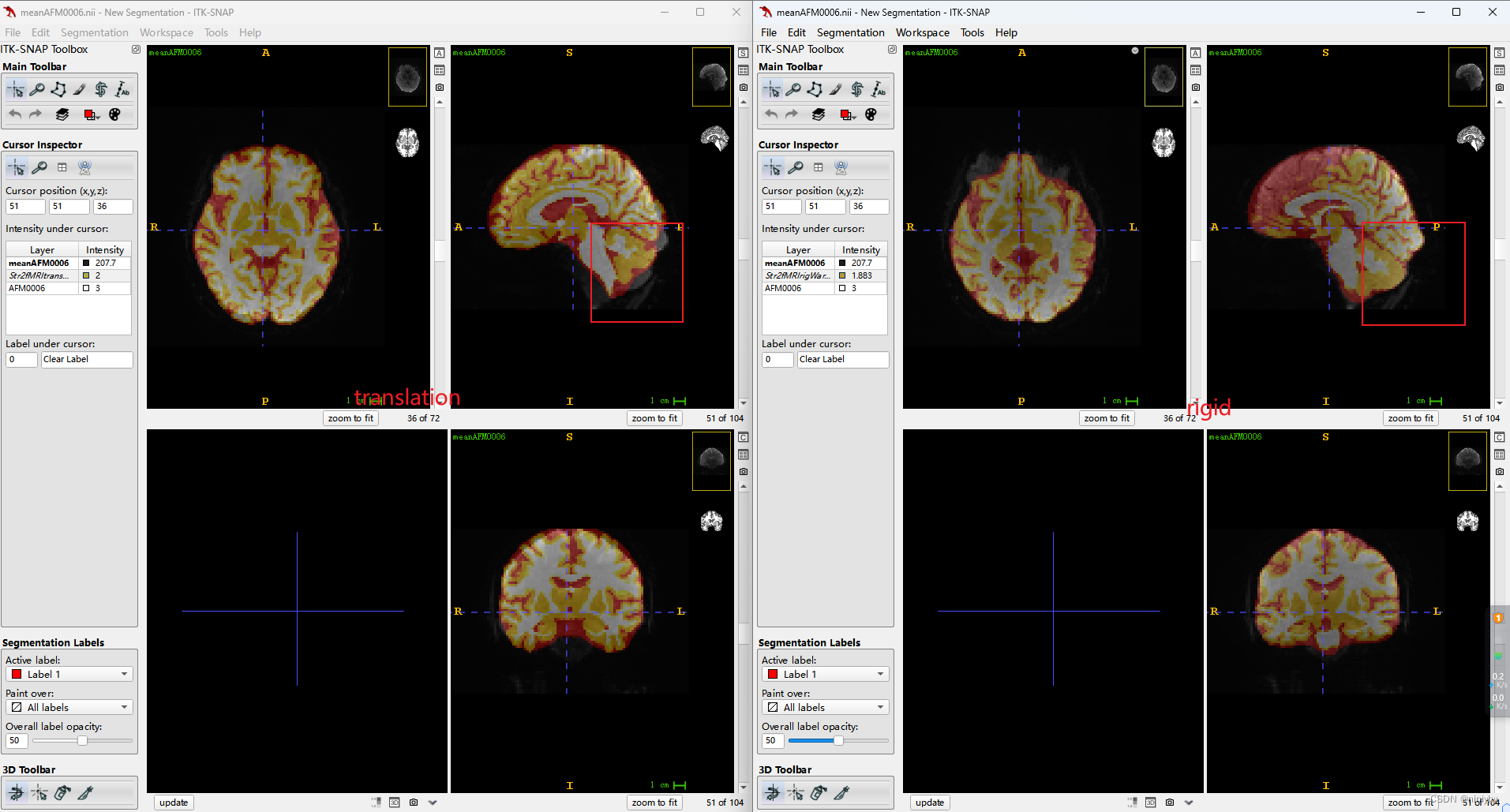
rigid 要配的比translation 好,trans有部分都没有盖上
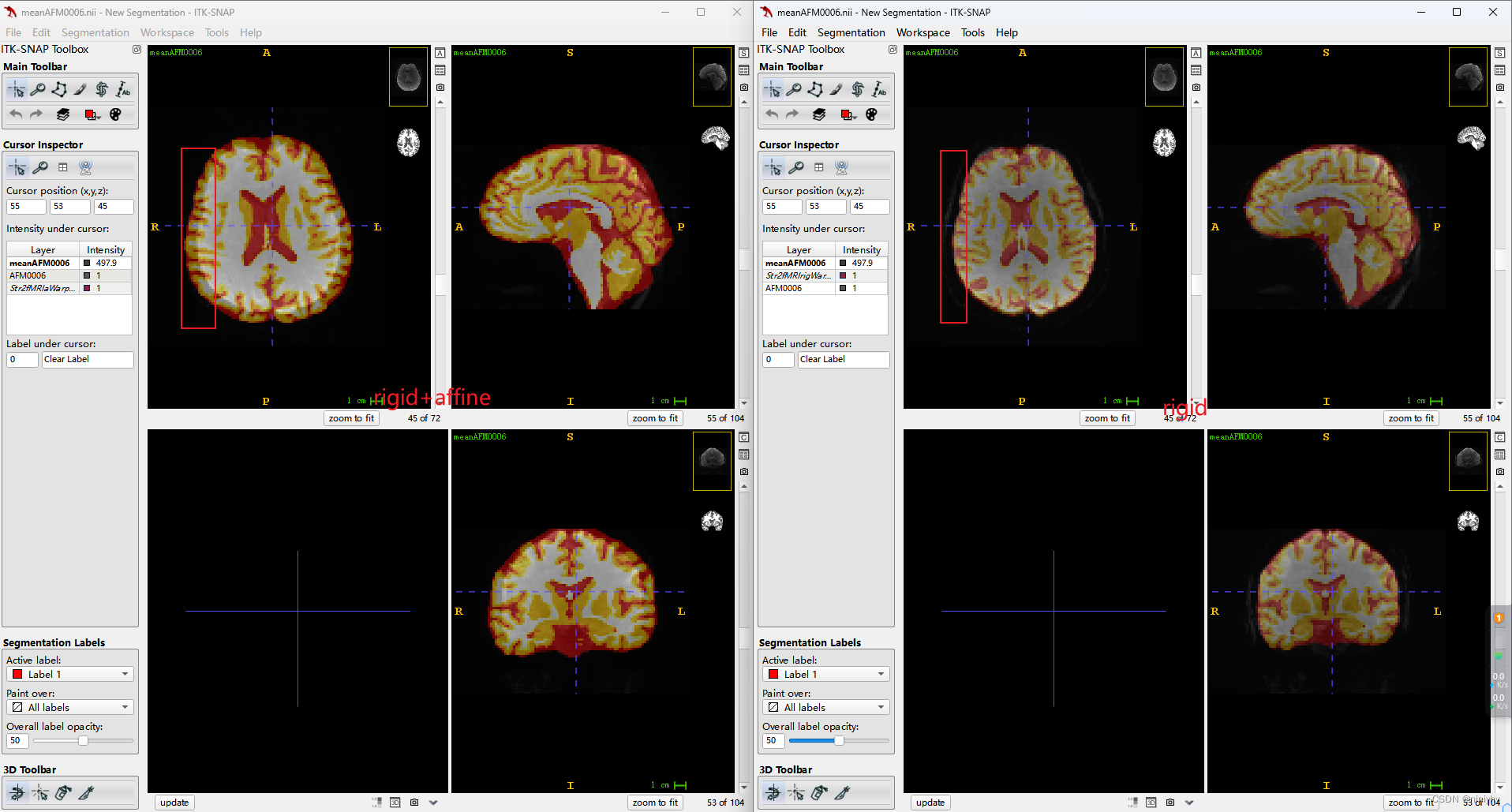
a: rigid + affine (2 stages) 会把颅骨也当做brain
stu to MNI
antsRegistrationSyN.sh -d 3 -f MNI_T1_2mm_brain.nii.gz -m brain.nii.gz -o rega2t
rega2t0GenericAffine.mat ,
rega2t1warp.nii.gz 分别表示线性变换和非线性变换估计出的映射关系
rega2twarped.niigz 表示配准后的图像,通过比较这个图像与标准大脑的差异,可以检查配准的质量
fmri to stu
antsRegistrationSyN.sh -d 3 -f brain.nii.gz -m example_func_brain.nii.gz -t ‘r’ -o regf2a
regf2a0GenericAffine.mat 表示从个体功能像到结构像的映射关系regf2awarped.nii.gz 表示配准后的功能像,按照结构像检查的相同的步骤进行功能像配
antsApplyTransforms -d 3 -i example_func_brain.nii.gz -o example_func2standard.nii.gz
-r MNI_T1_2mm_brain.nii.gz -t rega2t1Warp.nii.gz
-t rega2t0GenericAffine.mat -t regf2a0GenericAffine.mat
antsRegistration call:
--------------------------------------------------------------------------------------
/public/software/apps/ants/bin/antsRegistration
--verbose 0
--dimensionality 3 --float 0
--collapse-output-transforms 1
--output [ -n,-nWarped.nii.gz,-nInverseWarped.nii.gz ]
--interpolation Linear --use-histogram-matching 0
--winsorize-image-intensities [ 0.005,0.995 ]
--initial-moving-transform [ /public_bme/data/yuanhy/Pet_center/Huashan/fMRIImg2/AFM0006/meanAFM0006.nii,/public_bme/data/yuanhy/Pet_center/Huashan/T1_tissue2/AFM0006/AFM0006.nii.gz,1 ]
--transform Rigid[ 0.1 ]
--metric MI[ /public_bme/data/yuanhy/Pet_center/Huashan/fMRIImg2/AFM0006/meanAFM0006.nii,/public_bme/data/yuanhy/Pet_center/Huashan/T1_tissue2/AFM0006/AFM0006.nii.gz,1,32,Regular,0.25 ]
--convergence [ 1000x500x250x0,1e-6,10 ]
--shrink-factors 8x4x2x1
--smoothing-sigmas 3x2x1x0vox
/public/software/apps/ants/bin/antsRegistration
--verbose 0
--dimensionality 3 --float 0
--collapse-output-transforms 1
--output [ -n,-nWarped.nii.gz,-nInverseWarped.nii.gz ]
--interpolation Linear --use-histogram-matching 0
--winsorize-image-intensities [ 0.005,0.995 ]
--initial-moving-transform [ /public_bme/data/yuanhy/Pet_center/Huashan/fMRIImg2/AFM0006/meanAFM0006.nii,/public_bme/data/yuanhy/Pet_center/Huashan/T1_tissue2/AFM0006/AFM0006.nii.gz,1 ]
--transform Rigid[ 0.1 ]
--metric MI[ /public_bme/data/yuanhy/Pet_center/Huashan/fMRIImg2/AFM0006/meanAFM0006.nii,/public_bme/data/yuanhy/Pet_center/Huashan/T1_tissue2/AFM0006/AFM0006.nii.gz,1,32,Regular,0.25 ]
--convergence [ 1000x500x250x0,1e-6,10 ]
--shrink-factors 8x4x2x1
--smoothing-sigmas 3x2x1x0vox --transform Affine[ 0.1 ]
--metric MI[ /public_bme/data/yuanhy/Pet_center/Huashan/fMRIImg2/AFM0006/meanAFM0006.nii,/public_bme/data/yuanhy/Pet_center/Huashan/T1_tissue2/AFM0006/AFM0006.nii.gz,1,32,Regular,0.25 ] --convergence [ 1000x500x250x0,1e-6,10 ]
--shrink-factors 8x4x2x1
--smoothing-sigmas 3x2x1x0vox --transform SyN[ 0.1,3,0 ]
--metric MI[ /public_bme/data/yuanhy/Pet_center/Huashan/fMRIImg2/AFM0006/meanAFM0006.nii,/public_bme/data/yuanhy/Pet_center/Huashan/T1_tissue2/AFM0006/AFM0006.nii.gz,1,32]
--convergence [ 100x70x50x0,1e-6,10 ]
--shrink-factors 8x4x2x1
--smoothing-sigmas 3x2x1x0vox
1-5
量表-》MRI看形变-》PET(FDG代谢,AV45 Aβ)
使用语言模型 agent 引导 判断加某种模态
cost 如何衡量
T1 看萎缩,用得少
T2用于判断是否是其他疾病影响的认知衰退,从而判断是否可能是AD(加一个encoder即可)
fMRI用的少,临床上不怎么用
MTP 模态间不需要配准,一次性获得多个模态的数据
1-4
展示的subject选择
fold1 23
fold2 9
fold3 18
fold4 3
fold5 29
orig l 1.649
orig r 1.737
pre l 1.8252
pre r 1.93265
dis l 0.355846
dis r 0.307583
目前拿到的华山数据是116个ROI,时间点是488个
使用的是AAL atlas 116个
- Raw data
HUASHAN (T1, fMRI, PET, DTI)
/public_bme/share/HUASHAN/PET_center
全景(AFM1-AFM43):
Dimension(尺寸): 64 * 64 * 42 * 220
Voxel Size(体素大小): 3.4375 * 3.4375 * 3.4
TR(重复时间): 2250ms
TE(回波时间): 30ms
Slice Thickness(层厚): 3.4
Flip Angle(翻转角度): 77度
六院:
Dimension(尺寸): 104 * 104 * 72 * 488
Voxel Size(体素大小): 2 * 2 * 2
TR(重复时间): 800ms
TE(回波时间): 37ms
Slice Thickness(层厚): 2
Flip Angle(翻转角度): 52度
要用的应该是这两个序列
EP2D_BOLD_SMS_AP_HCP_2MM_IPAT12_0002 488个time point
李老师文章中用的是AP,FDG-PET
EP2D_BOLD_SMS_PA_HCP_2MM_0003 60个time point
-
AFM,AKM,AM分别是什么意思?
不用管它没有特殊含义 -
没有插值,用于单模态
- 第一步:功能向配准到结构向(可以得到非线性变换的矩阵+刚性变换矩阵)->标准空间配准到结构向(可以得到非线性变换的矩阵)
- 用标准空间配准到结构向 的逆矩阵 +功能向配准到结构向的逆矩阵和刚性变换(一共3个)反配回去功能向
- 有插值,用于多模态,都配到结构向去
- 第一步:功能像配准到结构像(可以得到非线性变换的矩阵+刚性变换矩阵)->标准空间配准到结构像(可以得到非线性变换的矩阵)
- 用标准空间配准到结构向 的逆矩阵 +功能向配准到结构向的矩阵和刚性变换(一共3个)反配回去结构像
可以先用这个做一整套,也可以用这个做到配准前,然后再接着用飞鸿老师给的脚本配准,之后再进行一些滤波平滑等操作。
数据处理加配准的dpabi 参考链接
Abbreviations
A - Slice Timing
R -Realign
W Normalize
S - Smooth
D Detrend
F - Filter
C - Covariates Removed
B - ScruBBing
sym - Normalized to a sym metric template
Rawdata中
FunRaw 每一个subject有488个timepoint
T1Raw 每一个subject有192或者208slice(分别代表三个不同方向的切片)
Dimension(尺寸): 104 * 104 * 72 * 488
Voxel Size(体素大小): 2 * 2 * 2
TR(重复时间): 800ms
TE(回波时间): 37ms
Slice Thickness(层厚): 2
Flip Angle(翻转角度): 52度
TR-0.8s
slice num 72
更正一下:
我要做的是先将T1配到FUN(A变换),T1配到标准空间(B变换)
再FUN->标准空间(
A
?
1
A^{-1}
A?1B)
apps/ants
salloc --cpus-per-task=8 --mem=32G --time=02:30:00 -p bme_cpu --exclusive
$app/q_ants.sh -d 3 -f $temp_path_fmri/$temp -m 5tt/tissue_pe.nii.gz -o 5tt/Str2MNIAnt_pe -n 10
把 5tt/tissue_pe.nii.gz配到
t
e
m
p
_
p
a
t
h
_
f
m
r
i
/
temp\_path\_fmri/
temp_path_fmri/temp
个体空间的结构像配到个体空间的功能像
输出的后缀为5tt/Str2MNIAnt_pe
去看一下这个命令的输出

antsApplyTransforms -d 3 -i 5tt/tissue_pe.nii.gz -r $temp_path_fmri/$temp -o 5tt/Str2MNIAnt_peWarped_NN.nii.gz -t 5tt/Str2MNIAnt_pe1Warp.nii.gz -t 5tt/Str2MNIAnt_pe0GenericAffine.mat --interpolation NearestNeighbor
将刚刚的转换矩阵应用到5tt/tissue_pe.nii.gz 【182,218,182】
参考是
t
e
m
p
p
a
t
h
f
m
r
i
/
temp_path_fmri/
tempp?athf?mri/temp
(0是线性转换的文件,1是非线性转换的文件)
非线性转换的数据:5tt/Str2MNIAnt_pe1Warp.nii.gz 【193,229,193】
线性转换的文件:5tt/Str2MNIAnt_pe0GenericAffine.mat
输出是 Str2MNIAnt_peWarped_NN.nii.gz 【193,229,193】
得到了每一个subject的结构像
/public_bme/data/yuanhy/Pet_center/Huashan/2_shells
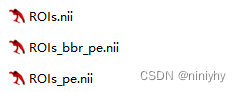
5TT.nii.gz, 5TT_bbr_pe.nii.gz, 5TT_pe.nii.gz, 5tt_vol0.nii.gz: 这些文件可能代表5组织类型(5 Tissue Types)的图像,通常包括白质、灰质、脑脊液、血管和背景。5tt是这些组织类型的缩写。不同的后缀可能代表不同的处理步骤或参数。
ROIs.nii.gz, ROIs_bbr_pe.nii.gz, ROIs_pe.nii.gz: 这些文件包含感兴趣区域(Regions of Interest)的信息。ROI通常是研究者特别关注的大脑区域。
Str2MNIAnt_peInverseWarped.nii.gz, Str2MNIAnt_peWarped.nii.gz, Str2MNIAnt_peWarped_NN.nii.gz, Str2MNIAnt_pe1InverseWarp.nii.gz, Str2MNIAnt_pe1Warp.nii.gz: 这些文件看起来像是经过某种空间变换(warping)的图像,可能是为了将图像从一个空间对齐到另一个空间,例如从原始扫描空间到MNI(Montreal Neurological Institute)标准空间。
grey_matter.nii.gz, white_matter.nii.gz: 这些文件分别代表灰质和白质的图像。
har_pe.nii.gz, hoa_pe.nii.gz, csf.nii.gz, peaks.nii.gz, tissue_pe.nii.gz: 这些文件可能包含其他类型的大脑图像数据,如HARDI(High Angular Resolution Diffusion Imaging)数据、HOA(High Order Angular)数据、脑脊液(Cerebrospinal Fluid)图像、扩散峰度图像等。
s_100_7_pe.nii.gz, s_1000_7_pe.nii.gz, …: 这些文件可能代表不同参数或不同处理步骤下的图像数据。具体的含义需要查阅相关的文档或询问数据提供者。
RF_voxels.nii.gz: 这个文件可能包含与随机森林(Random Forest)或其他机器学习算法相关的体素(voxel)信息。
z1024_pe.nii.gz, z980_pe.nii.gz: 这些文件可能是某种特定层面或切片的图像数据。
请注意,以上解释都是基于文件名和常见的医学图像处理实践给出的推测。要准确理解这些文件的含义和内容,最好是查阅相关的文档或询问提供这些数据的研究者或机构。
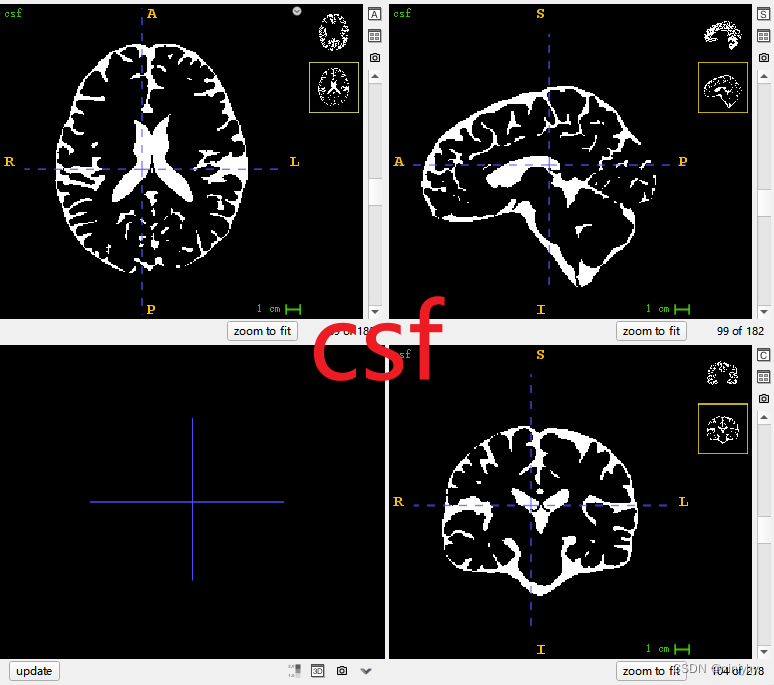
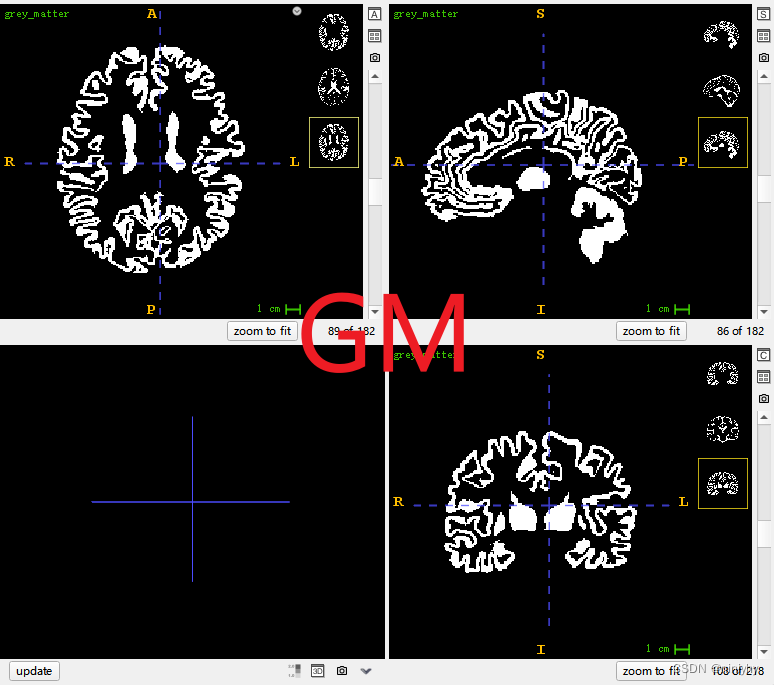
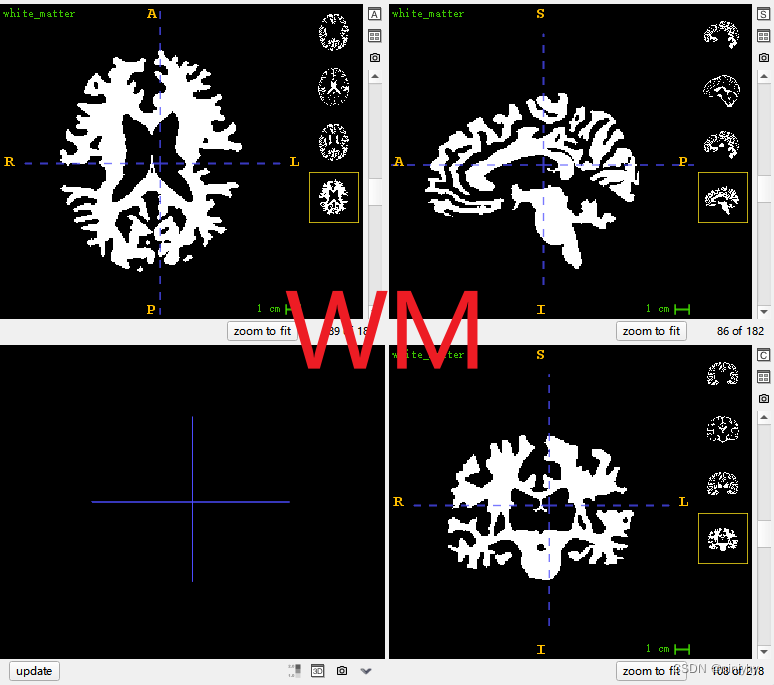
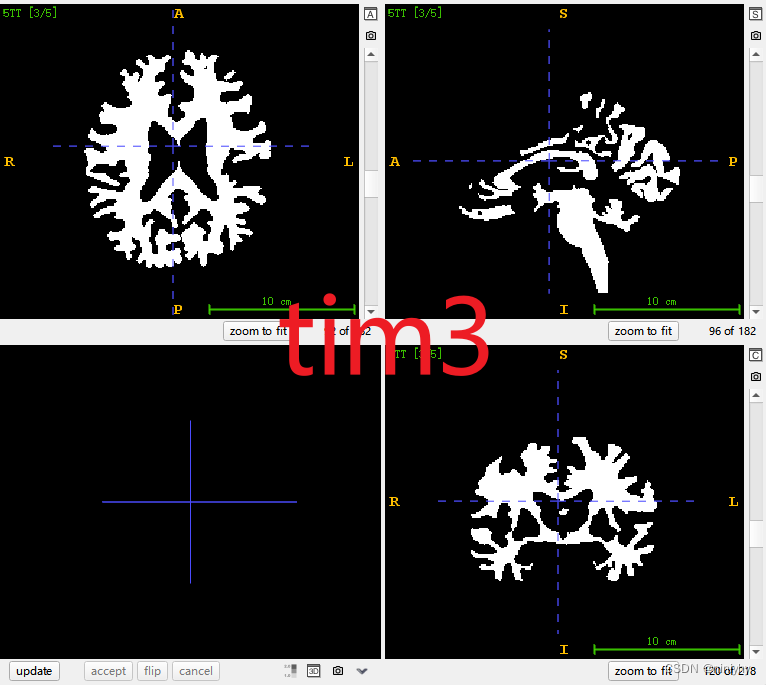
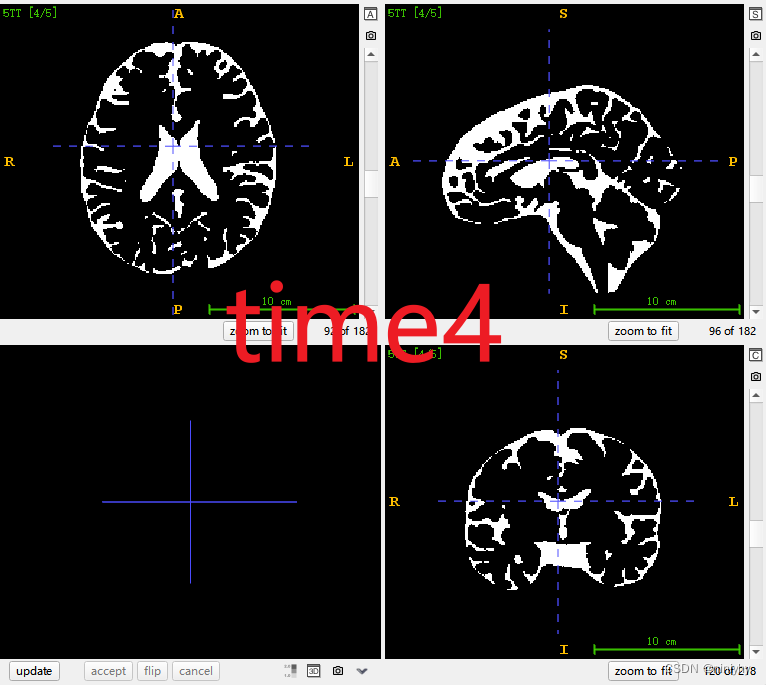
5tt time point
time1+time2 是GM
time3 就是WM
time4 脑脊液

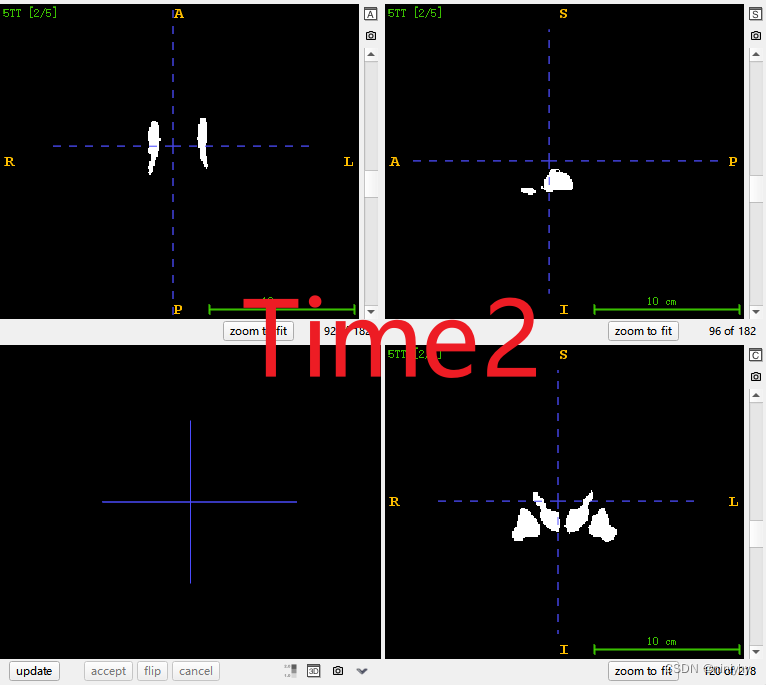

“bbr” 可能表示 Boundary-Based Registration(基于边界的注册)。这意味着生成 “5TT_bbr_pe.nii” 文件时可能使用了基于图像边界的注册算法。
bbr的边界确实不一样,5tt_vol0只有灰质(只有一个时间点,没有中间那个东西,也就是只有Time1的)

84个ROI
fMRI 【112,112,66】

以上十三个是有bold signal信号,但是看时间点不是488个,是976个或者更少

更新如下
huashan raw data /public_bme/share/HUASHAN/PET_center
更新以下是2_shell有的但是fMRIAP没有
{‘AFM0121’, ‘AFM0124’, ‘AFM0202’, ‘AFM0204’, ‘AFM0120’, ‘AFM0122’, ‘AFM0203’, ‘AFM0278’, ‘AFM0092’, ‘AFM0123’, ‘AFM0492’, ‘AFM0208’}
s_shell 669个subject
fMRI 657个subject
/public_bme/data/yuanhy/Pet_center/Huashan/fMRIImgbackup 原始数据
/public_bme/data/yuanhy/Pet_center/Huashan/fMRIImg 做好了头动矫正


DIM=3
FIXEDIMAGES=()
MOVINGIMAGES=()
INITIALTRANSFORMS=()
OUTPUTNAME=output
NUMBEROFTHREADS=0
SPLINEDISTANCE=26
SYNGRADIENTSTEP=0.1
TRANSFORMTYPE=‘s’
PRECISIONTYPE=‘d’
NUMBEROFBINS=32
MASKIMAGES=()
USEHISTOGRAMMATCHING=0
COLLAPSEOUTPUTTRANSFORMS=1
RANDOMSEED=0
REPRO=0
/public/software/apps/ants/bin/antsRegistration
--verbose 0 --dimensionality 3 --float 0
--collapse-output-transforms 1
--output [/public_bme/data/yuanhy/Pet_center/Huashan/2_shellsTest/AFM0006/5tt/Str2MNIAnt_pe,
/public_bme/data/yuanhy/Pet_center/Huashan/2_shellsTest/AFM0006/5tt/Str2MNIAnt_peWarped.nii.gz,
/public_bme/data/yuanhy/Pet_center/Huashan/2_shellsTest/AFM0006/5tt/Str2MNIAnt_peInverseWarped.nii.gz ]
--interpolation Linear
--use-histogram-matching 0
--winsorize-image-intensities [ 0.005,0.995 ]
--initial-moving-transform [ /public_bme/data/yuanhy/Pet_center/Huashan/fMRIImgTest/AFM0006/meanAFM0006.nii,
/public_bme/data/yuanhy/Pet_center/Huashan/2_shellsTest/AFM0006/5tt/tissue_pe.nii.gz,
1 ]
----------------------------------------------------------
--transform Rigid[ 0.1 ]
--metric MI[/public_bme/data/yuanhy/Pet_center/Huashan/fMRIImgTest/AFM0006/meanAFM0006.nii,
/public_bme/data/yuanhy/Pet_center/Huashan/2_shellsTest/AFM0006/5tt/tissue_pe.nii.gz,
1,32,Regular,0.25 ]
--convergence [ 1000x500x250x0,1e-6,10 ]
--shrink-factors 8x4x2x1
--smoothing-sigmas 3x2x1x0vox
----------------------------------------------------------
--transform Affine[ 0.1 ]
--metric MI[/public_bme/data/yuanhy/Pet_center/Huashan/fMRIImgTest/AFM0006/meanAFM0006.nii,/public_bme/data/yuanhy/Pet_center/Huashan/2_shellsTest/AFM0006/5tt/tissue_pe.nii.gz,1,32,Regular,0.25 ]
--convergence [ 1000x500x250x0,1e-6,10 ]
--shrink-factors 8x4x2x1
--smoothing-sigmas 3x2x1x0vox
----------------------------------------------------------
--transform SyN[ 0.1,3,0 ]
--metric MI[ /public_bme/data/yuanhy/Pet_center/Huashan/fMRIImgTest/AFM0006/meanAFM0006.nii,/public_bme/data/yuanhy/Pet_center/Huashan/2_shellsTest/AFM0006/5tt/tissue_pe.nii.gz,1,32]
--convergence [ 100x70x50x0,1e-6,10 ]
--shrink-factors 8x4x2x1
--smoothing-sigmas 3x2x1x0vo
将三个tissuemap加在一起得到概率图(做一个带权的)
tissue 是个体空间的,老师给我的脚本是将
要预测的是AV45-PET
处理PET

大部分主流的就是FBP、OSEM3D和TrueX三种
统计一下每个方法都有多少个被试

https://aramislab.paris.inria.fr/clinica/docs/public/dev/Pipelines/PET_Surface/
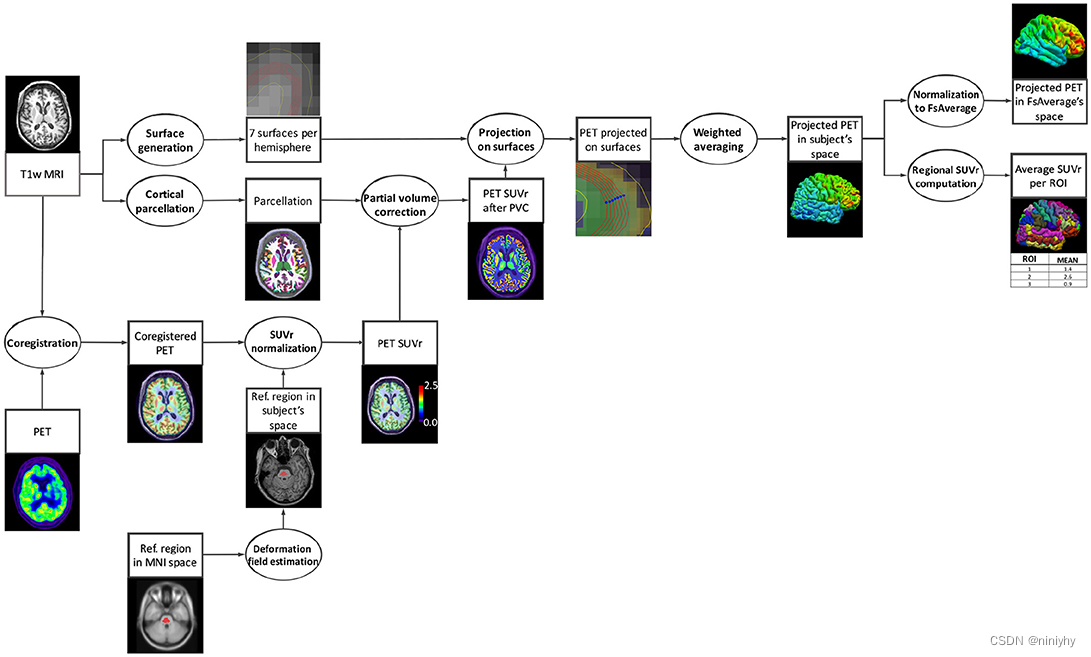
PetSurfer 这个工具可以做PETROI的提取
白医生推荐
https://alz-journals.onlinelibrary.wiley.com/doi/full/10.1002/alz.045455
https://www.researchgate.net/figure/Translation-results-of-early-PET-image-according-to-the-reconstruction-method-Every_fig3_374223115
数据
处理好的数据 /public_bme/data/liumx/Huashan3/Preproc
原始数据 /public_bme/share/HUASHAN/PET_center
dmri /public_bme/data/FeihongLiu/Test/Pet_center/Huashan/
/public_bme/data/yuanhy/Pet_center/Huashan/fMRIImgbackup 原始数据
/public_bme/data/yuanhy/Pet_center/Huashan/fMRIImg2 第二次做dcm2nii的原始数据
/public_bme/data/yuanhy/Pet_center/Huashan/fMRIImg 做好了头动矫正
tissue tamplate D:\Projects\p#001DTI2PET\label_template_mni_sym_addsymsubcort_masked.nii
s_shell 669个subject
fMRI 657个subject
{'AFM0204', 'AFM0120', 'AFM0121', 'AFM0122', 'AFM0278', 'AFM0492', 'AFM0124', 'AFM0203', 'AFM0208', 'AFM0202', 'AFM0123', 'AFM0092'}
以上为有smri但是没有fmri
The number of time points in the :AFM0165 NIfTI file is: 486
The number of time points in the :AFM0047 NIfTI file is: 486
这两个subject timepoint不是488

这三个应该是重复数据
所以实际有用数据是657-3 = 654个
这些分别是什么文件?
/public_bme/data/yuanhy/Pet_center/Huashan/fMRIImg2 对这个进行realign 预计 完成√
/public_bme/data/yuanhy/Pet_center/Huashan/T1_tissue2进行stc2MNI 完成√
/public_bme/data/yuanhy/Pet_center/Huashan/fMRIImg2/AFM0006/fun2MNI_r.nii.gz mean 配准到白质MNI上(单模态:T1白质和T1的template)
/public_bme/data/yuanhy/Pet_center/Huashan/2_shells/AFM0054/T1/T1_acpc_proc_brain.nii.gz T1brain [182,218,182]
2_shells : 原始的T1 5tt 文件
2_shellsbackup 原始的5tt文件中的tissu文件
fMRIAP fMRIdcm 文件
fMRIImg fMRI转成nifti文件(第一次转换)
fMRIImgbackup fMRI转成nifti文件(第一次转换 备份
fMRIImg2 进行了头动矫正的fMRI
FunImg 只有609个fMRI转成nifti文件(不是现在所需要用的)
T1_tissue T1 tissue文件(命名带tissue)
T1_tissuebackup T1 tissue文件(命名带tissue)备份
T1_tissue2 T1 tissue配准到了标准空间的tissue(命名不带tissue)
T1_tissuebackup2 T1 tissue文件(命名不带tissue)备份
PET_crecmt.csv
PET_crecmt2.csv
template中没有框起来的是我自己生成的
intensity_brain 是去了脑壳的template
gm_msk wm_msk,csf_msk 是分左右脑的tissue 拿出来的
gm_mask wm_mask,csf_mask 是不区分左右脑的tissue
intensity_tissue3v是不区分左右脑的灰质白质脑脊液的mask
"D:\yhy\data\huashan\2_shells\AFM0006\tmp_odf_reg2" 新跑了一下多模态配准
D:\yhy\data\huashan\2_shells\AFM0006\mrbrain是配准后的tissue 而不是原始的
展示的subject选择
fold1 23
fold2 9
fold3 18
fold4 3
fold5 29
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:chenni525@qq.com进行投诉反馈,一经查实,立即删除!
- Python教程
- 深入理解 MySQL 中的 HAVING 关键字和聚合函数
- Qt之QChar编码(1)
- MyBatis入门基础篇
- 用Python脚本实现FFmpeg批量转换
- 【Leetcode】接雨水(双指针、单调栈)
- Flink 数据序列化
- Erlang、RabbitMQ下载与安装教程(windows超详细)
- 性能测试分析案例-用缓存优化程序的运行效率
- 《吐血整理》进阶系列教程-拿捏Fiddler抓包教程(14)-Fiddler断点(breakpoints)实战,篡改或伪造数据
- MySQL中CASE when 实战
- 软件工程期末复习
- [FireshellCTF2020]Caas c语言预编译的文件包含
- 3dmax灯光缓存参数怎么设置?解析来了!
- 【ARM Trace32(劳特巴赫) 使用介绍 14 -- Go.direct 介绍】