R语言VRPM包绘制多种模型的彩色列线图
列线图,又称诺莫图(Nomogram),它是建立在回归分析的基础上,使用多个临床指标或者生物属性,然后采用带有分数高低的线段,从而达到设置的目的:基于多个变量的值预测一定的临床结局或者某类事件发生的概率。列线图(Nomogram)可以用于多指标联合诊断或预测疾病发病或进展。
近些年来在高质量SCI临床论文中用的越来越多。列线图将回归模型转换成了可以直观的视图,让结果更容易判断,具有可读性,例如:
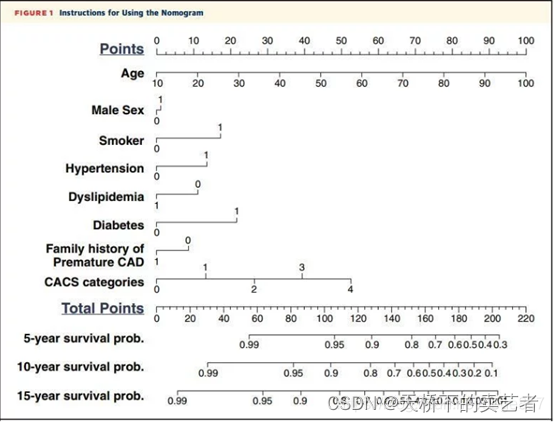
咱们既往已经多篇文章介绍绘制列线图,今天咱们来介绍一下VRPM包绘制彩色列线图,这个包可以绘制多个模型的列线图,咱们一一来介绍。
首先VRPM包已经在R官方下架,不能直接安装,咱们可以
install.packages("devtools") # 安装devtools包
devtools::install_github("nanxstats/VRPM") # 安装VRPM包
或者在我的公众号吧这个包下载下来(公众号回复:VRPM包),手动安装
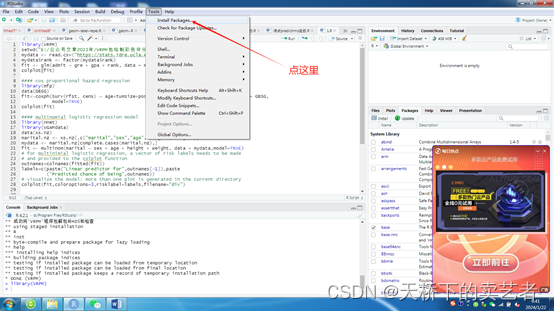
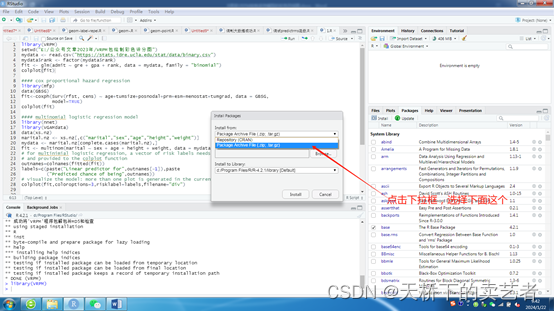
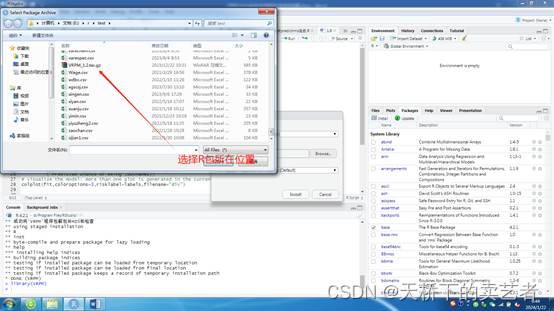
如果遇到下面报错的话,表明’fields’, ‘R2HTML’这两个包的版本不合适,

咱们需要把’fields’, 'R2HTML’这两个包从新安装一下,然后再次安装就可以了
安装好VRPM包后咱们就可以开始了,先导入R包
library(VRPM)
library(survival)
设置好咱们的目录,这样有个好处,VRPM包是直接把图片保存到默认目录的,这样你比较好找
setwd("E:/公众号文章2023年/VRPM包绘制彩色评分图")
导入数据,把分类变量转成因子
mydata <- read.csv("mydata.csv")
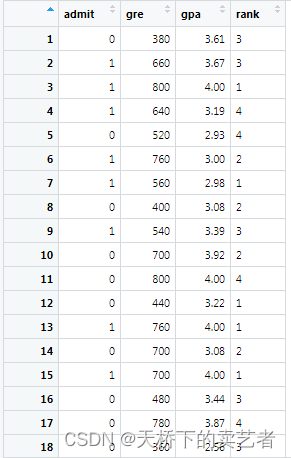
这是很简单的数据,admit是个分类的结局变量,其他的是协变量,(公众号回复:列线图数据,可以获得这个数据。)
把分类变量转成因子
mydata$rank <- factor(mydata$rank)
建立模型,先来个逻辑回归的
fit <- glm(admit ~ gre + gpa + rank, data = mydata, family = "binomial")
绘制列线图
colplot(fit)
绘图后并不在R界面生成我们需要在默认目录下找,就是刚才咱们设置的目录
(“E:/公众号文章2023年/VRPM包绘制彩色评分图”)
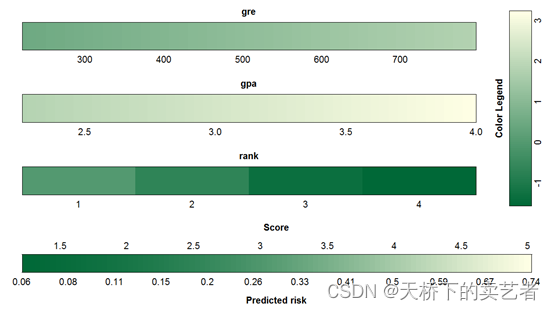
这样一个彩色列线图就生成了,根据颜色深浅表示了危险程度。咱们还可以对颜色进行一些修改
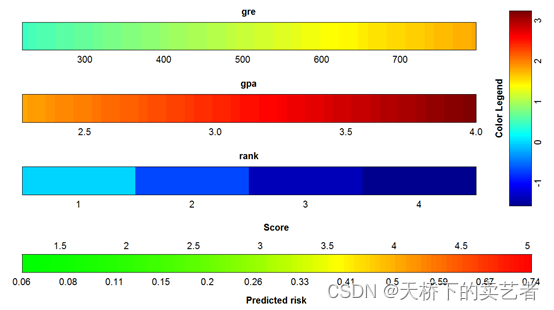
colplot(fit,coloroptions=1)
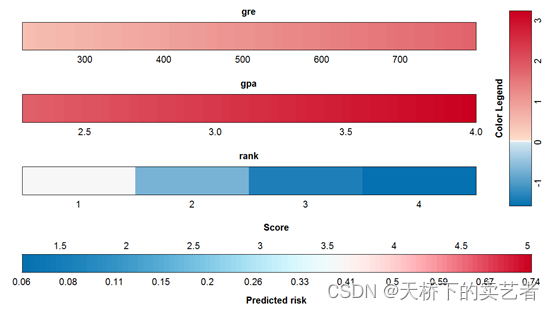
colplot(fit,coloroptions=3)
接下来咱们来个cox回归的,使用的是mfp包自带的数据
library(mfp)
data(GBSG)
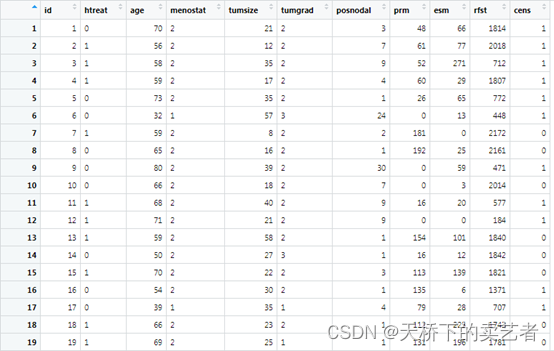
数据中的rfst是生存时间,cens是结局变量,其他的是协变量
建立模型
fit<-coxph(Surv(rfst, cens) ~ age+tumsize+posnodal+prm+esm+menostat+tumgrad, data = GBSG,
model=TRUE)
绘图
colplot(fit)
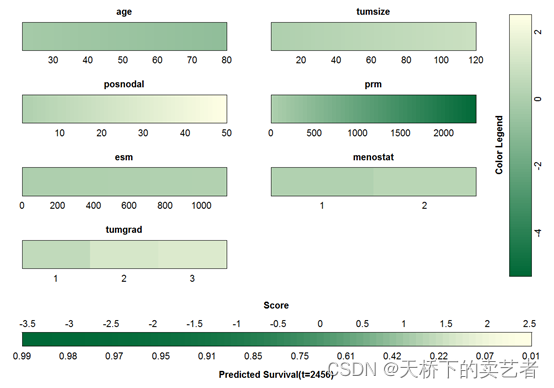
下面咱们来个结局变量Y是多分类的逻辑回归模型,使用的是VGAMdata包自带的xs.nz数据
l
ibrary(nnet)
library(VGAMdata)
data(xs.nz)
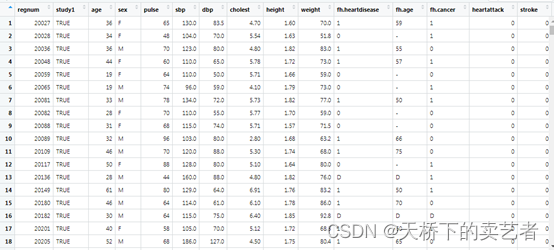
这个数据很大,咱们提取需要的变量来组成模型
marital.nz <- xs.nz[,c("marital","sex","age","height","weight")]
mydata <- marital.nz[complete.cases(marital.nz),]
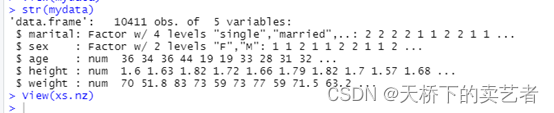
咱们可以看到结局变量marital是4分类变量
str(mydata)
建立模型
fit <- multinom(marital ~ sex + age + height + weight, data = mydata,model=TRUE)
对于多项式逻辑回归,有多个结局概率,咱们需要指定你研究的是指标
咱们先把结局的4个变量提取出来,分别是4个婚姻状态“单身”“已婚”“离婚”“寡居”
outnames=colnames(fitted(fit))

咱们单身为参考变量,绘制列线图,先生成图片的标题
labels=c(paste("Linear predictor for",outnames[-1]),paste
("Predicted chance of being",outnames))

接下来绘图,会生成多个图片,它是分成2个图片一组的,发表的时候可能需要拼一下图
colplot(fit,coloroptions=3,risklabel=labels,filename="div")

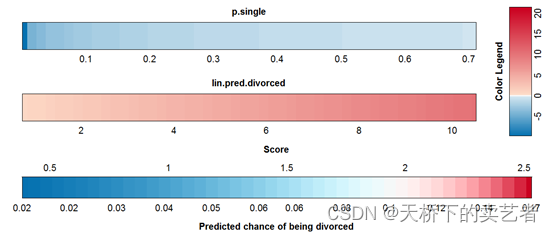
接下来是married的
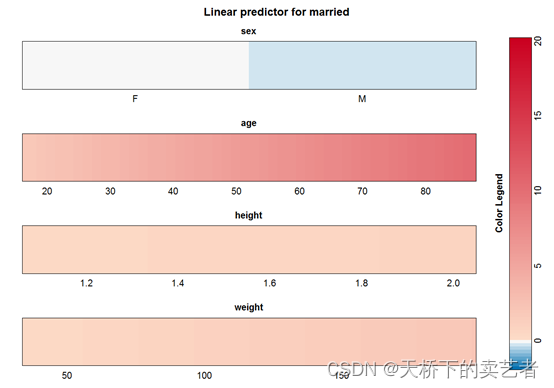
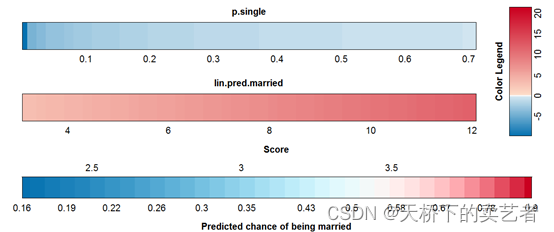
接下来是widowed的
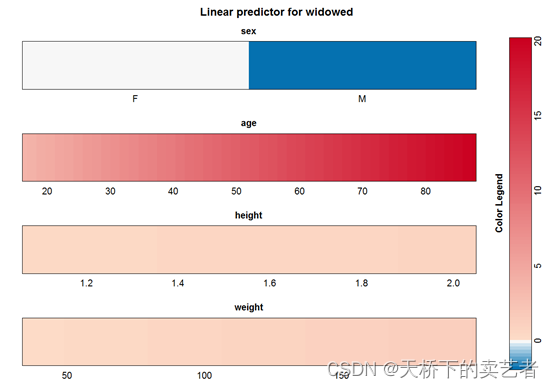

图片很多还有几张就不发上来了。
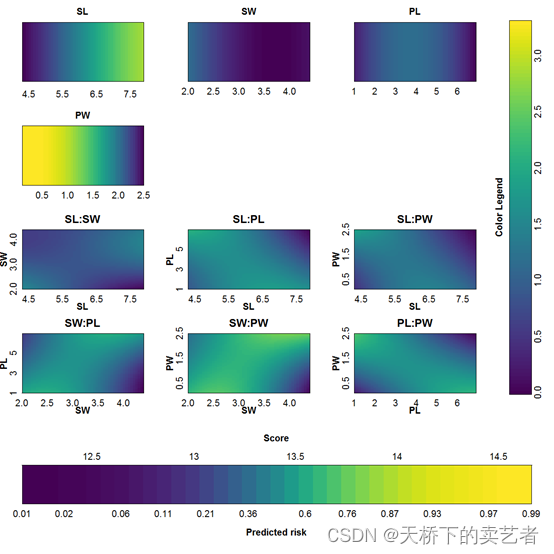
接下来咱们绘制个支持向量机分类的,使用R自带的鸢尾花数据
library(kernlab)
data(iris)
咱们把其他两种类型的花设置为other
levels(iris$Species)[levels(iris$Species)=="setosa"] <- "other"
levels(iris$Species)[levels(iris$Species)=="virginica"] <- "other"
对数据重新命名
names(iris)=c("SL","SW","PL","PW","Species")
生成模型
model <-ksvm(Species ~ ., data = iris,prob.model=TRUE,kpar=list(0.03),C=10)
对模型进行预处理,
newmodel=preplotperf(model,iris,indy=5,zerolevel="min")
绘图
colplot(newmodel,filename="IRIS2",zerolevel="min",coloroptions=5)
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:chenni525@qq.com进行投诉反馈,一经查实,立即删除!
- Python教程
- 深入理解 MySQL 中的 HAVING 关键字和聚合函数
- Qt之QChar编码(1)
- MyBatis入门基础篇
- 用Python脚本实现FFmpeg批量转换
- 17. 电话号码的字母组合 --力扣 --JAVA
- openmediavault(OMV) (24)在线网盘(2)kodcloud
- latch: CDC 与buffer busy waits
- 智慧水务总体设计
- LeetCode.2865. 美丽塔 I
- 动手学深度学习-自然语言处理:应用
- 实例分割模型Mask2Former解析
- MultiArch 与 Ubuntu/Debian 的交叉编译(一)
- 正运动技术荣获2023年度“AI天马”认定
- 人工智能计算机视觉处理设计开发工程师证书报名条件及报考流程