生信人R语言
发布时间:2024年01月07日
09. 任意基因任意癌症表达量和临床性状关联
#视频中的代码
rm(list = ls())
options(stringsAsFactors = F)
#Ovarian serous Cystadenocarcinoma(TCGA,Nature 2011)
#All complete Tumors (316 samples)/1 Genes
#Gene Set/Pathway is altered in 28 (8.9%) of queried samples
a = read.table('plot-data-ARHGAP18-TCGA-OV-cbioportal.txt',header = T, sep = '\t', fill = T)
colnames(a) = c('id','stage','gene','mut')
dat = a
library(BiocManager)
BiocManager::install("metaBMA")
library(ggstatsplot)
ggbetweenstats(data = dat,x=stage,y = gene)
library(ggplot2)
ggsave('plot-data-ARHGAP18-TCGA-OV-cbioportal.png')10. 表达矩阵的样本相关性
load("D:/Soft/airway_exprSet.Rdata")
dim(exprSet)#查看样本维度;8个维度,六万多个样本
#cor函数验证样本之间的相关性;接近于1,则相关性越高
cor(exprSet[,1],exprSet[,2])#取表格的第一列和第二列
cor(exprSet)
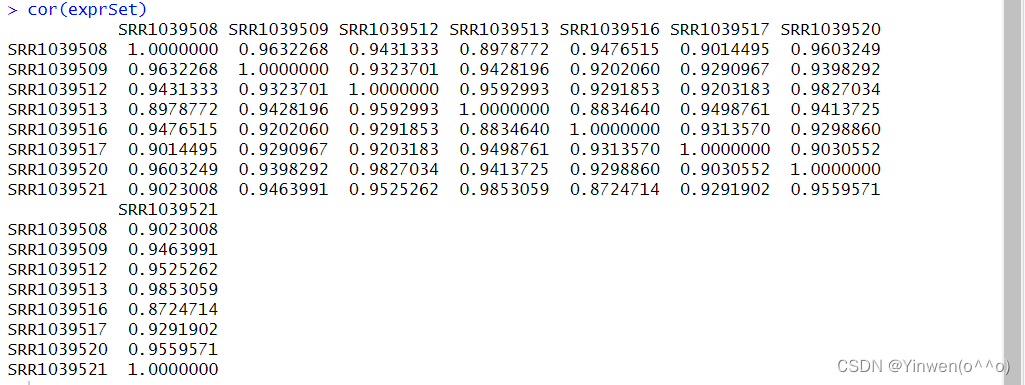
#根据相关性作图
pheatmap::pheatmap(cor(exprSet))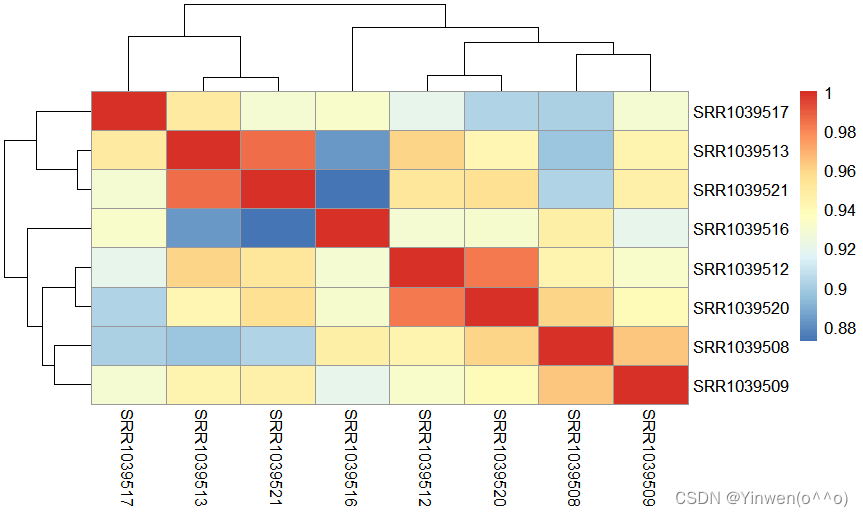
?
#把矩阵取小
x=exprSet[1,]
x>1#返回值为T或F
sum(x>1)
x=exprSet[2,]
x>1#返回值为T或F
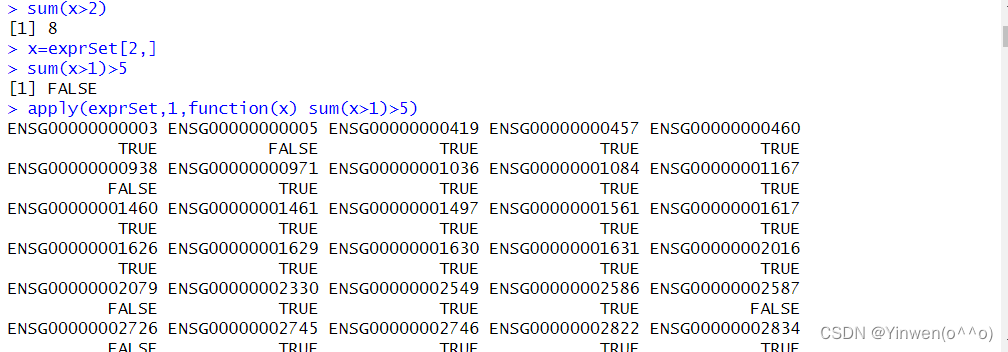
sum(x>1)>5#基因表达量为大于5则符合要求
apply(exprSet,1,function(x) sum(x>1)>5)
dim(exprSet)#查看样本维度1
exprSet=exprSet[apply(exprSet,1,function(x) sum(x>1)>5),]
dim(exprSet)#查看样本维度2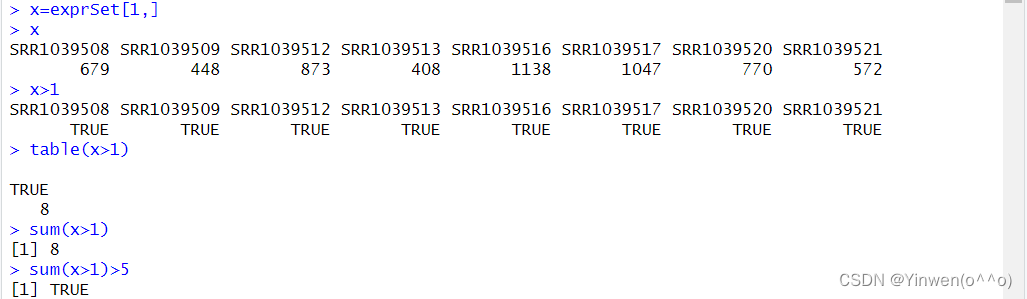
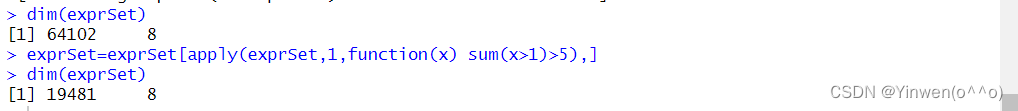
#查看已安装的R包
#安装edgeR
#colnames(installed.packages())
#install.packages('BiocManager')
BiocManager::install('edgeR')
exprSet=log(edgeR::cpm(exprSet)+1)
exprSet=exprSet[names(sort(apply(exprSet,1,mad),decreasing = T)[1:500]),]
dim(exprSet)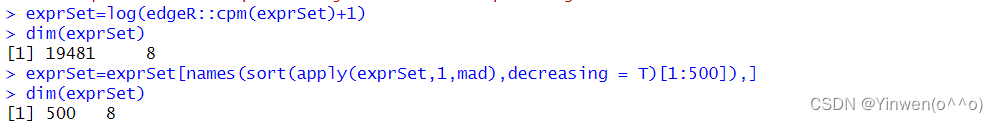
M=cor(log2(exprSet+1))
pheatmap::pheatmap(M)
dev.off()#关闭画板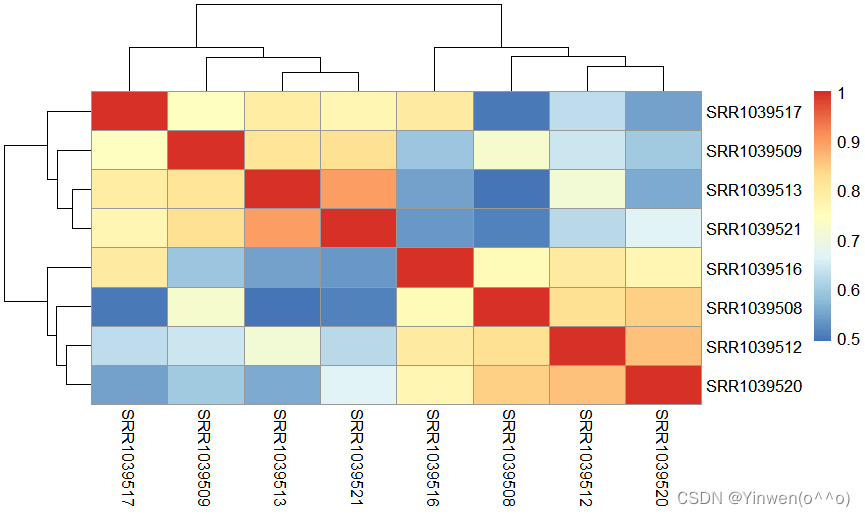
?
?
?
?
?
?
?
?
?
?
?
?
?
?
?
?
?
?
?
?
?
?
?
?
?
文章来源:https://blog.csdn.net/m0_56780122/article/details/135208603
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:chenni525@qq.com进行投诉反馈,一经查实,立即删除!
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:chenni525@qq.com进行投诉反馈,一经查实,立即删除!
最新文章
- Python教程
- 深入理解 MySQL 中的 HAVING 关键字和聚合函数
- Qt之QChar编码(1)
- MyBatis入门基础篇
- 用Python脚本实现FFmpeg批量转换
- 【 YOLOv5】目标检测 YOLOv5 开源代码项目调试与讲解实战(2)-如何利用yolov5进行预测
- MySQL 基础语法
- 【LeetCode刷题笔记(12-1)】【Python】【有效的字母异位词】【排序/字符统计】【简单】
- 【华为机试】2023年真题B卷(python)-数列描述
- 频谱论文:约束飞行轨迹下基于生成对抗网络的三维无线电地图重构
- 电阻选型考虑什么, 点灯电路,USB2.0电路中电阻选取
- 从0开始学C++ 第二十八课 数据结构深入 - 栈和队列
- 美国西南航空员工激励为企业管理带来的启示
- 【Java程序设计】(C00145) 基于Springboot+Vue的前后端分离的学生成绩管理系统-有报告
- Grouping Increases