基于经纬度信息提取气候因子图层数据
发布时间:2023年12月21日
在进行生物地理学研究时,我们时常会涉及到提取特定位置的气候变量值,而ArgMap操作过于麻烦(
主要是要付费
\color{red}{主要是要付费}
主要是要付费),而R包raster提供的功能完美解决了该问题。
1 涉及的包和数据集
R包:
library(raster) # 主要包
library(terra)
library(ncdf4)
物种位置数据:随便到GBiF上下载一组自己喜欢的数据即可;
气候因子图层:worldclim自行下载。
2 数据读取及坐标系转换
读取数据:
occur <- read.csv("Ocur_GBiF.csv",header=T, row.names = "ID") # 位置信息
bio1 <- raster("wc2.1_10m_bio/wc2.1_10m_bio_1.tif") # bio1图层
设置参考坐标系,一般选取WGS84:
crs.wgs <- "+proj=longlat +datum=WGS84 +no_defs +ellps=WGS84 +towgs84=0,0,0"
图层及位置信息的坐标系转换:
crs(bio1) <- crs.wgs
occ <- SpatialPointsDataFrame(
occur[,c('longitude','latitude')],
proj4string=CRS(crs.wgs),
data=occur
)
3 提取气候变量信息
根据位置信息提取气候变量值:
lo_bio1 <- raster::extract(bio1,occ)
现在利用for循环批量提取1~19变量:
for (i in 1:19) {
var = raster(paste0("wc2.1_10m_bio/wc2.1_10m_bio_",i,".tif"))
var_val = raster::extract(var, occ)
occur = cbind(occur, var_val)
colnames(occur)[i+2] <- paste0("bio",i)
}
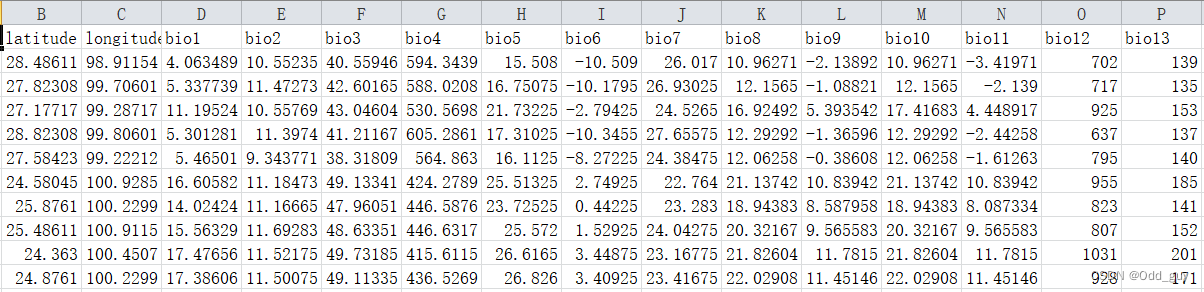
write.csv(occur, 'bio_val.csv', quote = FALSE)
最终结果保存于bio_val.csv里。
END
文章来源:https://blog.csdn.net/SUN5_The_answer/article/details/135131492
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:chenni525@qq.com进行投诉反馈,一经查实,立即删除!
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:chenni525@qq.com进行投诉反馈,一经查实,立即删除!
最新文章