使用 MONAI 加载和保存各种格式的医学图像
本教程属于实战,手把手教你加载各种医学图像数据(nii.gz, .dcm, .png等)。并学会查看医学图像数据的元数据(shape, affine, orientation)。学会使用monai全方位了解你的数据,并把它用于之后的深度学习训练。以及学会保存transform处理后的图像以及分割结果。干货很多,动手跟着一起来。
查看本教程前,请自行下载参考代码,边跑代码边看教程,学习效率更高哦
使用默认 image reader 加载 Nifti 图像
monai 根据文件后缀选择默认阅读器:
- nii、nii.gz -> NibabelReader
- png、jpg、bmp -> PILReader
- npz、npy -> NumpyReader
- 其他 -> ITKReader
开始实验前准备你的 nifti 格式图像,
filename = '/Users/Downloads/WORD-V0.1.0/imagesTr/word_0002.nii.gz'
如果没有,可以下载monai的示例
# download a test image
tempdir = tempfile.mkdtemp()
test_url = "https://github.com/Project-MONAI/MONAI-extra-test-data/releases/download/0.8.1/avg152T1_RL_nifti.nii.gz"
filename = os.path.join(tempdir, "avg152T1_RL_nifti.nii.gz")
monai.apps.download_url(test_url, filepath=filename)
接下来使用LoadImage加载图像
data = LoadImage(image_only=True, ensure_channel_first=True, simple_keys=True)(filename)
print(f"image data shape: {data.shape}")
print(f"meta data: {data.meta.keys()}")

参数解析
image_only: 默认为True,只返回图像;if False, 返回的data就是tuple, image=data[0], metadata(元数据)=data[1]ensure_channel_first:默认为False, if True, 会给图像加一个channel,假如图像shape=[512, 512, 64], 加了channel就变成[1, 512, 512, 64]. 这在深度学习训练中基本都是要设为True, 向本教程的目的是加载图像的话,就不必设为True
其余参数可以在pycharm源代码里查看
接下来,重点解释一下元数据,这是医学图像独有的信息。可以使用data.meta获取所有的元数据。包括
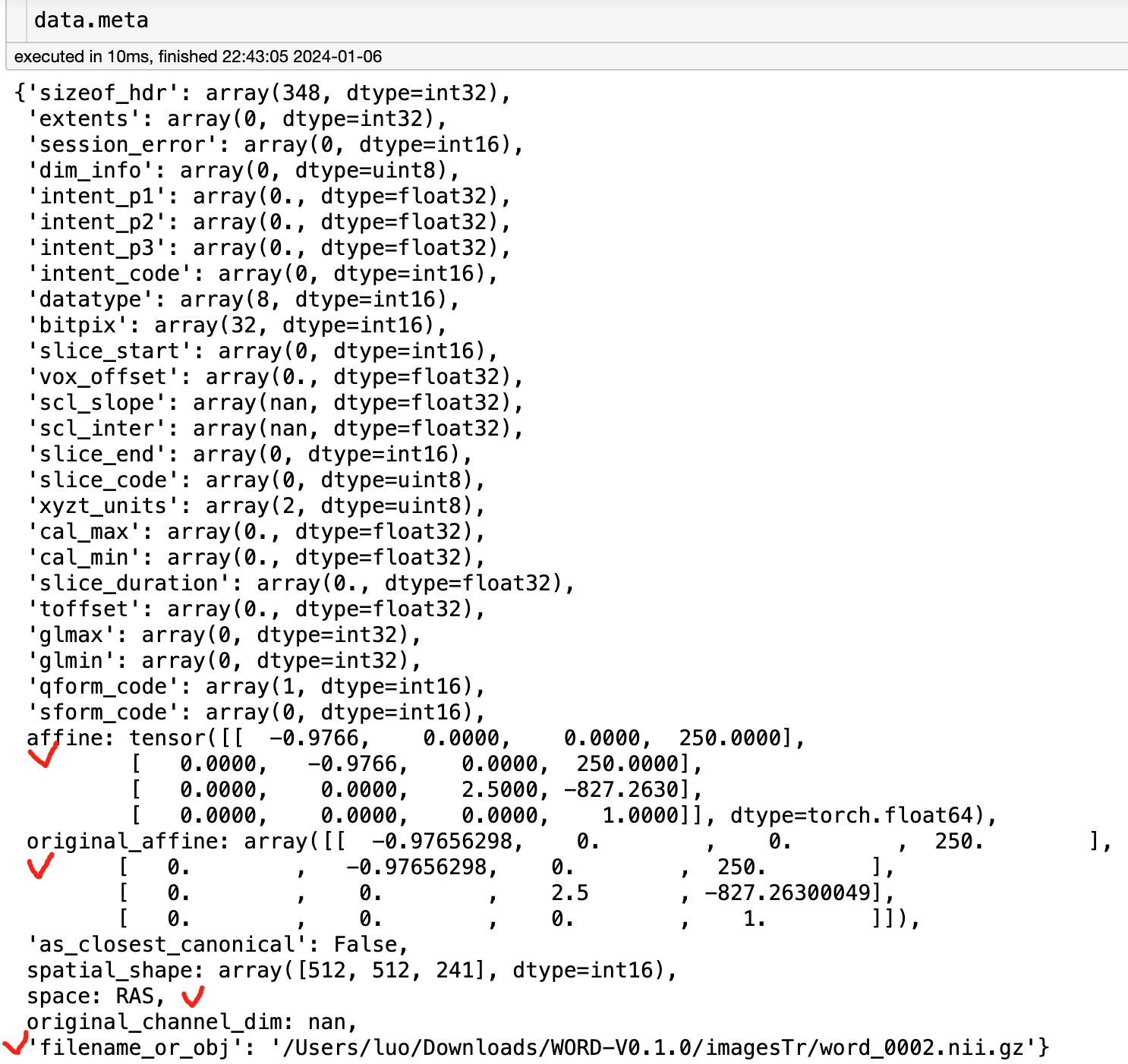
打勾的几个数据就是我平时用的比较多的,比如做图像分割的时候,要保存图像和分割结果,就需要知道affine信息。分割结果命名想同图像命名对应的时候,就需要知道filename_or_obj.

加载进来的3D图像如何展示出来呢?可以使用monai.visualize.matshow3d功能。
data1 = monai.transforms.Orientation("IPL")(data) # (Left, Right), (Posterior, Anterior), (Inferior, Superior).
fig = monai.visualize.matshow3d(volume=data1,
# title='abdomen CT',
figsize=(20,20),
frames_per_row=6,
frame_dim=-3,
channel_dim=0,
every_n=5,
vmin=-300, vmax=600,
cmap='gray',
fill_value=255)

关于matshow3d函数的更多使用技巧,参考这篇文章【添加文章地址】
加载3D DICOM 图像
LoadImage同样支持加载DICOM图像和DICOM文件夹
filenames = ['/Users/Downloads/CBA60D87FAB145C3A9EB31A1C371C07E/0F9B3029D79B4B7DBE18776D284BB713.dcm',
'/Users/Downloads/CBA60D87FAB145C3A9EB31A1C371C07E/0F9B3029D79B4B7DBE18776D284BB713.dcm']
folder_path = '/Users/Downloads/CBA60D87FAB145C3A9EB31A1C371C07E'
# load single image
# data = LoadImage(image_only=True)(filenames)
# load DICOM series
data = LoadImage(image_only=True, ensure_channel_first=True)(folder_path)
print(f"image data shape: {data.shape}")
使用data.meta也可以查看元数据

加载 2D PNG图像
# 下载png图像
tempdir = tempfile.mkdtemp()
filename = os.path.join(tempdir, "MONAI-logo_color.png")
monai.apps.download_url("https://monai.io/assets/img/MONAI-logo_color.png", filepath=filename)
data, meta = LoadImage(image_only=False, reader="PILReader", reverse_indexing=False)(filename)
fig = monai.visualize.matshow3d(data, frame_dim=-1)
print(f"image data shape: {data.shape}")
print(f"meta data: {meta}")

保存图像
例如在图像分割中,想保存分割结果或者预处理后的图像。可以使用SaveImage方法。代码以Nifti图像格式为例
假设加载图像并进行了一些transform
# 假设加载图像并进行了一些transform
filenames = '/Users/Downloads/WORD-V0.1.0/imagesTr/word_0002.nii.gz'
transform = Compose(
[
LoadImaged(keys="image", image_only=True, ensure_channel_first=True),
Resized(keys="image", spatial_size=[256, 256, 240])
]
)
test_data = {"image": filenames}
result = transform(test_data)
print(f"image data shape:{result['image'].shape}")
print(result["image"].affine)
保存处理后的图像
img = result["image"]
saver = SaveImage(
output_dir='./output',
output_ext=".nii.gz",
output_postfix="trans",
# output_dtype=np.uint8, # type改变,保存出来的图像变化大,慎重选择type
resample=False,
squeeze_end_dims=True,
writer="NibabelWriter",
)
img = saver(img)
SaveImage将图像(可以是torch tensor或numpy ndarray的形式)和元数据字典保存到文件中。
保存的文件名将为{input_image_name}_{output_postfix}{output_ext},其中input_image_name是从提供的元数据字典中提取的。如果没有提供元数据,将使用从0开始的递增索引作为文件名前缀。
重要参数解析
- output_ext:输出扩展名
- output_postfix:输出后缀,默认为trans,如果输出是分割结果,可以设为seg, 最后文件名就是:{input_image_name}_seg.nii.gz
- resample: 布尔值。if True, 就会重采样到原始图像大小。适用于在transform中对image做了采样后,保存分割结果又重新采样回去。使得分割结果同原始图像一样大小。此操作会利用原始图像元数据信息的spatail_shape以及original_affine.
- squeeze_end_dims:if True, 去掉为1的维度,比如chanenl维度一般为1,保存的时候【C,H,W,D】-> 【H, W, D】
- writer: 可以设置为 None,自动根据 output_ext 来选择合适的读写器。也可以是[“NibabelWriter”、“ITKWriter”、“PILWriter”]
当然,你也可以手动指定 metadata, 以及 filename
saver(img, meta_data=img.meta, filename='img_trans.nii.gz')
注意:这个要看版本,1.3版本没有filename
总结:图像的输出非常依赖于元数据(metadata), 对医学图像不是很熟悉的,重点关注本次内容中关于元数据的部分。monai的好处是加载图像后,图像的元数据(image dir, affine, spatial shape等信息全部都保存下来了,只需要img.meta就可以调出来)。
文章持续更新,可以关注微公【医学图像人工智能实战营】获取最新动态,一个关注于医学图像处理领域前沿科技的公众号。坚持以实践为主,手把手带你做项目,打比赛,写论文。凡原创文章皆提供理论讲解,实验代码,实验数据。只有实践才能成长的更快,关注我们,一起学习进步~
我是Tina, 我们下篇博客见~
白天工作晚上写文,呕心沥血
觉得写的不错的话最后,求点赞,评论,收藏。或者一键三连

本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:chenni525@qq.com进行投诉反馈,一经查实,立即删除!
- Python教程
- 深入理解 MySQL 中的 HAVING 关键字和聚合函数
- Qt之QChar编码(1)
- MyBatis入门基础篇
- 用Python脚本实现FFmpeg批量转换
- 数据结构之AVL Tree
- 使用级联毫米波传感器的成像雷达参考设计1(TI文档)
- python爬虫效率提升——多进程
- C#中的文件操作
- 10分钟快速上手LLM大模型Python前端开发(一)
- 湖北点赋网络科技:2024咖啡饮品市场展望 解锁咖啡新趋势
- 扫雷游戏详解
- 【教3妹学编程-算法题】最大频率元素计数
- 论文阅读<MULTISCALE DOMAIN ADAPTIVE YOLO FOR CROSS-DOMAIN OBJECT DETECTION>
- 实验二 体系结构